A- и B-формы ДНК
- С помощью программы fiber пакета 3DNA были построены A- (в файле gatc-a.pdb)
и B-формы (в файле gatc-b.pdb) дуплекса ДНК,
последовательность одной из нитей которого - 4 раза повторенная
последовательность "gatc".
- Вышеуказанные файлы были проанализированы с помощью программы RasMol:
A-форма B-форма Файл dna10.pdb Тип спирали (правая или левая) правая правая правая Шаг спирали (Å) 28,02 33,75 29,81 Число оснований на виток 11 10 10 Ширина большой бороздки (Å) 7,985 (T7A.P-C20B.P) 17,915 (T19B.P-A10A.P) 15,192 (A3A.P-T5A.P) Ширина малой бороздки (Å) 16,808 (A10A.P-G25B.P) 11,691 (G29B.P-C8A.P) 9,490 (T8A.P-C7A.P) - Из архива была получена структура dna10.pdb.
� В этой структуре интересно то, что ширина ее малой и большой бороздок варьирует при их
различном измерении, в то время как в A- и B- формах ДНК ширина бороздок стабильна. Как видно из
таблицы, число оснований на виток анализируемой структуры совпадает с таковым B-формы, и, кроме
� того, при изображении структуры с торца, отверстия не наблюдается, а основания перпендикулярны
главной оси, что так же свидетельствует о сходстве с B-формой.
- При помощи программ find_pair и analyze был проведен анализ трёх структур ДНК.
Ниже приведены значения конформационно важных торсионых углов (α, β,
γ, δ, ε, ζ, χ).
� A-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2 2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2 9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 16 C -51.7 174.8 41.7 79.1 --- --- -157.2
B-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 C -29.9 136.4 31.1 143.4 --- --- -98.0
структура dna10 Strand I base alpha beta gamma delta epsilon zeta chi 1 C --- --- -173.5 69.9 -149.9 -68.5 -153.5 2 C -79.4 174.1 39.6 142.6 -105.1 163.4 -78.3 3 A -71.5 147.4 50.0 142.7 -172.3 -86.2 -81.7 4 G -69.7 169.6 44.7 139.6 -131.1 175.8 -79.5 5 T -70.1 141.2 56.8 138.1 176.8 -112.6 -107.9 6 A -60.6 -175.1 57.9 146.4 -169.9 -86.5 -113.2 7 C -69.7 167.5 49.8 90.5 -173.7 -87.2 -127.2 8 T -59.7 171.8 52.6 141.4 -98.8 164.9 -90.0 9 G -74.8 152.6 46.7 146.1 -171.5 -90.4 -77.2 10 G -64.9 160.9 53.0 113.1 --- --- -104.8
структура dna10 Strand II base alpha beta gamma delta epsilon zeta chi 1 G -64.9 160.9 53.0 113.1 --- --- -104.8 2 G -74.8 152.6 46.7 146.1 -171.5 -90.4 -77.2 3 T -59.7 171.8 52.6 141.4 -98.8 164.9 -90.0 4 C -69.7 167.5 49.8 90.5 -173.7 -87.2 -127.2 5 A -60.6 -175.1 57.9 146.4 -169.9 -86.5 -113.2 6 T -70.1 141.2 56.8 138.1 176.8 -112.6 -107.9 7 G -69.7 169.6 44.7 139.6 -131.1 175.8 -79.5 8 A -71.5 147.4 50.0 142.7 -172.3 -86.2 -81.7 9 C -79.4 174.1 39.6 142.6 -105.1 163.4 -78.3 10 C --- --- -173.5 69.9 -149.9 -68.5 -153.5
Для структуры dna10 приведены значения обеих цепей, поскольку они различны, � в отличие от A- и B- форм, у которых значения углов обеих цепей одинаковы. � Легко заметить, что у A- и B- форм значения углов для различных оснований (столбцы) разнятся не более чем на 0,1, в то время как у структуры dna10 эта разность значительно больше.Интересно то, что программа analyze содержит информацию, предполагающую форму входящей ДНК. Из файла dna10.out получаем:
Classification of each dinucleotide step in a right-handed nucleic acid structure: A-like; B-like; TA-like; intermediate of A and B, or other cases step Xp Yp Zp XpH YpH ZpH Form 1 CC/GG -3.39 8.90 -1.68 -3.74 9.00 1.04 2 CA/TG -2.12 8.21 -1.39 1.34 7.92 -2.57 3 AG/CT -3.53 8.90 -1.11 -4.79 8.53 2.78 B 4 GT/AC -3.10 8.90 -0.61 -2.64 8.89 -0.78 B 5 TA/TA -2.54 8.49 0.39 -2.91 8.45 0.93 B 6 AC/GT -3.10 8.90 -0.61 -2.64 8.89 -0.78 B 7 CT/AG -3.53 8.90 -1.11 -4.79 8.53 2.78 B 8 TG/CA -2.12 8.21 -1.39 1.34 7.92 -2.57 9 GG/CC -3.39 8.90 -1.68 -3.74 9.00 1.04
Эта информация свидетельствует о том, что наши предположения о том, что структура dna10 является B-формой ДНК, скорее всего верны.
- При помощи программы pdb2img, были получены изображения структур
� в виде стопочных моделей.
A-форма

B-форма
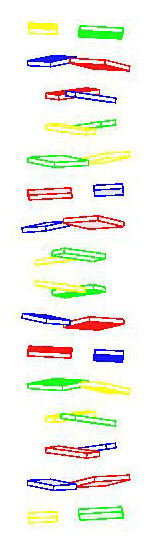
структура dna10