Практикум 3
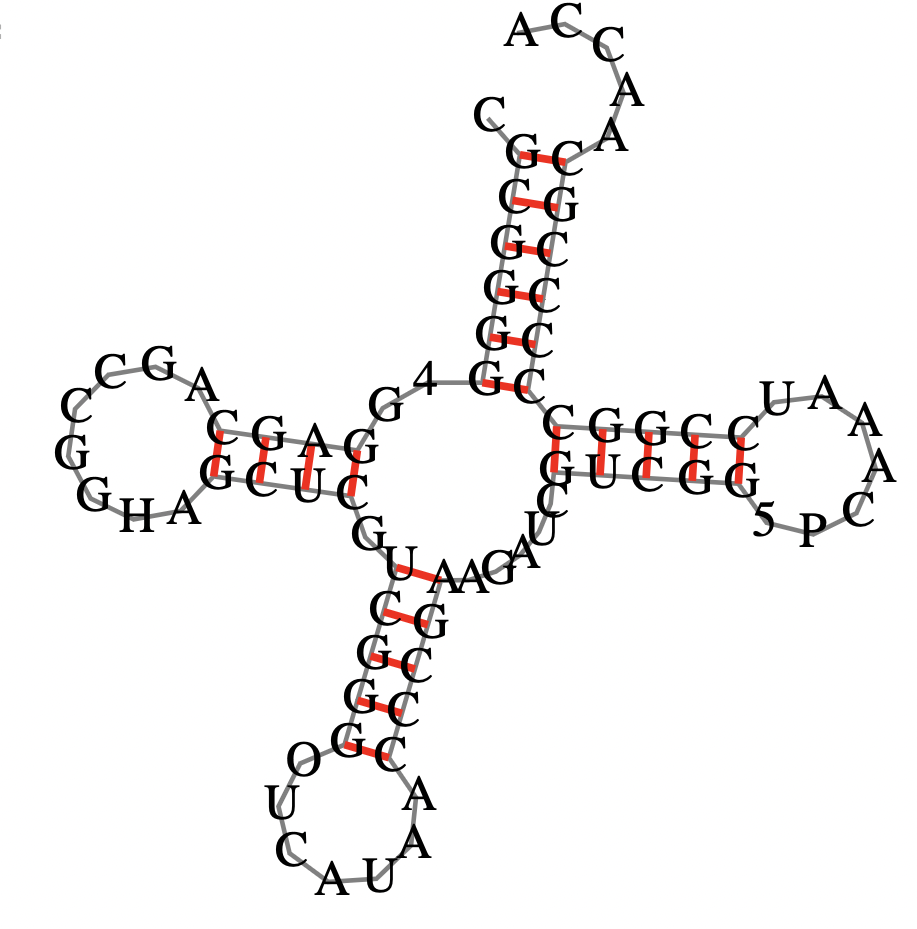
Предсказание вторичной структуры заданной тРНК
Для того чтобы найти инвертированные участки в последовательности используется программа einverted.Однако с моей последовательностью с PDB 2fmt были проблемы, которые решились при сильном изменении параметров.В итоге использовалась данная команда:
! einverted -sequence rna.seq -gap 12 -threshold 0 -gap 8 -match 5 -mismatch 0 -outfile outfile -outseq seqout
В итоге были получены файлы outfile и seqout .
Далее с помощью алгоритма Зукера была предсказана вторичная структура тРНК.Результат хорошо соотносится с полученным при использовании find_pair.
| Участок структуры | Позиции в структуре(find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'-902-907-3' 5'-966-971-3' Всего 6 пар | Предсказано 5 пар из 6 | Предсказано 6 пар |
| D стебель | 5'-10-13-3' 5'-22-25-3' Всего 4 пары | Предсказано 3 пары из 4 | Предсказано 4 пары |
| Т стебель | 5'-49-53-3' 5'-61-65-3' Всего 5 пар | — | Предсказано 5 пар из 5 |
| Антикодоновый стебель | 5'-38-44-3' 5'-26-32-3' Всего 7 пар | — | Предсказано 5 пар из 5 |
| Число канонических пар нуклеотидов | 22 | 8 | 20 |
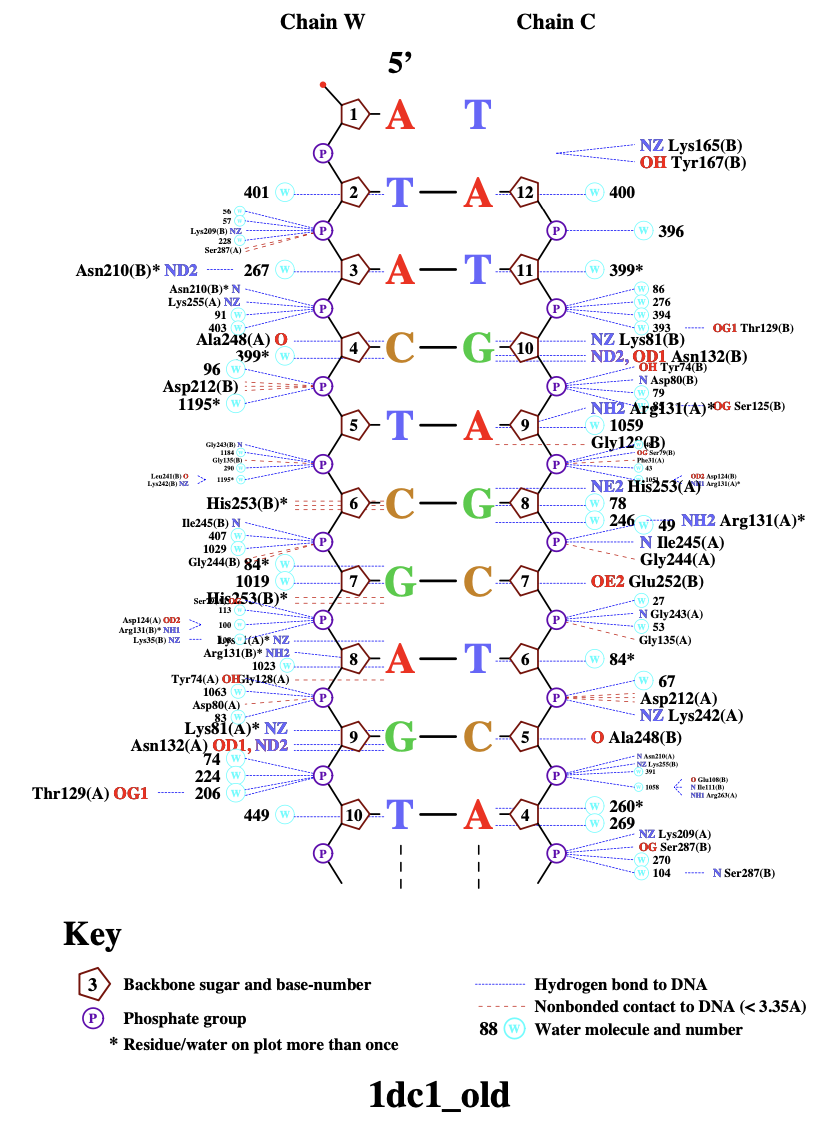
Поиск ДНК-белковых контактов в заданной структуре
Упражнение 1

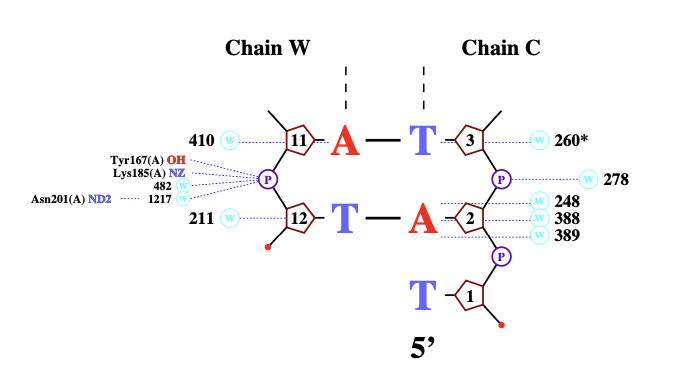
Упражнение 2
| Контакты атома белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| Остатками 2'-дезоксирибозы | 10 | 62 | 72 |
| Остатками фосфорной кислоты | 30 | 35 | 65 |
| Остатками азотистых оснований со стороны большой бороздки | 4 | 11 | 15 |
| Остатками азотистых оснований со стороны малой бороздки | 2 | 9 | 11 |
Упражнение 3
Файл pdb белка был с помощью remediator переведен в старый формат, после чего программой nucplot была создана схема ДНК-белковых контактов.