Практикум 2
Построение различных форм ДНК с помощью программы Fiber
Было построить модели структур A-, B- и Z-формы ДНК с помощью пакета Fiber. Последовательность ДНК состояла из пяти последовательностей GATC.Использованная команда(дя других структур меняется только опция):
fiber -seq=GATCGATCGATCGATCGATC -a gatc-a.pdb
Структуры можно скачать:тут (структура А) , тут (структура В)
и тут (структура Z) .
| А | B | Z |
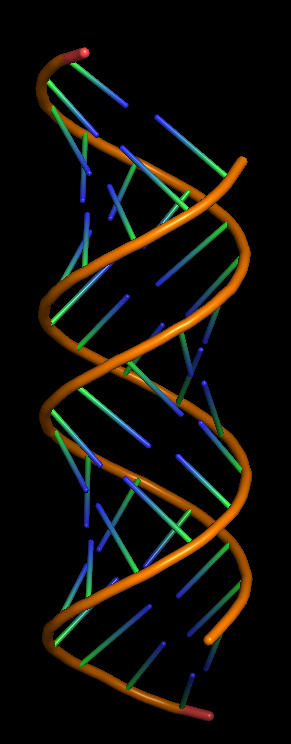 |
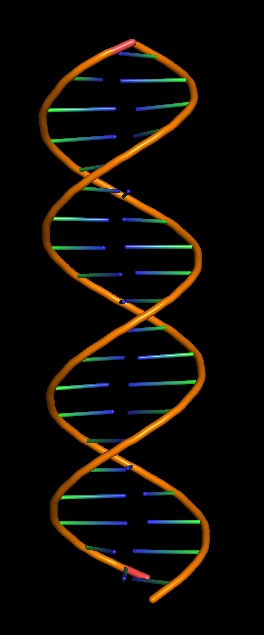 |
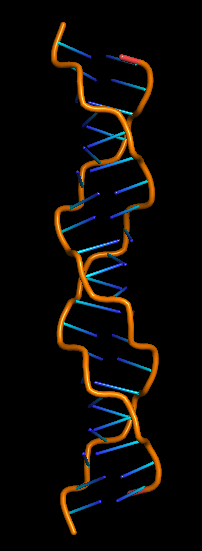 |
 |
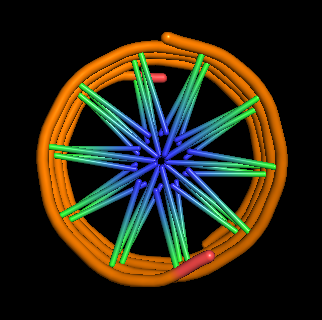 |
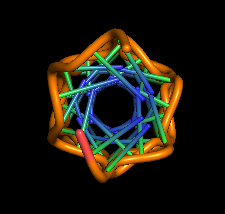 |
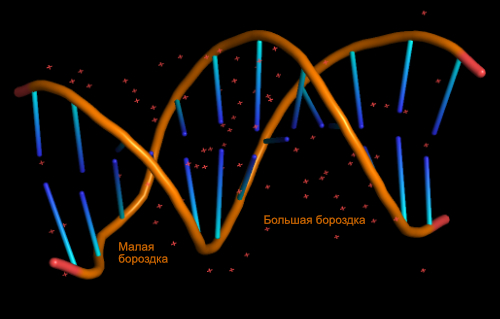
Реальные структуры ДНК.Большие и малые бороздки ДНК.
Для сравнения реальной структуры ДНК с экспериментальной была выбрана форма В и структура 1bna.
На рисунке выделены большая и малая бороздки.
Теперь выберем какой-нибудь цитозин какие атомы обращении в сторону большей или малой бороздки.Рисунок был построен с помощью программы MarvinSketch,а также атомы покрашены по принципу красные-большая, синие-малая.
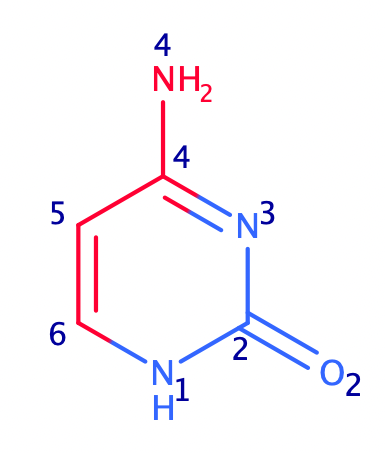
В сторону большой бороздки:C4,C5,C6,N4
В сторону малой бороздки:N3,C2,O2,N1
Далее программой JMol были проведены измерения форм ДНК. Результаты представлены в таблице ниже.
| А | В | Z | |
|---|---|---|---|
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Å) | 28,03 | 33,75 | 43,5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки,Å | 7,98 ([DT]11:A.P #208 - [DG]33:B.P #657) | 18,1 ([DG]16:B.P #302 - [DT]7:A.P #122) | 8,868 ([DG]13:A.P #247 - [DG]33:B.P #657) |
| Ширина малой бороздки,Å | 16,97 ([DT]11:A.P #208 - [DC]24:B.P #474) | 8,9 ([DG]10:A.P #181 - [DT]19:B.P #366) | 23,5 ([DC]28:B.P #556 - [DC]8:A.P #146) |
Структура тРНК.
Торсионные углы.
Для работы был задан код PDB 2fmt7.Чтобы получить данные для анализа использовались команды:
remediator --old ''2fmt.pdb'' > ''2fmt_old.pdb
find_pair -t 2fmt_old.pdb stdout | analyze
В итоге был получен файл 2fmt_old.out, который также включал значение торсионный углов.После подсчета средних углов и сравнения с теоретическими был сделан вывод, что данная структура больше всего похожа на форму А.
Водородные связи.
В том же файле были представлены эти данные:

Были получены:
Координаты стеблей
Неканонические пары
Пары стабилизирующие структуру РНК