Практикум 4
Составление списка гомологичных белков, включающих паралоги
Были скачены протеом отобранных бактерий из репозитория, после чего объеденены в единый файл proteomes.fasta командой:
cat ./proteomes/*.fasta > proteomes.fasta
После чего этот файл был проиндексирован командой
makeblastdb -in proteomes.fasta -dbtype prot -parse_seqids
Далее локальным запуском blastp были найдены гомологи белка CLPX_ECOLI из полученной выше базы данных, также был установлен порог для e-value равный 0,001.
blastp -query P0A6H1.fasta -db proteomes.fasta -out final.tab -evalue 0.001 -outfmt 7
Выдача команды находится тут.
Реконструкция и визуализация
Все находки были объединены в единый файл, который можно посмотреть тут.
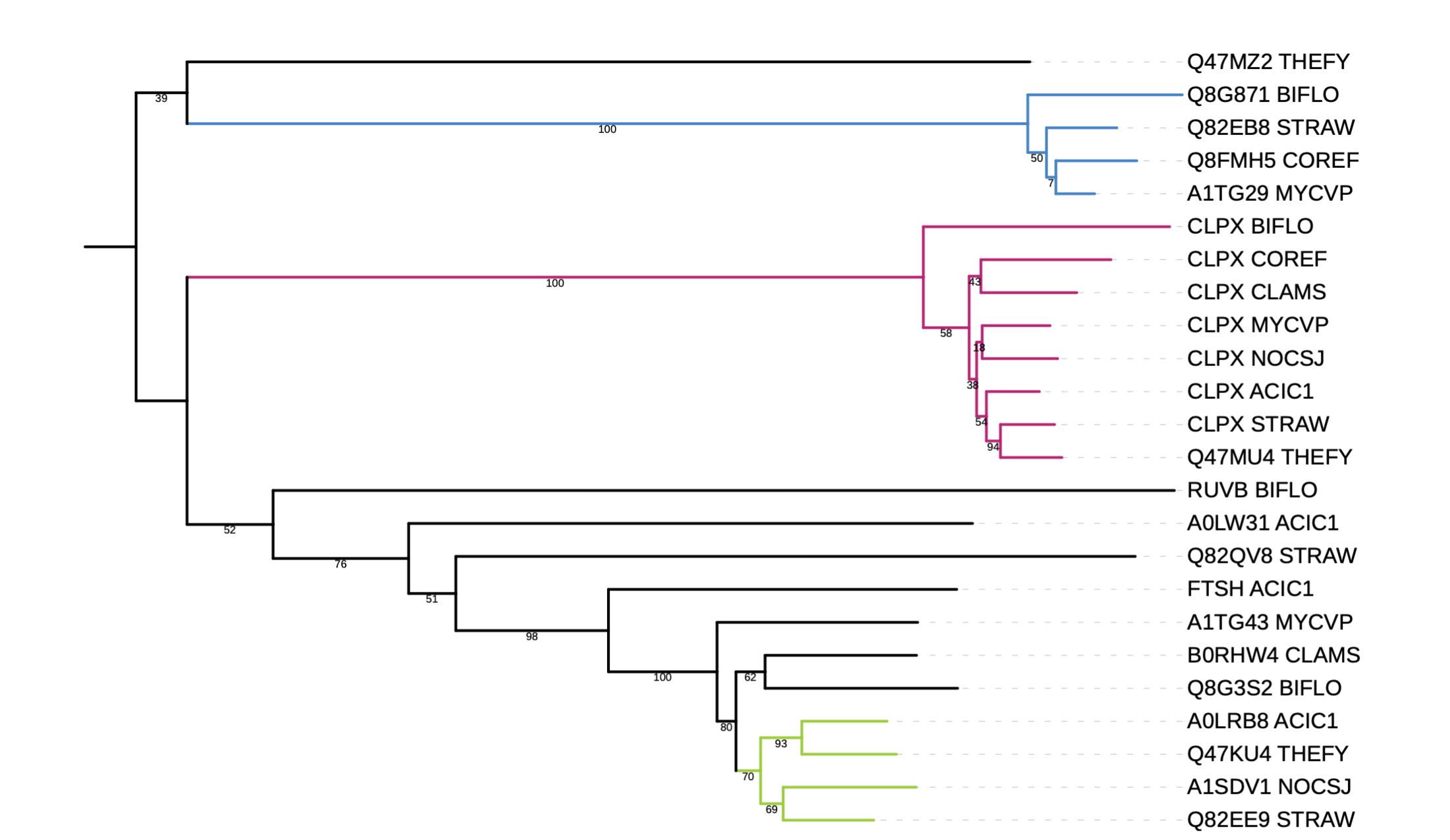
Далее было построено дерево с заданными нам параметрами, которое в формате Newick можно найти тут
Считаем что реконструированное дерево точно правильное, тогда можно привести примеры ортологов и паралогов.
| Ортологи | Паралоги |
|---|---|
| A0LRB8_ACIC1,Q47KU4_THEFY | A0LRB8_ACIC1,FITSH_ACIC1 |
| CLPX_COREF,CLPX_CLAMS | Q82EE9_STRAW,Q82QV8_STRAW |
| Q8FMH5_COREF,A1TG29_MYCVP | RUVB_BIFLO,Q8G3S2_BIFLO |
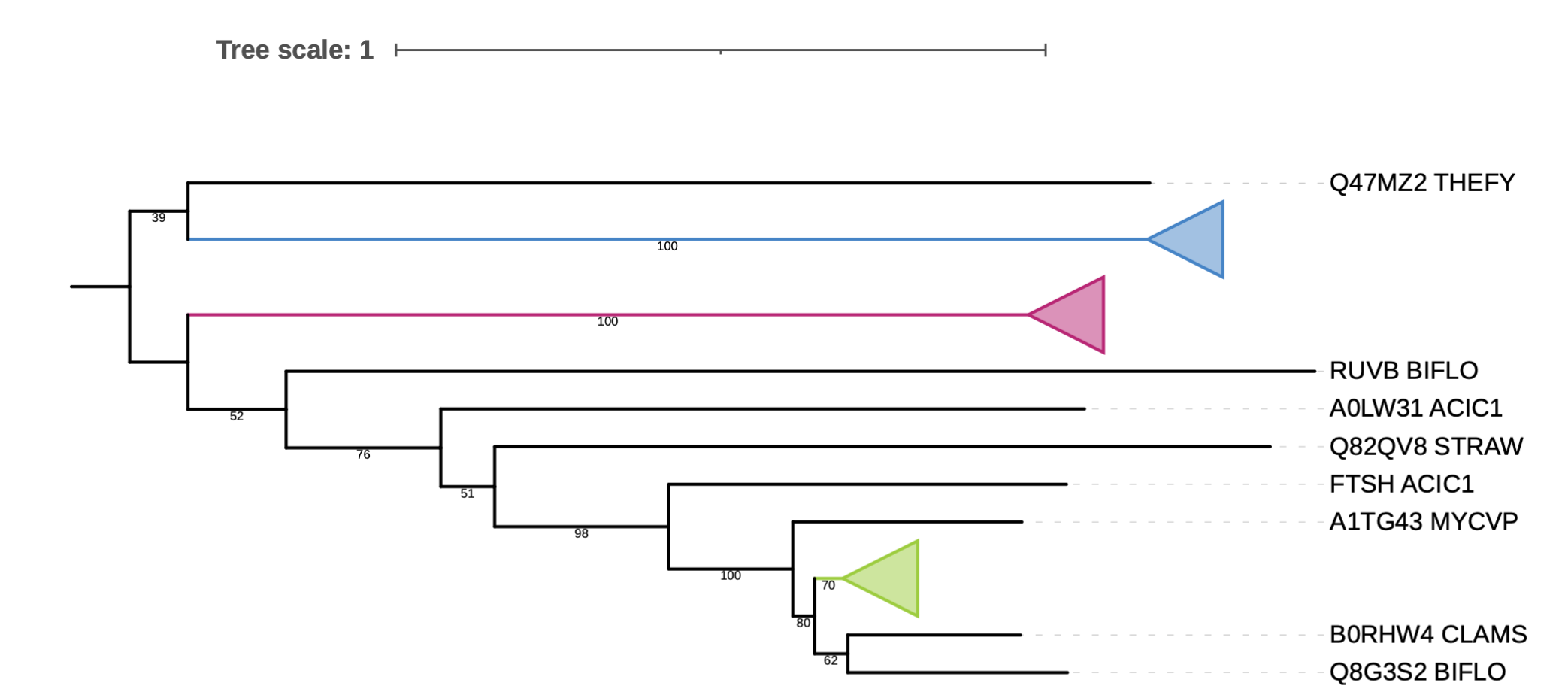
Далее на полученном дереве были покрашены ортологичные группы.