Практикум 6
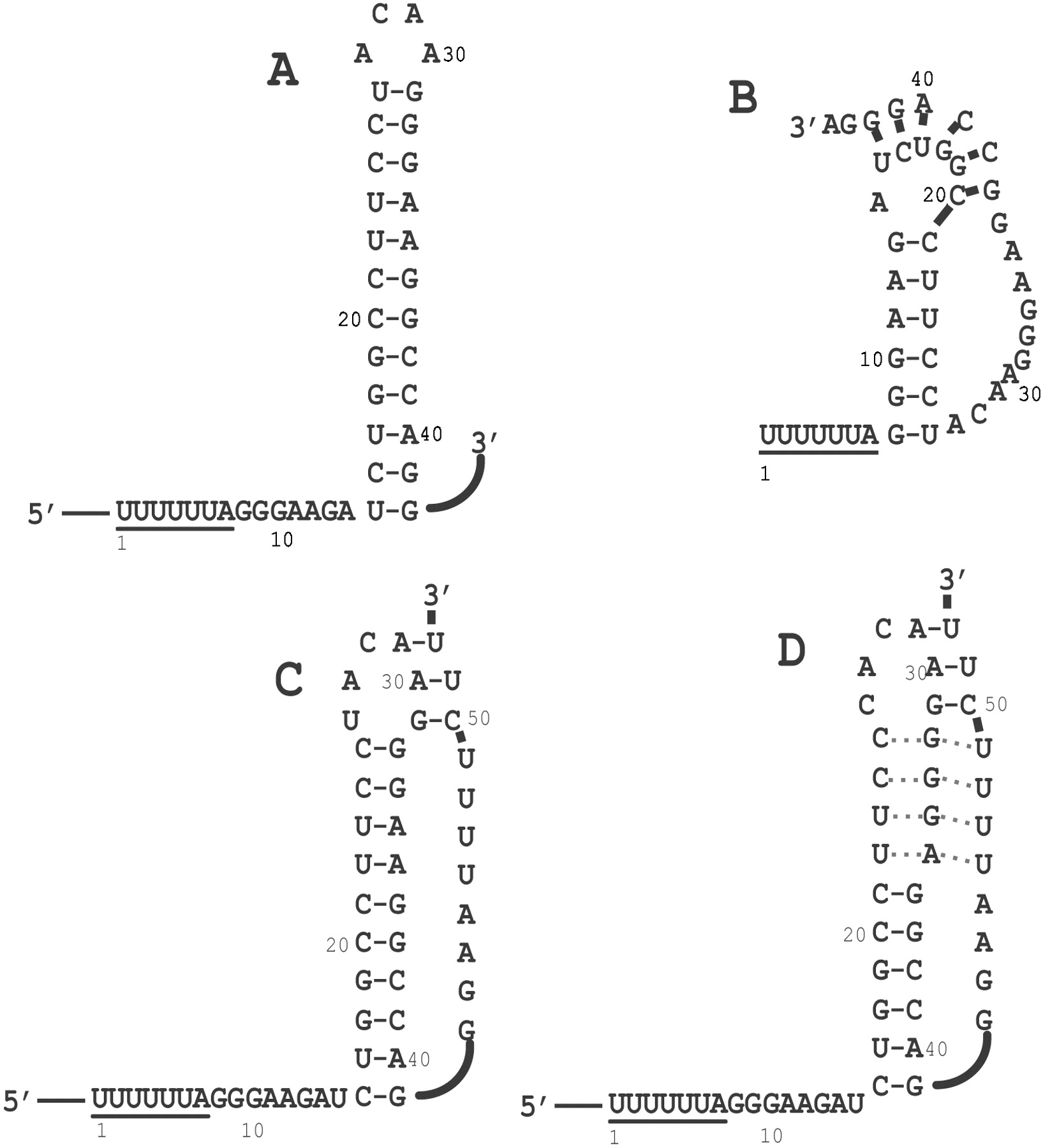
Сигнал рибосомного сдвига рамки считывания HIV-1
Сигнал рибосомного сдвига рамки рамки считывания HIV-1 - это такой рибосомный сдвиг рамки (PRF), который ВИЧ использует для возможности трансляции нескольких белков из одной последовательности.
Вирусу иммунодефицита человека требуется запрограммированный сдвига рамки рибосомы-1 для экспрессии гена Pol. Сайт в ВИЧ-1 - гептамер 5'-UUUUUUA-3', где происходит сдвиг рамки, за которой следует спейсерная область и расположенная ниже структура «стебель-петля» РНК. Рибосомный сдвиг рамки ВИЧ-1 заставляет примерно 5% рибосом смещаться в рамку считывания -1, в итоге производится полипротеин Gag-Pol. Эффективность сигнала составляет 5%, она определяет соотношение продуцируемых вирусных белков и необходима для репликации, что определяет инфекционность вируса. Если снизить эффективность может ингибировать репликацию вируса. Несмотря на выше сказаное соотношение Gag к Gag-Pol относится как 20 к 1, то есть сигнал низкоэффективен.
Построение матрицы PWM
Для выполнения задания использовался скрипт Георгия Муравьева, который можно увидеть здесь.
Скрипт для работы получает таблицу с информацией о генах человека в формате tsv, и выдает следующие файлы:
Была получена позиционная весовая матрица с псевдоттсчетами
| Позиционная весовая матрица | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
| A | 2.13 | 2.16 | 2.32 | 2.32 | 2.83 | 2.64 | 2.12 | 3.74 | -4.08 | -4.08 | 2.34 | 2.48 | 1.98 |
| T | 2.19 | 2.03 | 2.28 | 1.78 | 1.69 | 1.89 | 1.48 | -4.08 | 3.74 | -4.08 | 1.82 | 1.94 | 2.27 |
| G | 2.59 | 3.14 | 2.75 | 2.80 | 3.02 | 2.60 | 2.87 | -3.72 | -3.72 | 4.11 | 3.34 | 2.41 | 3.11 |
| C | 3.11 | 2.65 | 2.80 | 3.05 | 2.08 | 2.87 | 3.21 | -3.72 | -3.72 | -3.72 | 2.17 | 3.11 | 2.64 |
На основе таблицы были рассчитаны веса последовательности и построена гистограмма распределения.Порог веса, выше которой находка может считаться правильной составил 37, в итоге была составлена таблица с результатами проверки.
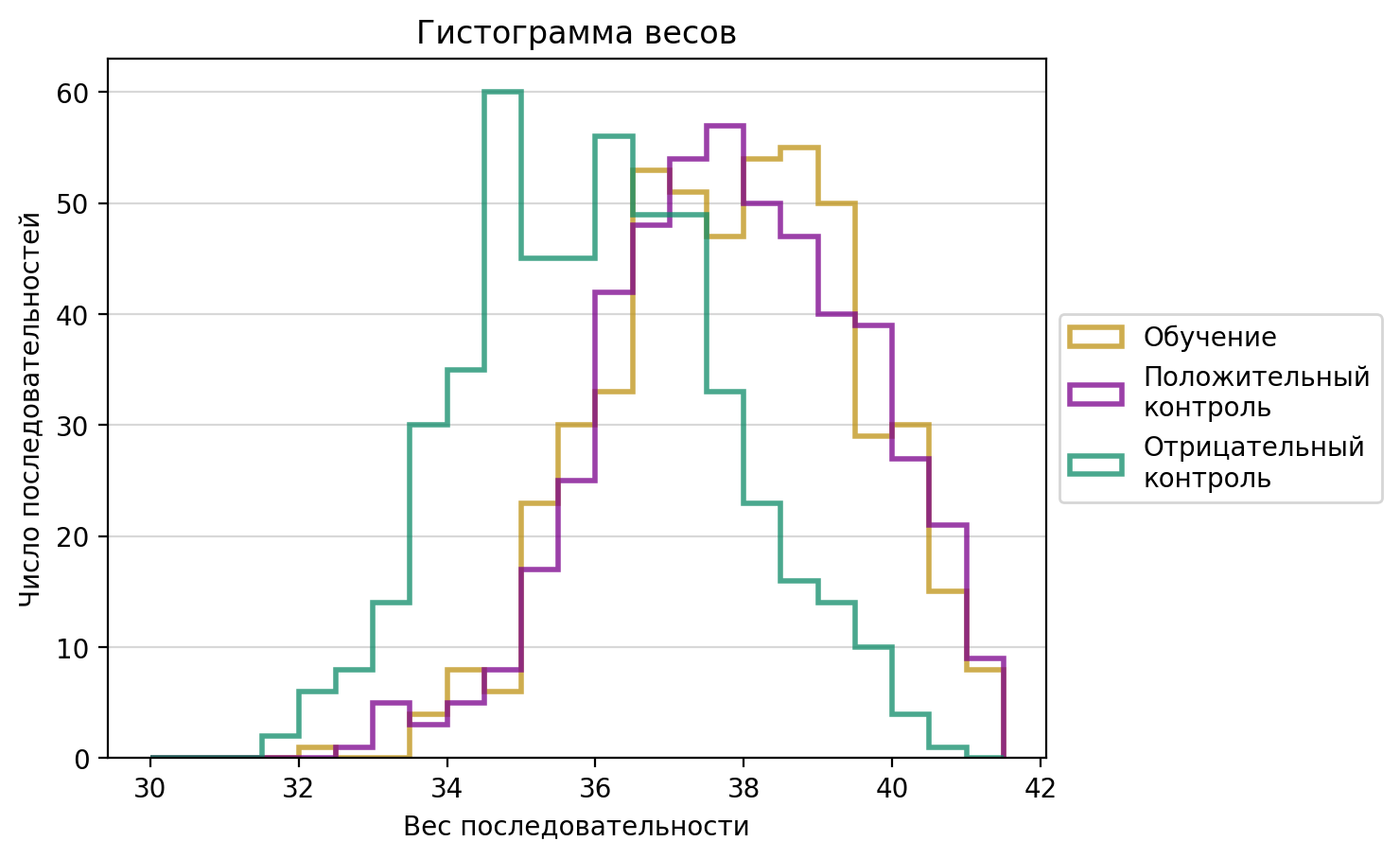
| Позиционная весовая матрица | |||
|---|---|---|---|
| Обучение | Положительный контроль | Отрицательный контроль | |
| + Сигнал | 342(68.4%) | 346(69.2%) | 150(30.0%) |
| - Сигнал | 158(31.6%) | 154(30.8%) | 350(70.0%) |
Информационное содержание сигнала,Logo
Была получена матрица информационного содержания и построено LOGO сервисом WebLOGO 3.
| Позиционная весовая матрица | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
| A | 7.71 | 8.05 | 10.04 | 10.16 | 20.55 | 15.83 | 7.59 | 67.56 | 0.00 | 0.00 | 10.40 | 12.62 | 6.16 |
| T | 8.39 | 6.60 | 9.56 | 4.49 | 3.90 | 5.31 | 2.78 | 0.00 | 67.56 | 0.00 | 4.80 | 5.73 | 9.44 |
| G | 10.30 | 21.54 | 12.82 | 13.64 | 18.44 | 10.43 | 15.02 | 0.00 | 0.00 | 74.13 | 27.97 | 8.02 | 20.64 |
| C | 20.64 | 11.22 | 13.64 | 19.17 | 4.96 | 15.02 | 23.65 | 0.00 | 0.00 | 0.00 | 5.64 | 20.79 | 10.95 |
| IC(j) | 47.05 | 47.40 | 46.06 | 47.46 | 47.86 | 46.60 | 49.05 | 67.56 | 67.56 | 74.13 | 48.81 | 47.16 | 47.20 |
Число сайтов GAATTC в полном геноме одного штамма E.coli
Был использован геном штама Escherichia coli str. K-12 substr. MG1655.
В результате было найдено 646 сайтов GAATTC в геноме, но ожидаемое число таких сайтов в геноме равно 1097. Такое различие статистически значимо, так как p-value=1.770*10^(-49) при биномиальном распределении.