Практикум 7
Подготовка данных
Для работы был выбран штамм ATCC BAA-2146 бактерии Klebsiella pneumoniae.Для нее была скачена сборка, которую можно найти здесь, и файл с аннотацией, который можно найти здесь.Далее с помощью сервиса Operon-mapper был получен список оперонов, который можно увидеть здесь. Cтоит отметить что в выводе также есть функциональная аннотация и ID кластеров ортологичных генов или COGs.
Для дальнейших действий был использован ноутбук Сергея Бушуева - его можно увидеть здесь.В итоге были получены следующие файлы:
Запуск MEME
Далее был проведен локальный запуск MEME:
meme train.fa -dna -minw 5 -maxw 50 -nmotifs 3
Было найдено три мотива:
Только первый проходит по E-value, поэтому дальнейшая работа велась с ним (хотя по длине больше подходит второй мотив).
Текстовую выдачу можно увидеть здесь.

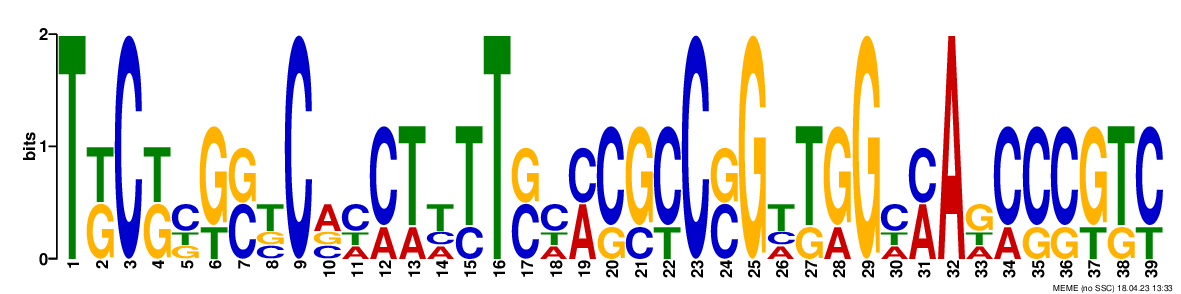
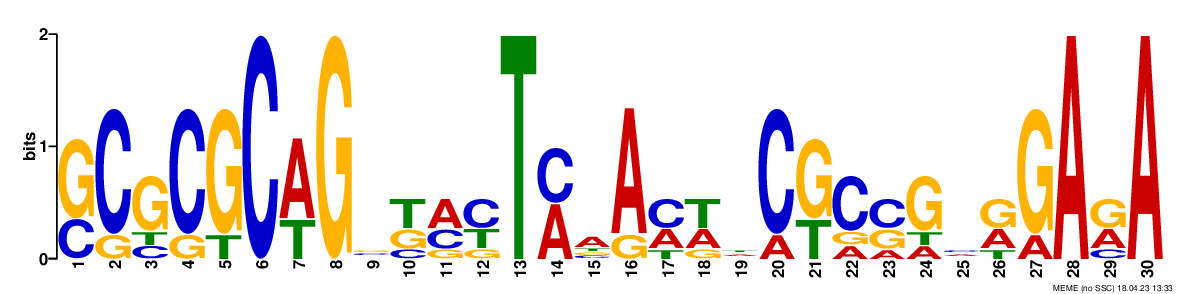
Запуск FIMO
Для поиска первого мотива в положительном и отрицательном контролях были запущены следующие команды:
fimo --norc -motif RCCBTTDTGCTAHAVTVCSCACCCYTTT -thresh 0.01 ./meme_out/meme.txt test.fa
fimo --norc -motif RCCBTTDTGCTAHAVTVCSCACCCYTTT -thresh 0.01 ./meme_out/meme.txt neg_contr.fa
Сначала был использован e-value = 0.001, но находок было совсем мало(5 из 50 для положительного контроля,3 из 50 для негативного). Поэтому дальше велась работа с порогом e-value на 0.01.
В итоге было получено 38 находок из 50 последовательностей в положительном контроле, выдачу можно увидеть здесь,и найдено 44 находки в 29 последовательностях из 50 в негативном контроле, выдачу можно увидеть здесь.Отсюда можно сделать вывод, что возможно смена порога e-value была не нужна.