Практикум 8
Поиск консервативного мотива
Для работы было выбрано семейство белков лизиновых декарбоксилаз, что доказано биоинформатически, но пока не экспериментально - PF03641.
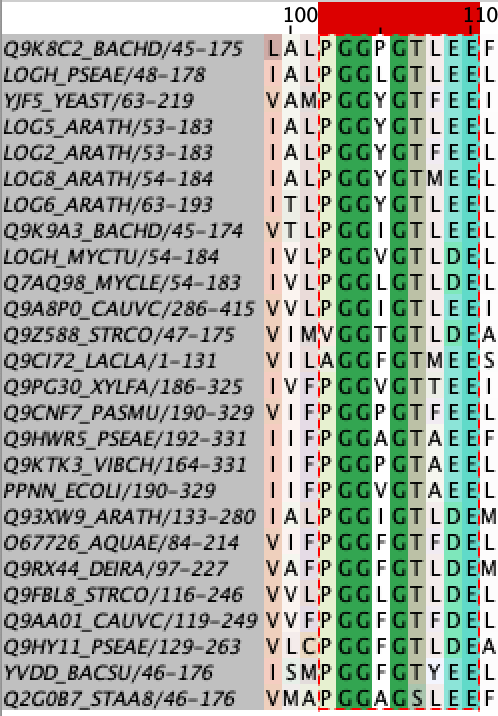
Выравнивание seed для этого семейства включает в себя 26 последовательностей, само выравнивание можно увидеть здесь.
Был найден консервативный мотив PGG.GT.[DE]E встречающийся в 23 последовательностях. Стоит отметить, что если заменить в двух последовательностях аминокислотные остатки на месте Р они будут совпадать с паттерном,а в одной последовательности треонин заменен на серин, а они структурно схожи.
Далее был проведен поиск этого мотива по базе данных SwissProt на сайте MyHits. Было найдено 29 находок которые были или ферментами активирующими цитокин, они включают в себя исследуемое семейство и их близких родственников, или непосредственно самим семейством. Результат поиска можно увидеть здесь.
Поиск мотива специфичного для клады
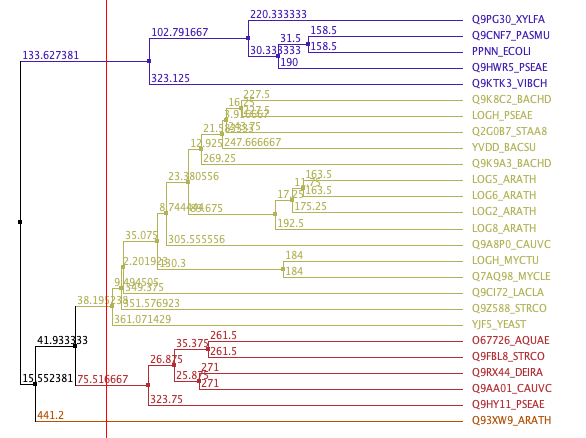
Далее по выравниванию было построено дерево алгоритмом UPGMA. В итоге было выделено 4 группы, одна тривиальная, в двух по 5 последовательностей и в одной 15 последовательностей, дальнейший анализ велся с ней.
Был найден мотив [GSQ][GRL]...[GA]..[PV], стоит отметить что в местах где в мотиве есть вариативность(2,3 варианта аминокислотных остатков) она наблюдается у одной последовательности на вариант, также в последовательностях с вариативностью она одна (если есть замена одного аминокислотного остатка, то остальные как у большинства). Также все места где точка, там аминокислоты с сходным характером (например гидрофобные).
Такой мотив не был обнаружен в других последовательностях,что говорит о его специфичности для выбранной клады.

PSI-BLAST
Для анализа был выбран белок с AC идентификатором - C4Z088. Это ингибитор клеточного деления, блокирующий образование полярных Z-колец.
| Таблица итераций PSI-BLAST | |||||
|---|---|---|---|---|---|
| Номер итерации | Число находок выше порога | Идентификатор худшей находки выше порога | E-value находки | Идентификатор лучшей находки ниже порога | E-value находки |
| 1 | 162 | Q2P036.1 | 0.004 | Q5F5V4.1 | 0.005 |
| 2 | 188 | O25693.2 | 2e-09 | - | - |
| 3 | 188 | Q9ZM51.1 | 1e-11 | A7H8E6.1 | 0.012 |
| 4 | 188 | Q9ZM51.1 | 5e-13 | A7H8E6.1 | 0.016 |
Уже с третей итерации,число находок с E-value меньше порохового перестало меняться, также различие худшей выше порога и лучшей ниже порога значительно (9 порядков),из чего модно сделать вывод о хорошей обособленности группы. Следовательно семейство белков обосновано сходством их последовательностей.
Проверка числа ТА в бактериальном геноме
Была взята референсная сборка бактерии [Arcobacter] porcinus, которую можно найти здесь.Ожидаемое число ТА сайтов составило 267216, реальное число 213266. При этом p-value = 0.0 значит такое отличие является статистически значимым.