Предсказание генов эукариот.
1. Описание выданного контига и одного из генов на нем, для которого предсказан альтернативный сплайсинг:
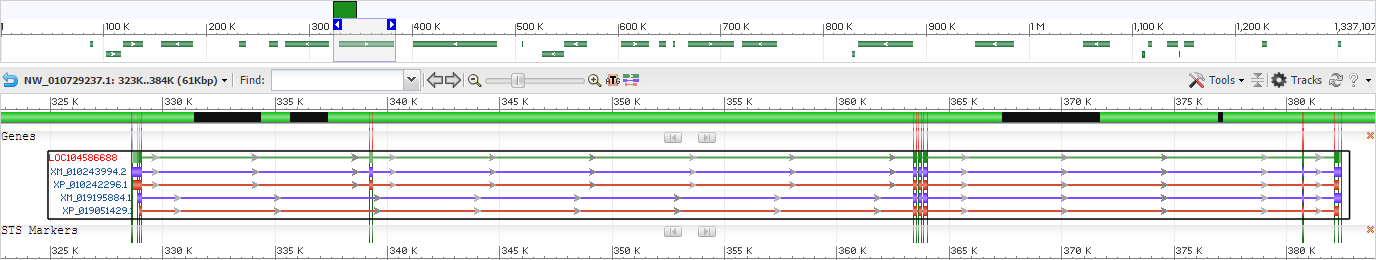
Мой контиг был плохой, поэтому был описан резервный контиг NW_010729237 Лотоса орехоносного (Nelumbo nucifera, таксономия: cellular organisms; Eukaryota; Viridiplantae; Streptophyta; Streptophytina; Embryophyta; Tracheophyta; Euphyllophyta; Spermatophyta; Magnoliophyta; Mesangiospermae; eudicotyledons; stem eudicotyledons; Proteales; Nelumbonaceae; Nelumbo). В контиге 1335163 bp, 28 генов, 41 CDS.
Рисунок 1. Ген LOC102560820.
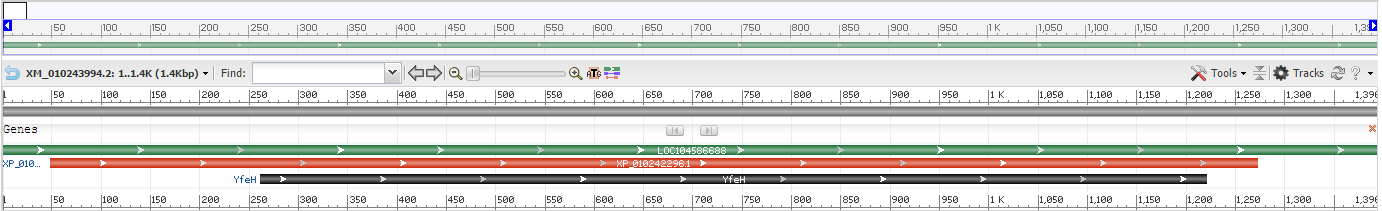
Рисунок 2. мРНК XM_010243994.2 и продукт её трансляции.
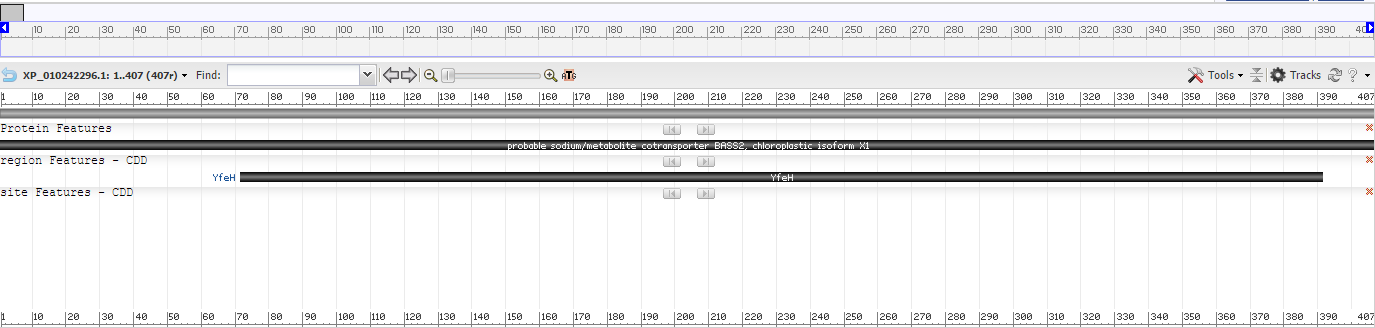
Рисунок 3. Изоформа X1 белка XP_010242296.1.
2. Предсказание генов и белок-кодирующих областей в выданном контиге с помощью web-сервера AUGUSTUS:
В качестве организма, из генома которого будут браться параметры модели, был выбран Solaneum lycopersicum (томат).
Выбранные параметры модели:
Prediction job ID: predyGgD8sue - ID запроса AUGUSTUS parameter project identifier: tomato - ID организма с параметрами для модели Genome file: cont.fasta - входной файл User set UTR prediction: false - мануальные настройки для более эффективного поиска UTR Report genes on: both strands - находить гены на обоих цепях Alternative transcripts: none - гены с альтернативным сплайсингом Allowed gene structure: predict any number of (possibly partial) genes - количество генов Ignore conflictes with other strand: false - искать гены независимо на двух цепях
Результаты, выданные Augustus:
augustus.aa - трансляции предсказанных генов в формате .fasta
augustus.gff - предсказания генов в формате .gff
augustus.gtf - предсказания генов в формате .gtf
augustus.mrna - предсказанные мРНК в формате .fasta
augustus.cdsexons - предсказанные экзоны в формате .fasta
augustus.gbrowse - координаты найденных генов, мРНК и т.д.
Затем, с помощью скрипта на питоне, был проведен анализ полученных данных:
Таблица 1. Анализ полученных данных:
| Число совпадающих генов | 124(47.9%) |
| Число генов, совпадающих по N-концу | 24(9.3%) |
| Число генов, совпадающих по С-концу | 48(18.5%) |
| Число генов, вообще не совпадающих по концам | 64.3(24.3%) |
Для гена, изображенного на Рис.1., правильно предсказан лишь 1 экзон из 6 (еще у двух правильно предсказаны концы, и у одного - начало), и в разы больше лишних предсказаний. Различия могут быть вызваны сложностью определения границ экзонов у эукариот, а так же недостаточностью информации (использование томата для парметров, а не самого лотоса например).