Нуклеотидный blast.
1. Таксономия и функция прочтенной мной нуклеотидной последовательности (из практ. 6):
Был запущен нуклеотидный BLAST последовательности WSLOX2_COI_F_C01_WSBS-Seq-1-08-15.fasta с алгоритмом blastn (Somewhat similar sequences) против базы данных Nucleotide collection (nr/nt). Параметры алгоритма - Exclude Uncultured/environmental sample sequences.
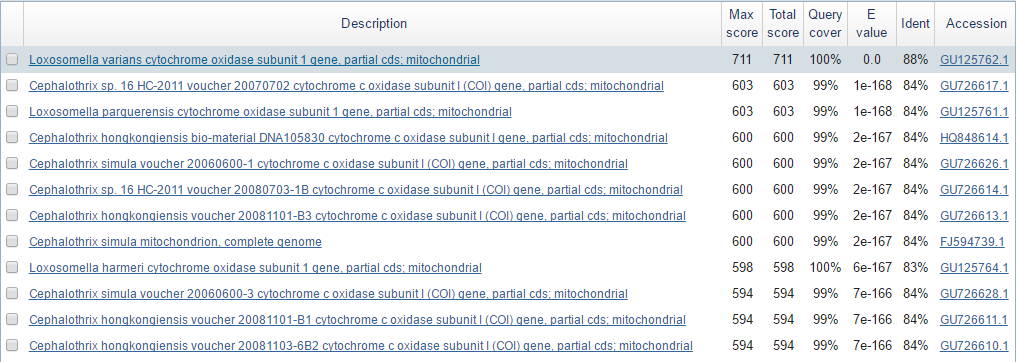
Рисунок 1. Выдача BLAST'а.
Последовательность, скорее всего, кодирует Cytochrome c oxidase subunit I. Видно, что Е-value, Query cover, Ident и Score находок очень хорошие, а у вторых двух и следующими за ними пяти эти показатели совпадают.
Биологические источники этих последовательностей - немертины двух видов, со следующей таксономией:
Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Protostomia; Lophotrochozoa; Nemertea; Anopla; Palaeonemertea; Cephalotrichidae; Cephalothrix.
И внутрипорошицевые двух видов, со следующей таксономией:
Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Protostomia; Lophotrochozoa; Entoprocta; Loxosomatidae; Loxosomella.
Выравнивание лежит в проекте JalView.

Рисунок 2. Выравнивание первых 8 находок.
Рисунок 9. Blast для Loxosomella varians по роду.
Рисунок 10. Blast для Cephalothrix varians по роду.
Рисунок 11. Blast для Empididae varians по роду.
Среди рода Loxosomella видно, что разброс очень большой, от 87% до 100% по покрытию и от 71% до 100% по идентичности. В Cephalothrix покрытие от 97% до 100%, идентичность от 84% до 100%. Значит, наш организм может принадлежать обоим этим родам. Для Acropsopilio в genbank только 1 вариант, так что о принадлежность к этому роду просто сказать ничего не можем. У Loxocorone тоже 1 вид. Для Empididae разброс идентичности от 87% до 100%, т.е. наш вид точно не входит в этот род. Если посмотреть на таксономию выбранных родов, то видно, что Loxosomella и Cephalothrix принадлежат одной систематической группе лишь на уровне Lophotrochozoa. Более точно определить систематику нельзя.
2. Сравнение списков находок нуклеотидной последовательности 3-я разными алгоритмами blast:
Таблица 1. Описание списков.
| Алгоритм | Число находок | E-value худшей находки | Ident худшей находки | Query cover худшей находки |
| megablast | 2 | 2e-147 | 84% | 95% |
| discontiguous megablast | 13 | 1e-87 | 74% | 89% |
| blastn | 19 | 2.7 | 100% | 2% |
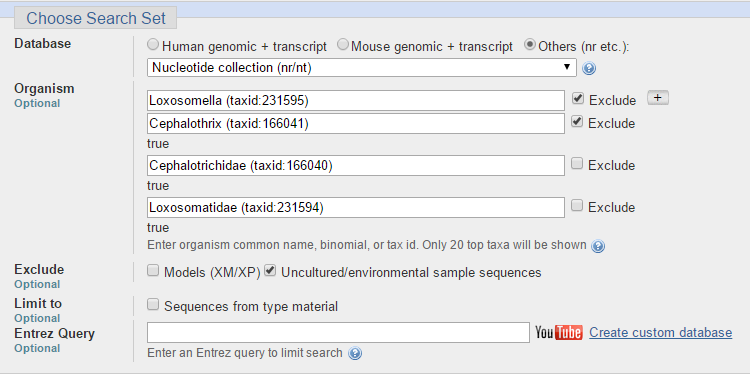
Рисунок 3. Параметры поиска.
Из следующих трех рисунков видно, что лучшие записи выдач всех трех алгоритмов одинаковы, однако одинаковые находки имеют разные показатели. В ряду алгоритмов megablast → discontiguous megablast → blastn число находок возрастает: просто добавляются находки похуже.

Рисунок 4. Выдача megablast.
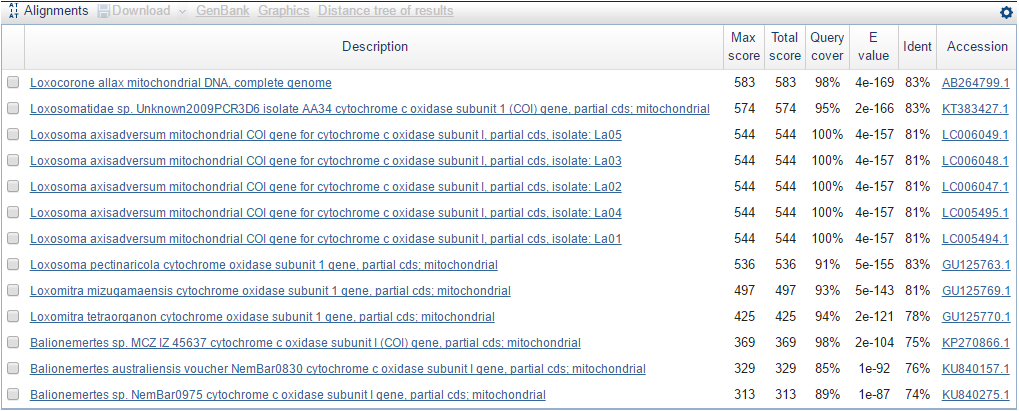
Рисунок 5. Выдача discontiguous megablast.
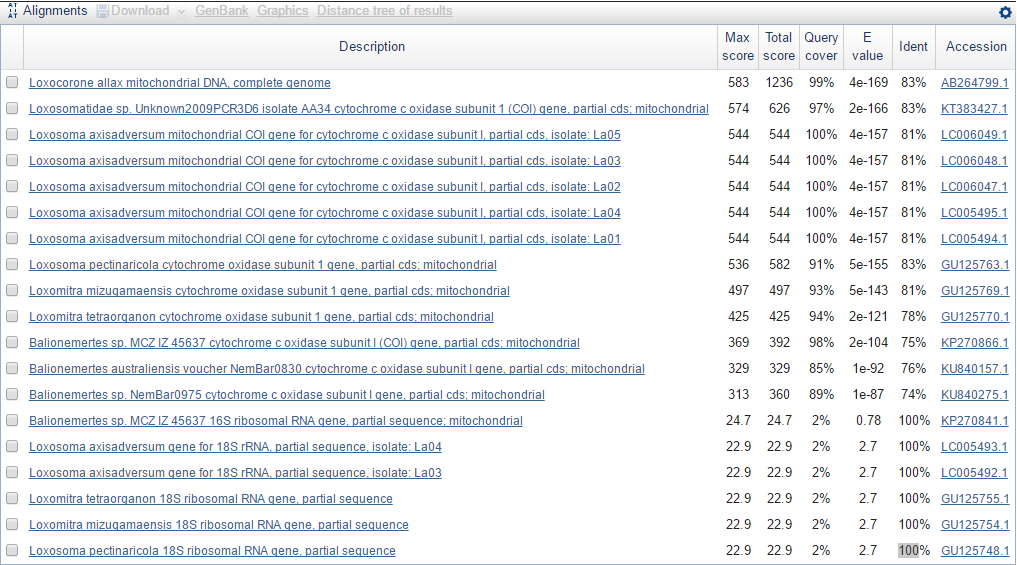
Рисунок 6. Выдача blastn.
3. Проверка наличия гомологов трех белков в геноме одного организма:
Был выполнен поиск гомологов трех белков у организма Amoboaphelidium protococarum при помощи алгоритма tblastn (результат).
Таблица 2. Описание находок.
| Название | Функция | Число находок | Лучшая находка | Ident лучшей | E-value лучшей |
| Citrate synthase, mitochondrial (CISY_HUMAN) | Участие в цикле Кребса, синтез цитрата | 4 | scaffold-693 | 69% | 2e-180 |
| Heat shock cognate 71 kDa protein (HSP7C_HUMAN) | Участие в фолдинге белков | 16 | scaffold-199 | 78% | 0.0 |
| Polyadenylate-binding protein 2 (PABP2_HUMAN) | Регуляция стабильности и трансляции мРНК | 16 | scaffold-100 | 63% | 2e-28 |
Параметры лучших находок для первых двух белков довольно неплохие, что позволяет предположить, что в геноме исследуемого организма закодированы белки, гомологичные данным. У лучшей находки третьего белка E-value достаточно большой, что позволяет предположить, что у исследуемого организма нету белков, гомологичных данному.
4. Один ген белка, закодированный в одном скэффолде "Amoboaphelidium":
Из списка скэффолдов был выбран scaffold-6. Затем, с помощью megablast с ограничением по Opistokonta и Fungi, был осуществлен поиск гена, закодированного в данном скэффолде.
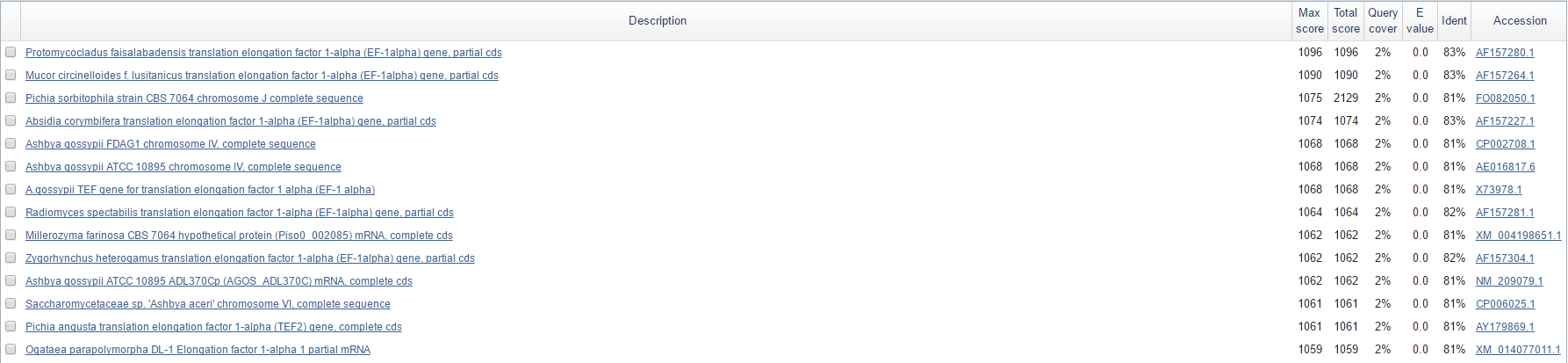
Рисунок 7. Выдача megablast.
Хорошие показатели находок и то, что они кодируют фактор элонгации трансляции 1-альфа 1, позволяют предположить, что именно этот ген закодирован в данном скэффолде.
Рисунок 8. Примерное расположение гена.
