Филогенетическое дерево 2.0
1. Построение дерева по нуклеотидным последовательностям
Нуклеотидные последовательности 16s rRNA были получены из базы полных геномов NCBI (из файлов с расширением .frn). Они были записаны в общий файл и выровнены с помощью сервиса Muscle. Полученное выравнивание было открыто в программе MEGA, где по нему было реконструировано филогенетическое дерево методом Maximum Likelihood.
 |
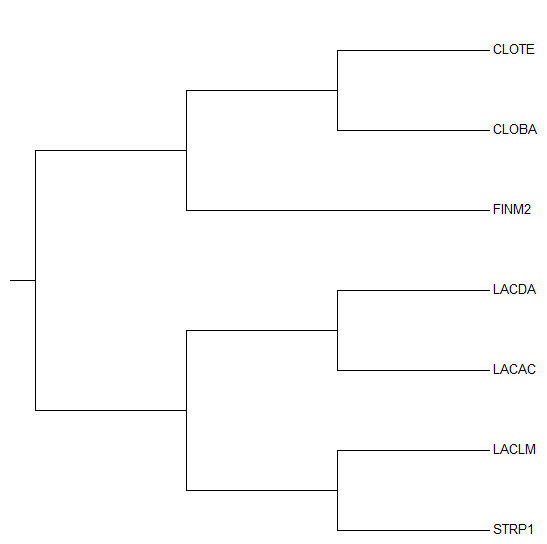 |
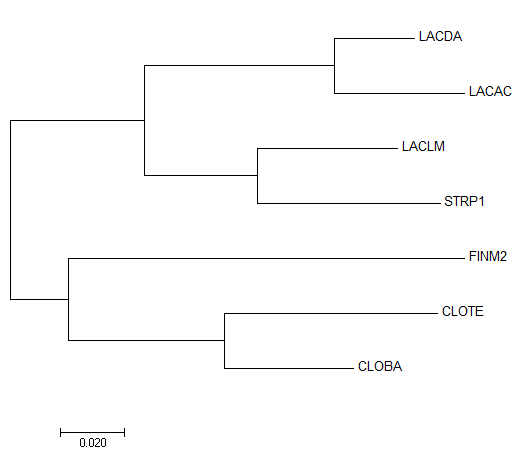
Рисунок 1. Филогенетическое дерево нуклеотидных последовательностей, полученное с помощью программы MEGA. |
Рисунок 2. Эталонное филогенетическое дерево. |
Полученное дерево полностью совпадает с эталонным, так что можно утверждать, что качество реконструкции по нуклеотидам лучше, чем по белкам.
2. Построение и анализ дерева, содержащего паралоги
В выбранных бактериях были найдены гомологи белка CLPX_BACSU с помощью BLAST'а (blastp) на сайте NCBI, устанавливая фильтр по организму, а в качестве банка — "nr". Из полученных последовательностей выбирались только достоверные гомологи , которые я затем объединил в один файл. Для удобства названия последовательностей были сокращены до мнемоники вида и идентификатора белка. Затем, белки были выровнены в программе MEGA, где затем было реконструировано филогенетическое дерево методом Maximum Likelihood.
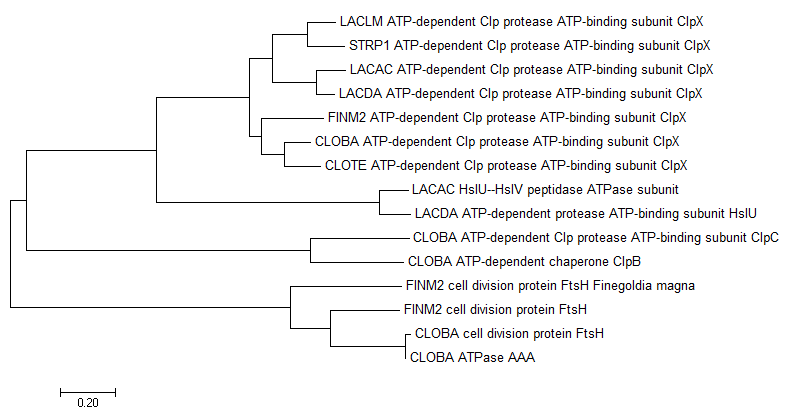
Рисунок 3. Дерево последовательностей гомологов, полученное с помощью программы MEGA.
Примеры ортологов
- FINM2_cell_division_protein_FtsH и CLOBA_cell_division_protein_FtsH
- FINM2_ATP-dependent_Clp_protease_ATP-binding_subunit_ClpX и CLOBA_ATP-dependent_Clp_protease_ATP-binding_subunit_ClpX
Примеры паралогов
- CLOBA_ATP-dependent_Clp_protease_ATP-binding_subunit_ClpC и CLOBA_ATP-dependent_chaperone_ClpB
- FINM2_cell_division_protein_FtsH и FINM2_cell_division_protein_FtsH_Finegoldia_magna
