Работа с KEGG ORTHOLOGY
1. Выбор пары ортологических рядов для дальнейшей работы
В базе данных KEGG был выбран метаболический путь Purine metabolism (метаболизм пуринов). В этом метаболическом пути была выбрана реакция [EC:3.6.1.5]: GTP -> GDP. Данную реакцию катализирует три ряда ортологических белков: K01510, K14641, K14642. Из них было выбрано два ряда с наименьшим количеством белков: K14641 (103, 99 UniProt), K14642 (33, 31 UniProt).
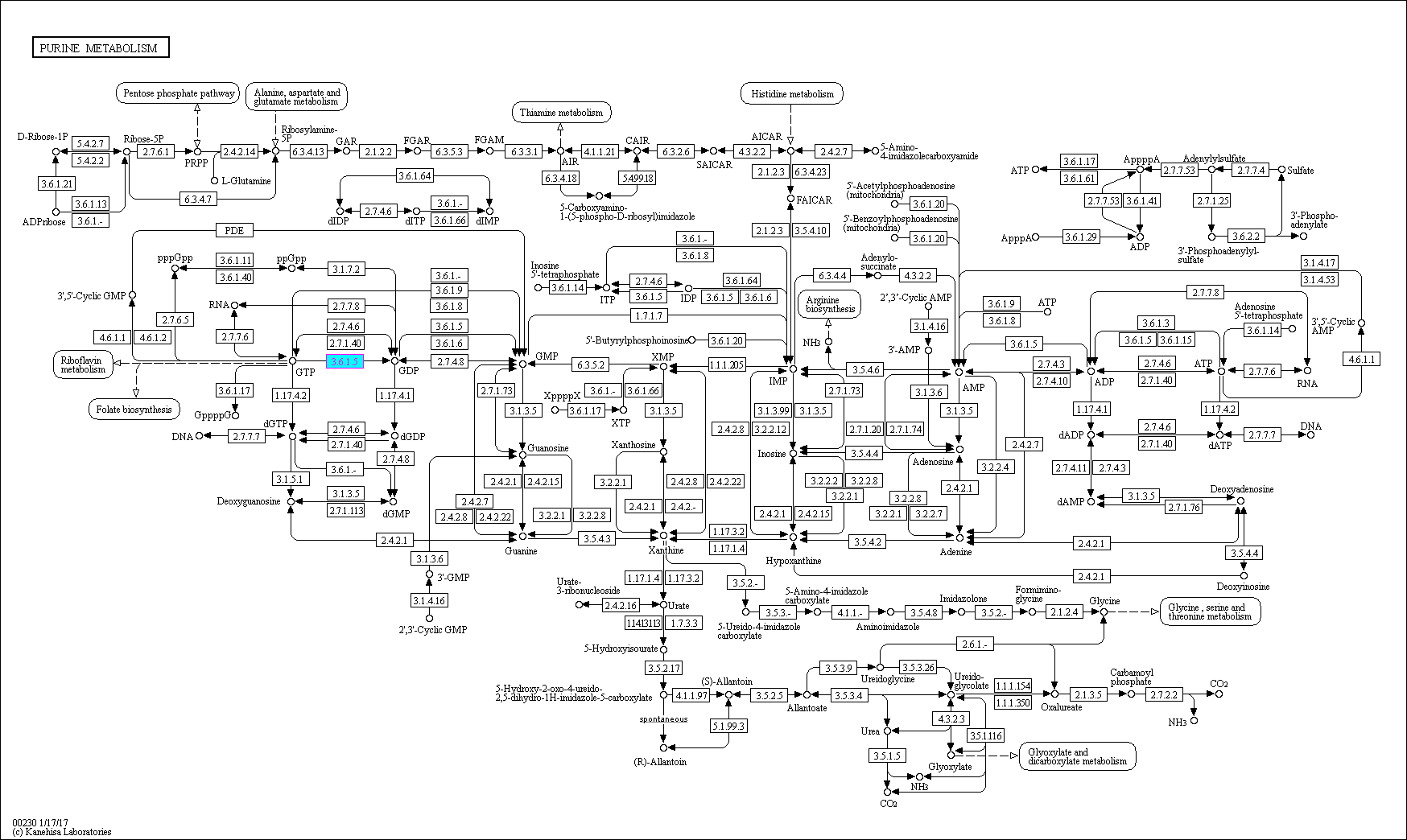
Рисунок 1. Метаболический путь Purine metabolizm с покрашенной выбранной реакцией.
2, 3. Получение совместного множественного выравнивания и проверка гомологичности белков в выравнивании
Сначала я получил последовательности белков из двух рядов с помощью сервиса Retrieve/ID mapping. Затем, с помощью скрипта на Python, в названия последовательностей были добавлены идентификаторы ортологических рядов; получились файлы K14641.fasta и K14642.fasta. После этого, было построено множественное выравнивание с помощью сервиса Muscle (align.fasta) в программе Jalview (align.jvp). Затем, из выравнивания были удалены некоторые белки, плохо выровненные с остальными или содержащие гэпы в консервативных колонках (22 белка, 20 из них из K14642). Полученное выравнивание: align_mod.fasta. Как видно из выравнивания, у белки являются довольно гомологичным.
4. Построение филогенетического дерева
В программе MEGA было построенно дерево методом Neighbor-Joining, со 100 бутстреп-репликами (дерево с расстояниями, консенсусное дерево). Дерево разлагается на две клады, соответствующие ортологическим группам, бутстреп поддержка этой ветви - 99. Заметна зависимость - чем длинне тривиальная ветвь, тем хуже выровнена последовательность.
