Практикум 10. Выравнивание геномов
В этом задании я решила выравнивать геномы представителей рода Planococcus, с одним из которых я уже работала при выполнении миниобзора. Для поиска последовательностей использовался сервис Browse by Organism на сайте NCBI. Запросы фильтровались по уровню сборки (хромосомная и выше). В итоге были выбраны виды: Planococcus faecalis AJ003 (CP019401) и Planococcus rifietoensis M8 (CP013659).
Карты DotPlot
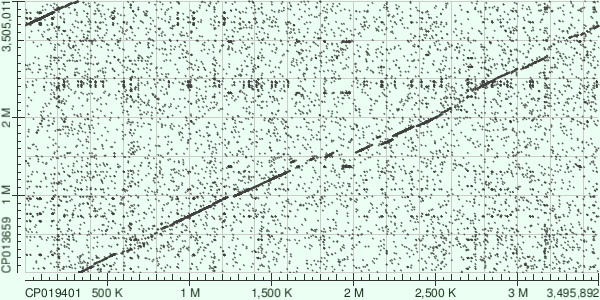
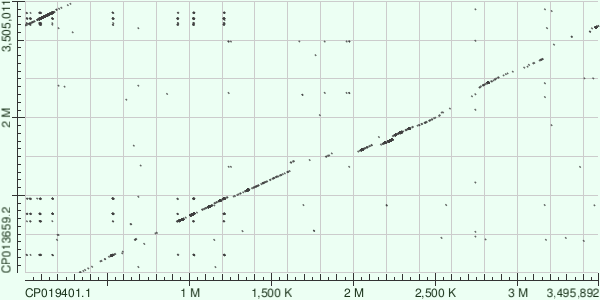
Заметны различия между этими двумя протоколами BLAST, их можно объяснить большей длиной слова megablast, из-за чего этот протокол пропускает более короткиее участки схожести. Карта локального сходства blastn имеет многочисленные повторы, которых нет на карте, полученной с помощью megablast. Сдвив диагонали в начале карт можно объяснить разными точками начала хромосом, выбранными иследователями. Небольшие сдвиги главной диагонали (многие из которых отсутствуют на карте megablast)скорее всего свидетельствуют о хромосомных делециях или вставках. Один участок (~2M по оХ) представляет особый интерес из-за его ориентации, это может быть как и вставка/делеция повторов, так и остатки инверсии. В целом геномы выравнились хорошо, организмы различаются относительно небольшим количеством перестроек.