Практикум 8. Нуклеотидный BLAST
Задание 1
Для работы в этом практикуме я решила взять участок четвертой хромосомы моего организма (Papaver somniferum). Ниже приведены данные о выбранном контиге:
Идентификатор: LOC113273001
Координаты: 110299918...110301318
Длина: 1401
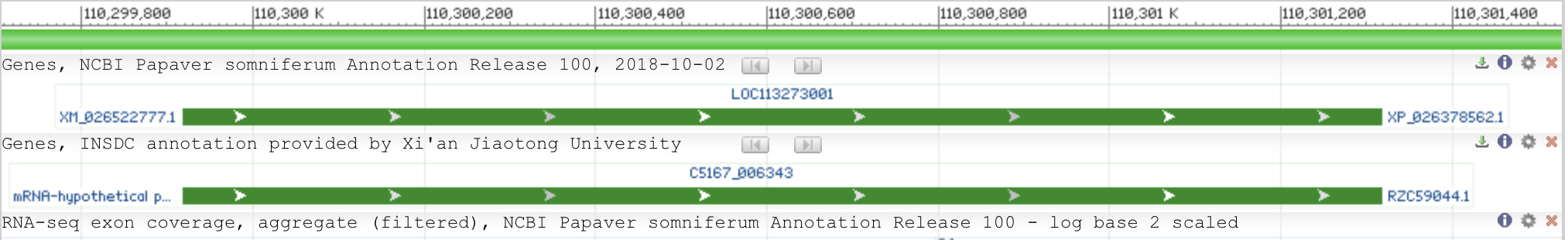
Файл с последовательностью контига
BLAST контига
megablast (word size = 28): Сначала я попробовала исключить цветковых, но пограмма не выдала значимых находок. При поиске среди цветковых находок 88. При исключении семейства Papaveraceae: 78, порядка Ranunculales: 56
blastn (word size = 11: С исключение цветковых: 59
blastx (word size = 6): С исключением цветковых: 100 (максимальное кол-во находок), с исключением Viridiplantae: 100 (при изменениее максимального числа до 5000 выдаёт 5000; белок митохондриальный...)
tblastx: Не хочет сотрудничать, даже при изменении параметров
Из проведенных опытов становится ясно, что мы имеем дело с контигом, в котором закодирован очень "популярный" у эукариот белок. В нашем случае наиболее рационально выравнивать именно по нуклеотидам, т.к. последовательность самого белка, видимо, достаточно консервативна.
Таким образом, можно можно сделать вывод, что в случаях, похожих на мой, blastn и megablast имеет смысл использовать для поисков близких родственников (blastn в группах более отдаленных, чем megablast). Blastx для поиска гомологии в крупных таксономических группах (если последовательность кодирует белок). Tblastx, видимо, лучше не использовать вообще, чтобы снизить количество связанного с работой стресса.
Задание 2
Последовательность РНК из генома снотворного мака, (полный геномный файл весит больше 2 Гб, и мне показалось крайне маловероятным найти значимые сходства вне РНК-овых последовательностей), я проиндексировала командой:
makeblastdb -in GCF_003573695.1_ASM357369v1_rna_from_genomic.fna -dbtype nucl -out db.fasta
По полученной базе данных я провела blastn (параметры по умолчанию) с последовательностями 16S и 23S рРНК кишечной палочки. Данный алгоритм был выбран т.к., препложительно, мак и бактерия достаточно отдалены друг от друга эволюционно и могут иметь значительные различия в пос-х рРНК. Для выравнивания использовались команды:
blastn -task blastn -query 16S.txt -db db.fasta -out 16S.out
blastn -task blastn -query 23S.txt -db db.fasta -out 23S.out
Для 16S нашлось несколько десятков потенциальных гомологов, из них с E-value от 6e-07 24 штуки. Из них лучшая находится в хлоропласте. Это логично, так как 16S у эукариот встречается в составе малых субъединиц рибосом органелл-производных прокариот (в т.ч. в плазмидах).
Для 23S рРНК было определено около 40 адекватных находок. Лучшая из них так же располагается в геноме хлоропласта по всё тем же причинам.