Практикумы 4.
1. Составление списка гомологичных белков, включающих паралоги
Файлы с протеомами семи бактерий были собраны в общий файл proteomes.fasta методом усердного копирования. Для поиска гомологов белка CLPX_ECOLI в моих бактериях был запущен blasp. Находки приведены в файле results.txt.
2. Реконструкция и визуализация
В файле fastas.fasta собраны последовательности находок. Используя данный файл было постоено дерево гомологов программой FastME c параметрами: "Gamma distributed rates across sites" — No, "Starting tree" — BIONJ, "No refinement", 100 бутстреп реплик. Полученное дерево в Newick-формате: fastas_tree.txt.
На полученном дерево можно выделить пары ортологов: CLPX MYCVP и CLPX RHOJR, CLPX STRAW и CLPX ACIC1, Q0S8C7 RHOJR и A1TG29 MYCVP
И пары паралогов: CLPX STRAW и Q82EB8 STRAW, A0LW31 ACIC1 и FTSH ACIC1, Q0S6Y7 RHOJR и Q0S8C7 RHOJR. Были получены следующие изображения деревьев:
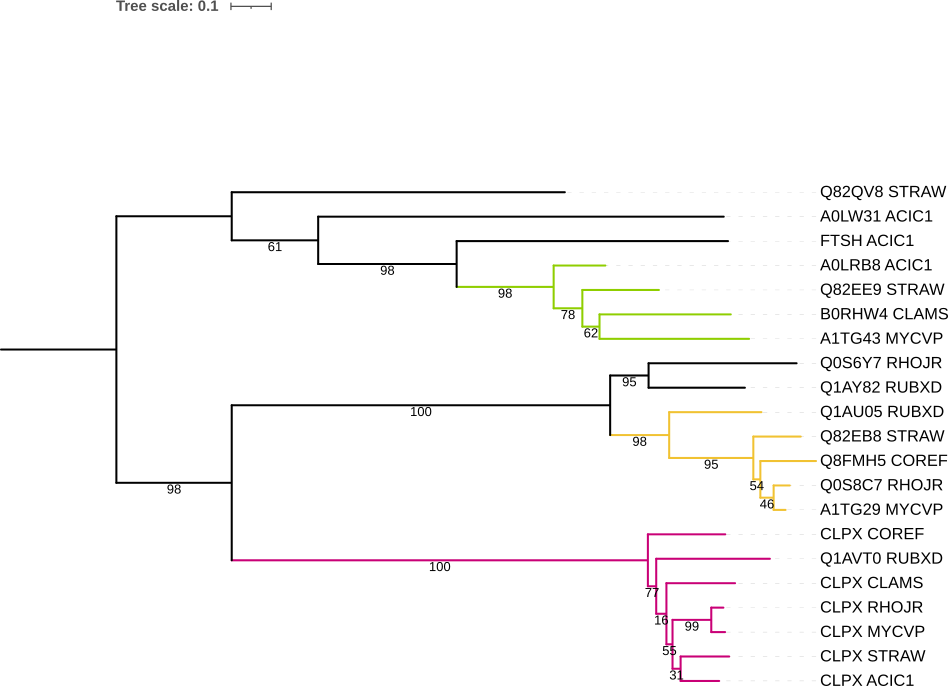
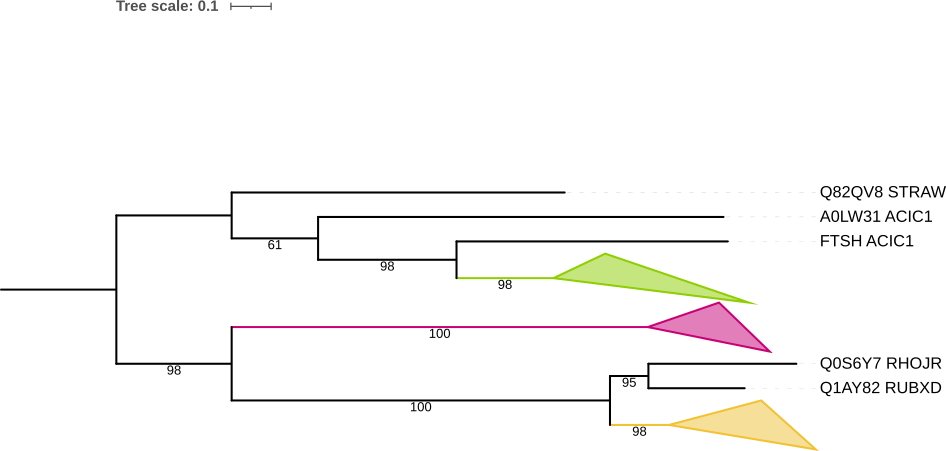
Группа, выделенная розовым: АТФ-зависимые Clp протеазы, в группу вошли все представители. Филогения не совпадает с бактериальной: COREF в последней входил в одну кладу с MYCVP и RHOJR, которая в свою очередь, одновременно с кладой CLAMS, STRAW & ACIC1, отделялась от RUBXD. Группа, выделенная жёлтым, содержит различные белки. Но её филогения совпадает с филогенией бактериального дерева. В зеленой группе находятся АТФ-зависимые цинковые металлопротеазы FtsH. Её филогения не совпадает с таковой бактериального дерева, в котором, среди прочего, CLAMS и MYCVP было расположены на разных ветвях.