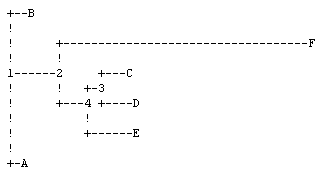Занятие 2.
Моделирование и реконструкция эволюции гена
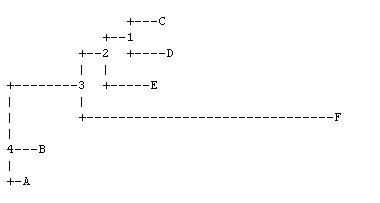
Модель судьбы моего гена в виде скобочной формулы:
(((А:10,B:10):30,((C:20,D:20):5,E:30):10):60,F:100);
Филогенетическое дерево:

Длины ветвей даны как число мутаций на 100 нуклеотидов гена.
Разбиение описываем в виде таблички, столбцы которой соответствуют листьям дерева, а строки - ветвям дерева. Дерево считаем бескорневым.
Внешние ветви описывать не имеет смысла(они отделяют только один лист от остальных).
A B C D E F
+ + - - - -
+ + - - + +
- - + + + -
Для получения "мутантов" использовали программу msbar пакета EMBOSS.
Составлен скрипт с учетом того, что последовательность 1273 нуклеотида.
Формула-число мутаций = длина гена*расстояние(длину ветвей)/100
Общая команда:
msbar infile outfile -point 4 -count n -auto
Комманды:
msbar SYY.txt F.fasta -point 4 -count 1273 -auto
msbar SYY.txt ABCDE.fasta –point 4 –count 763 –auto
msbar ABCDE.fasta AB.fasta -point 4 -count 382 –auto
msbar AB.fasta A.fasta -point 4 -count 127 -auto
msbar ABCDE.fasta CDE.fasta -point 4 -count 127 -auto
msbar CDE.fasta CD.fasta -point 4 -count 64 -auto
msbar CD.fasta C.fasta -point 4 -count 254 -auto
msbar CD.fasta D.fasta -point 4 -count 254 -auto
msbar CDE.fasta E.fasta -point 4 -count 382 -auto
msbar AB.fasta B.fasta -point 4 -count 127 –auto
Файл со скриптом scripta.txt Файлы с полученными последовательностями лежат в архиве Posledovatel'nosti-mutantu-ninja.zip
Реконструирование дерева алгоритмами UPGMA, Neighbor-joining и максимального правдоподобия
Создали файл со всеми последовательностями.
Далее с помощью, используя команды данные в указаниях к практикуму, создаем деревья.
Сначала был получен файл со всеми последовательностями листьев. Posledovatel'nosti-mutantu-ninja\VSE.txt
Сначала используем алгоритм максимального правдоподобия.
Для этого запускаем программу fdnaml
fdnaml VSE.txt -ttratio 1 –auto
gg\vse.fdnaml
Далее алгоритмы, основанные на оценке попарных расстояний:
Сначала мы посчитали попарные расстояния между последовательностями с помощью fdnadist.
fdnadist VSE.txt -ttratio 1 –auto
На выходе получили файл vse.fdnadist Posledovatel'nosti-mutantu-ninja\vse.fdnadist
После этого файл подаем на вход.
1.Neighbor-joining method:
fneighbor vse.fdnadist -auto (полученный файл был переименован в NJ.fneighbor)
gg\NJ.fneighbor
2. UPGMA( укорененное дерево):
fneighbor vse.fdnadist -treetype u –auto

H:\public_html\gg\vse.fneighbor
Укорененное дерево такое же как исходное, но не укорененные отличны.
Сравнение:
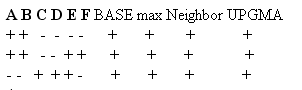
Анализ.
Алгоритм максимального правдоподобия является переборным. Дерево в данном случае получается неультраметраметрическим.
Агоритм Neighbor-joining является эвристическим. Работает с матрицей расстояний, полученной заранее. Дерево получается в данном случае неультраметрическим.
Алгоритм UPGMA является эвристическим. Работает с матрицей расстояний. Дерево получается укорененное ультраметрическое в виде кладограммы.
Топология всех деревьев совпадает, за исключением того, что дерево полученное UPGMA укорененное.
Деревья, созданные алгоритмами максимального правдоподобия и neighbor-joining одинаковы, единственное отличие в нумерации узлов. Наиболее точный результат выдал алгоритм UPGMA. Схожесть результатов может показывать на верность предсказанного эволюционного сценария. Сравнивать и говорить, что какой-то из алгоритмов ГОРАЗДО лучше другого нельзя, поскольку они все дали похожие результаты.