Задание 1. UCSC
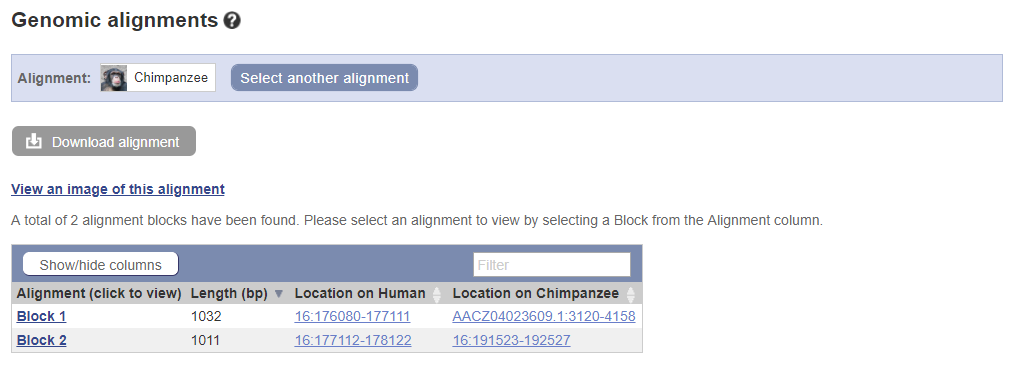
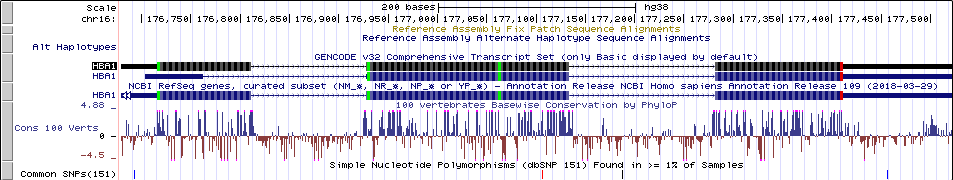
Для выполнения задания был выбран белок: hemoglobin subunit alpha 1. В таблице 1 указаны его параметры, таблицы 2 и 3 предназначены для альтернативных транскриптов. На рисунке 1 указана окрестность гена в Genome Browser с необходимыми трэками.
| Имя гена | HBA1 |
| Gencode ID | ENSG00000206172.8 |
| Цепь | + |
| Расположение | chr16:p13.3:176680-177522 |
| Альтернативных транскриптов | 2 |
| Gencode ID | ENST00000397797.1 |
| Координаты | chr16:176,704-177,522 |
| Число экзонов | 3 |
| Длина белка | 110 |
| Gencode ID | ENST00000320868.9 |
| Координаты | chr16:176,680-177,522 |
| Число экзонов | 3 |
| Длина белка | 142 |
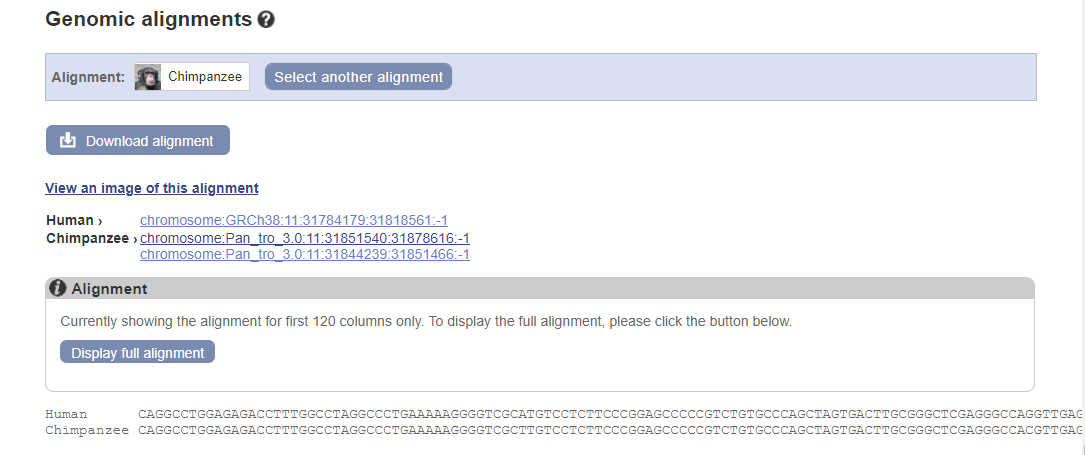
В Ensemble был найден человеческий ген HBA1, который был выровнен с геном шимпанзе (выравнивание прилагается). С помощью программы infoalign пакета EMBOSS была получена информация о различиях двух генов. Команда: infoalign H_C_al.fa -only -diffcount -seqlen -heading -name out. Результат:
# Name SeqLen Differ ENSP00000322421_Hsap_1-426 426 0 ENSPTRP00000067405_Ptro_1-426 426 3Таким образом мы получаем, что различаются лишь 0.7% нуклеотидов. Высокую схожесть можно было предвидеть еще при осмотре рисунка 1. Где в трэке SNP в экзоне встретился лишь один. Исследования показывают, что на полный геном человека и шимпанзе лишь 1.23% различных нуклеотидов. То что, в выбранном гене различия меньше различий по всему геному, возможно, говорит о высокой консервативности субъединиц гемоглобина.