Ход работы
- Работаем с 3 хромосомой в моей дирректории /nfs/srv/databases/ngs/aleksandrpavele
- Скачиваем chr3_1.fastq
- Анализируем качество чтений: fastqc chr3.1.fastq (Рис.1)
- Производим очищение чтений: java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr3.1.fastq trim3.1.fastq TRAILING:20 MINLEN:50
- Анализируем качество очищенных чтений: fastqc trim3.1.fastq (Рис.2)
- Картируем очищенные чтения на референс: hisat2 -x index3 -U trim3.1.fastq -S trim3.1align.sam --no-softclip
21152 reads; of these: 21152 (100.00%) were unpaired; of these: 154 (0.73%) aligned 0 times 20977 (99.17%) aligned exactly 1 time 21 (0.10%) aligned >1 times 99.27% overall alignment rate - Конвертируем выравнивания чтений в бинарный файл: samtools view -b trim3.1align.sam -o trim3.1align.bam
- Сортировка выравнивания: samtools sort trim3.1align.bam trim3.1alignsorted
- Индексируем отсортированные выравнивания: samtools index trim3.1alignsorted.bam
- Подсчет чтений: htseq-count -f bam -s no -i gene_id -m union trim3.1alignsorted.bam /nfs/srv/databases/ngs/Human/rnaseq_reads/gencode.v19.chr_patch_hapl_scaff.annotation.gtf > my_seq.count
- Ищем не нулевые строки: grep -w -v 0 my_seq.count > results.txt
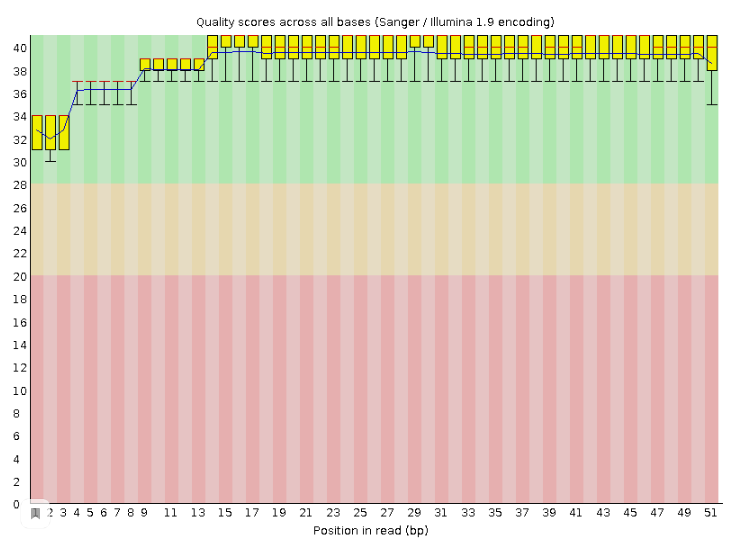
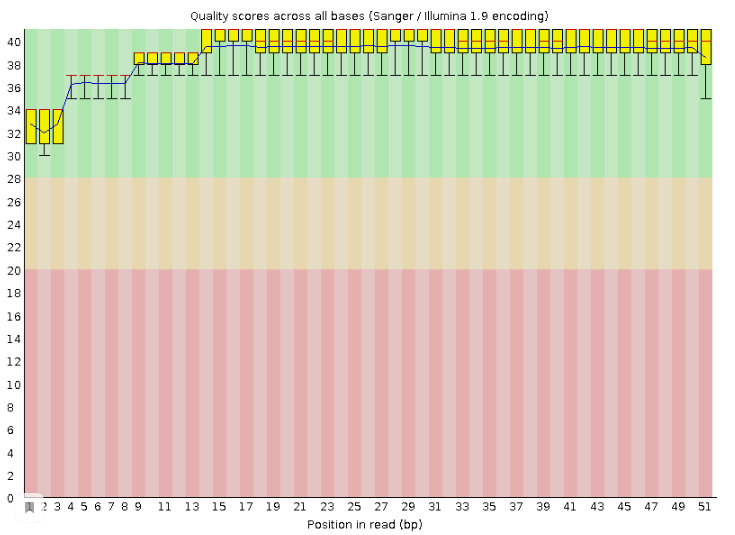
Отметим, что очистка никак не повлияла на качество чтений (разве что были удалены 59 ридов, видимо из-за параметра MINLEN:50) После картирования мы получили, что 99.17% чтений выравнялись на референс. Хоть в моих чтениях присутствуют риды, которые не легли на референс, а так же 21 рид, которые легли несколько раз, по-моему качество чтения хорошее. Кстати, в команде hisat2 был убран параметр --no-spliced-alignment, т.к. мы работаем с РНК, которая может быть задействована в сплайсинге, а значит выравнивания могут разрываться (участки перестанавливаться). Опции htseq-count: -f указывает формат входного файла, опция -s говорит, должна ли программа учитывать цепь, -i - как программа назовет гены, -m - как программа будет реагировать на разное наложение рида на ген/гены. Выдача grep:
ENSG00000072274.8 20495 ENSG00000252174.1 4 __no_feature 478 __not_aligned 154 __alignment_not_unique 42Из этого можно сделать выводы: 478 ридов не легли на границы генов, 42 чтения из-за -m union пересекаются сразу с несколькими генами, а 154 рида вообще не выровнились на референс. Помимо этого видно, что на ген ENSG00000072274.8 (TFR1 или ген рецептора трансферина человека), который учавствует в процессе эндоцитоза ионов железа из трансферрина в цитоплазму клетки, легло аж 20495 ридов. 4 же рида пересекаются с неэкспрессирующимся псевдогеном.