Задание
- Построить модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA
- Сравнение сгенерированной структуры одной из 3-х форм ДНК с со структурой той же формы полученной
экспериментальными данными
- Научиться находить большие и малые бороздки
- Определить, какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой
- С помощью MarvinSketch получите изображение основания, выделите красным цветом атомы, смотрящие в сторону большой бороздки, синим - в сторону малой
- Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
- Научиться определять торсионные углы нуклеотидов
- Определить значения торсионных углов в заданной структуре тРНК; определить, на какую из форм ДНК больше всего похожи тяжи этой структуры
- Научиться определять структуру водородных связей между основаниями
- Определить номера нуклеотидов, образующих стебли(stems) во вторичной структуре заданной тРНК
- Определить неканонические пары оснований в структуре тРНК
- Определить, есть дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру
Ход работы и результаты
Задание 1
С помощью пакета 3DNA, были построены 3 структуры (A-, B- и Z-формы ДНК)
Задание 2
С помощью программы Jmol, была заполнена таблица ниже и описано заданное азотистое основание (не нашел где они распределены, поэтому взял гуанин).
| A-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (нм) | 1.697 [20:A.P]-[24:B.P] | 1.721 [9:A.P]-[29:B.P] | 1.830 [28:B.P]-[10:A.P] |
| Ширина малой бороздки (нм) | 0.798 [27:B.P]-[8:A.P] | 1.169 [38:B.P]-[7:A.P] | 1.418 [38:B.P]-[8:A.P] |
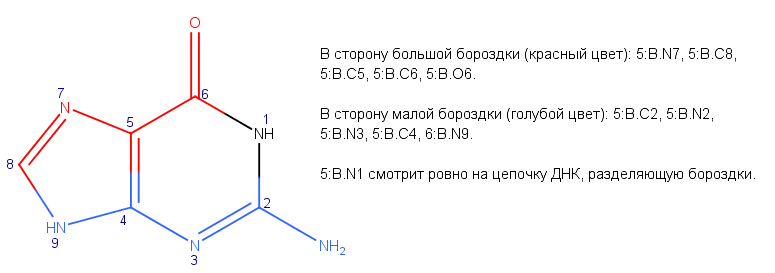
Задание 3
Так как распределение тРНК по ученикам я не нашел, я взял самую первую структуру, выдаваемую на сайте RCSB PDB (1G59). Сначала файл pdb тРНК были переведен в старый формат с помощью программы remediator. Все файлы полученные и используемые находятся в моей дирректории term3/block1/pr2. С помощью команды find_pair -t XXXX.pdb stdout | analyze были получены файлы XXXX.out в которых располагается информация о различных параметрах цепей РНК и ДНК, в том числе показатели торсионных углов. С помощью Excel были рассмотрены эти данные, что получилось указано в таблице 2. Было так же отмечено, что торсионные углы в Z-ДНК отличаются у C и G. Поэтому для них сделаны отдельные ряды в таблице.
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| тРНК | -66.35 | 56.25 | 51.85 | 92.5 | -152.05 | -74.7 | -156 |
| A | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B | -29.9 | 136.35 | 31.15 | 143.35 | -140.8 | -160.5 | -98.0 |
| Z(C) | -139.5 | -136.7 | 50.9 | 137.6 | -96.5 | 81.9 | -154.3 |
| Z(G) | 51.9 | 179.0 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
тРнк больше всего похожа и визуально на A-спираль ДНК, и ближе всего по показателям торсионных углов. Помимо этого были проанализированы водородные связи в структуре тРНК. Информация бралась из файла 1G59_old.out. x обозначает изменения в спирали. Хорошо видно 4 лепестка. Помимо этого + отмечаются изолированные пары, которые по видимому стабилизируют третичную структуру (хотя видимо нуклеотидные пары, которые отмечены x-ом тоже участвуют в стабилизации третичной структуры). Помимо этого * отмечаются не Уотсон-Криковские взаимодействия (интересно и то, что к ним относятся, например, и пары A-U, которые вроде как являются У-К, но возможно в данном случае они взаимодействуют не так как обычно друг с другом и водородные связи располагаются по-другому).
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.011) ....>B:.501_:[..G]G-----C[..C]:.572_:B<.... (0.010) |
2 (0.008) ....>B:.502_:[..G]G-*---U[..U]:.571_:B<.... (0.015) |
3 (0.011) ....>B:.503_:[..C]C-----G[..G]:.570_:B<.... (0.007) |
4 (0.010) ....>B:.504_:[..C]C-----G[..G]:.569_:B<.... (0.008) |
5 (0.010) ....>B:.505_:[..C]C-----G[..G]:.568_:B<.... (0.007) |
6 (0.011) ....>B:.506_:[..C]C-----G[..G]:.567_:B<.... (0.007) |
7 (0.012) ....>B:.507_:[..A]A-----U[..U]:.566_:B<.... (0.014) |
8 (0.007) ....>B:.549_:[..G]G-----C[..C]:.565_:B<.... (0.010) |
9 (0.006) ....>B:.550_:[..G]G-----C[..C]:.564_:B<.... (0.011) |
10 (0.007) ....>B:.551_:[..G]G-----C[..C]:.563_:B<.... (0.010) |
11 (0.007) ....>B:.552_:[..G]G-----C[..C]:.562_:B<.... (0.010) |
12 (0.009) ....>B:.553_:[..G]G-----C[..C]:.561_:B<.... (0.010) |
13 (0.016) ....>B:.554_:[..U]U-**--A[..A]:.558_:B<.... (0.008) |
14 (0.017) ....>B:.555_:[..U]U-**+-G[..G]:.517_:B<.... (0.015) x
15 (0.008) ....>B:.538_:[..A]A-**--C[..C]:.532_:B<.... (0.012) |
16 (0.009) ....>B:.539_:[..G]G-----C[..C]:.531_:B<.... (0.010) |
17 (0.012) ....>B:.540_:[..G]G-----C[..C]:.530_:B<.... (0.010) |
18 (0.012) ....>B:.541_:[..C]C-----G[..G]:.529_:B<.... (0.011) |
19 (0.011) ....>B:.542_:[..C]C-----G[..G]:.528_:B<.... (0.006) |
20 (0.008) ....>B:.543_:[..G]G-----C[..C]:.527_:B<.... (0.010) |
21 (0.007) ....>B:.544_:[..A]A-**--G[..G]:.526_:B<.... (0.006) |
22 (0.010) ....>B:.510_:[..G]G-----C[..C]:.525_:B<.... (0.010) |
23 (0.015) ....>B:.511_:[..U]U-----A[..A]:.524_:B<.... (0.013) |
24 (0.012) ....>B:.512_:[..C]C-----G[..G]:.523_:B<.... (0.007) |
25 (0.017) ....>B:.513_:[..U]U-*---G[..G]:.522_:B<.... (0.010) |
26 (0.014) ....>B:.508_:[..U]U-**--A[..A]:.546_:B<.... (0.008) |
27 (0.009) ....>B:.514_:[..A]A-**--A[..A]:.521_:B<.... (0.009) |
28 (0.008) ....>B:.515_:[..G]G-**+-C[..C]:.548_:B<.... (0.009) x
29 (0.008) ....>B:.518_:[..G]G-----C[..C]:.556_:B<.... (0.015) +
30 (0.009) ....>D:.501_:[..G]G-----C[..C]:.572_:D<.... (0.010) |
31 (0.010) ....>D:.502_:[..G]G-**--U[..U]:.571_:D<.... (0.016) |
32 (0.010) ....>D:.503_:[..C]C-----G[..G]:.570_:D<.... (0.008) |
33 (0.009) ....>D:.504_:[..C]C-----G[..G]:.569_:D<.... (0.006) |
34 (0.009) ....>D:.505_:[..C]C-----G[..G]:.568_:D<.... (0.009) |
35 (0.008) ....>D:.506_:[..C]C-----G[..G]:.567_:D<.... (0.008) |
36 (0.009) ....>D:.507_:[..A]A-----U[..U]:.566_:D<.... (0.015) |
37 (0.011) ....>D:.549_:[..G]G-----C[..C]:.565_:D<.... (0.012) |
38 (0.008) ....>D:.550_:[..G]G-----C[..C]:.564_:D<.... (0.010) |
39 (0.009) ....>D:.551_:[..G]G-----C[..C]:.563_:D<.... (0.014) |
40 (0.007) ....>D:.552_:[..G]G-----C[..C]:.562_:D<.... (0.011) |
41 (0.011) ....>D:.553_:[..G]G-----C[..C]:.561_:D<.... (0.009) |
42 (0.017) ....>D:.554_:[..U]U-**--A[..A]:.558_:D<.... (0.008) |
43 (0.019) ....>D:.555_:[..U]U-**+-G[..G]:.517_:D<.... (0.022) x
44 (0.011) ....>D:.538_:[..A]A-**--C[..C]:.532_:D<.... (0.013) |
45 (0.013) ....>D:.539_:[..G]G-----C[..C]:.531_:D<.... (0.008) |
46 (0.008) ....>D:.540_:[..G]G-----C[..C]:.530_:D<.... (0.009) |
47 (0.010) ....>D:.541_:[..C]C-----G[..G]:.529_:D<.... (0.008) |
48 (0.010) ....>D:.542_:[..C]C-----G[..G]:.528_:D<.... (0.006) |
49 (0.009) ....>D:.543_:[..G]G-----C[..C]:.527_:D<.... (0.012) |
50 (0.009) ....>D:.544_:[..A]A-**--G[..G]:.526_:D<.... (0.009) |
51 (0.010) ....>D:.510_:[..G]G-----C[..C]:.525_:D<.... (0.012) |
52 (0.018) ....>D:.511_:[..U]U-----A[..A]:.524_:D<.... (0.009) |
53 (0.011) ....>D:.512_:[..C]C-----G[..G]:.523_:D<.... (0.008) |
54 (0.016) ....>D:.513_:[..U]U-*---G[..G]:.522_:D<.... (0.007) |
55 (0.015) ....>D:.508_:[..U]U-**--A[..A]:.546_:D<.... (0.008) |
56 (0.010) ....>D:.514_:[..A]A-**--A[..A]:.521_:D<.... (0.010) |
57 (0.010) ....>D:.515_:[..G]G-**+-C[..C]:.548_:D<.... (0.008) x
58 (0.012) ....>D:.518_:[..G]G-----C[..C]:.556_:D<.... (0.032) +
Note: This structure contains 18[15] non-Watson-Crick base-pairs.