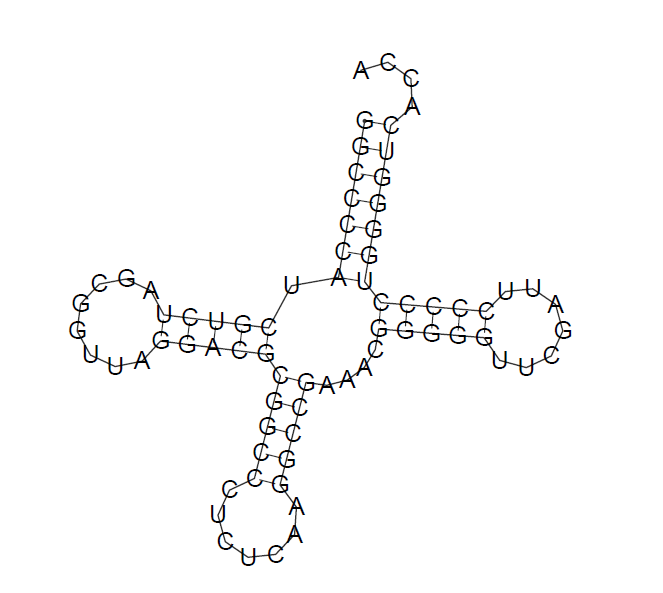
Задание 1. Упр.1 Предсказание вторичной структуры заданной тРНК
В моей дирректории была создана папка term3/block1/pr3. В ней будут располагаться все используемые и полученные файлы. тРНК была взята из прошлого практикума (1G59). С помощью программы einverted искались инвертированные участки в нуклеотидных последовательностях. Команда: einverted trna.fasta -gap 4 -thr 8. Выдача:
SEQUENCE: Score 14: 10/12 ( 83%) matches, 2 gaps
1 ggccccatcgtcta 14
|||| | || |||
31 ccggcgcag--gat 20
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
48 ggggg 52
|||||
64 ccccc 60
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps
3 cccca 7
|||||
69 ggggt 65
Задание 1. Упр.2 Предсказание вторичной структуры тРНК по алгоритму Зукера
Указываем путь и запускаем программу RNAfold:
export PATH=${PATH}:/home/preps/golovin/progs/bin
cat trna.fasta | RNAfold --MEA
ps2pdf 1G59\:B\|PDBID\|CHAIN\|SEQUENCE_ss.ps trna.pdf
Итого получаем pdf файл с предсказанной структурой тРНК.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'-501-572-3':5'-507-566-3' (7 пар) | 5 пар | 7 пар |
| D-стебель | 5'-510-525-3':5'-513-522-3' (4 пары) | - | 5 пар |
| T-стебель | 5'-549-565-3':5'-553-561-3' (5 пар) | 5 пар | 5 пар |
| Антикодоновый стебель | 5'-539-531-3':5'-544-526-3' (6 пар) | - | 5 пар |
| Общее число канонических пар нуклеотидов | 20 | 20 | 20 |
Задание 2 Поиск ДНК-белковых контактов в заданной структуре
Упражнение 1
Скрипт-файл с определенными множествами. Скрипт-файл с последовательными изображениями.
Упражнение 2
Для этого задания был переделан скрипт для define дополнительных групп атомов, например, атомов смотрящих в сторону большой и малой бороздок.(скрипт). Потом производим манипуляции с select и within, например, для подсчета числа полярных взаимодействий с остатками 2'-дезоксирибозы используем: select *.?*' and polaratom and within(3.5, polaratom and protein). Информация о контактах представлена в таблице 2.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 11 | 65 | 76 |
| остатками фосфорной кислоты | 18 | 14 | 32 |
| остатками азотистых оснований со стороны большой бороздки | 0 | 7 | 7 |
| остатками азотистых оснований со стороны малой бороздки | 11 | 11 | 22 |
Упражнение 3
Перед началом файл был переведен с помощью команды remediator в старый формат pdb. Потом с помощью команды nucplot был получен файл с изображением связей между ДНК и белком.
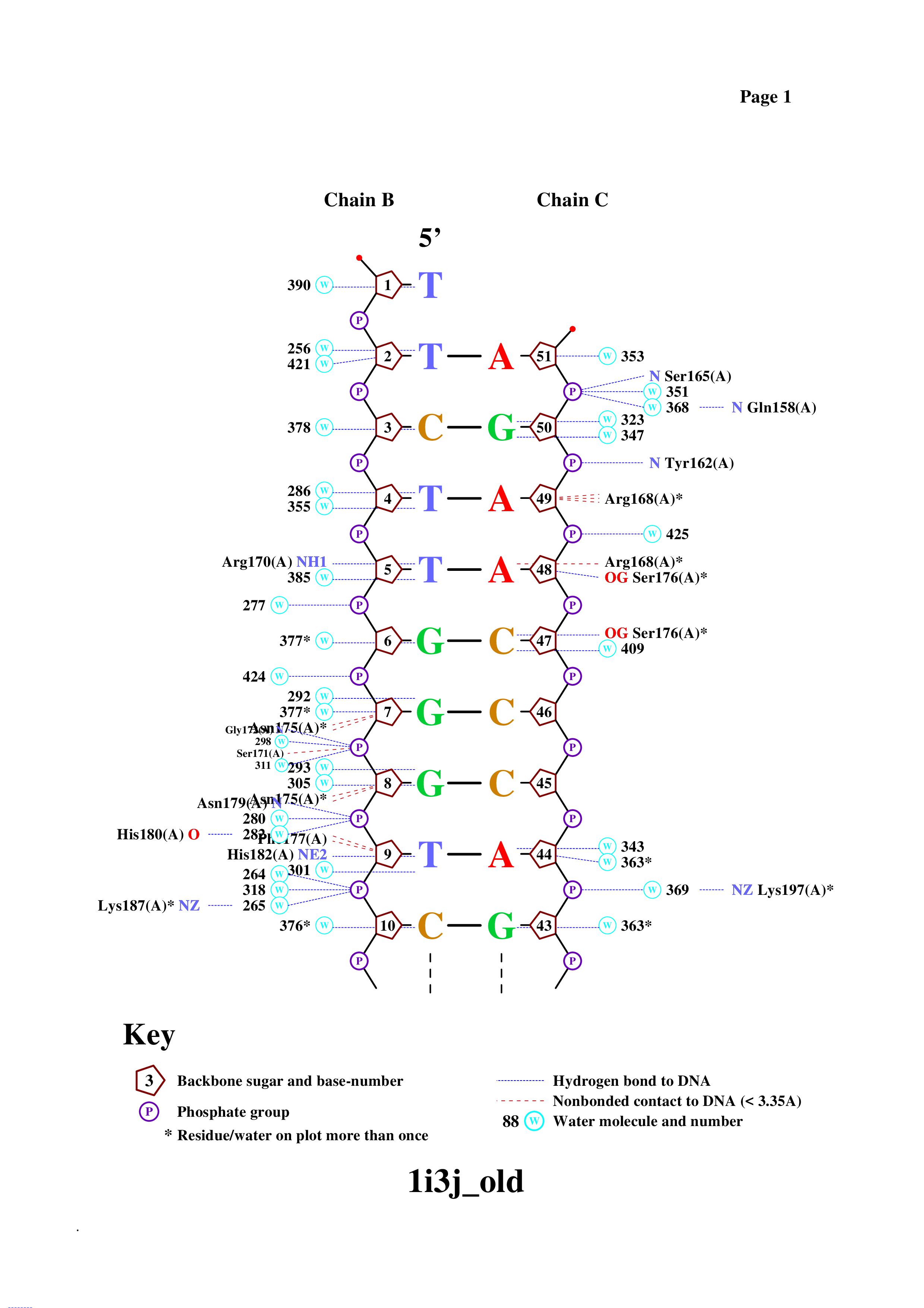
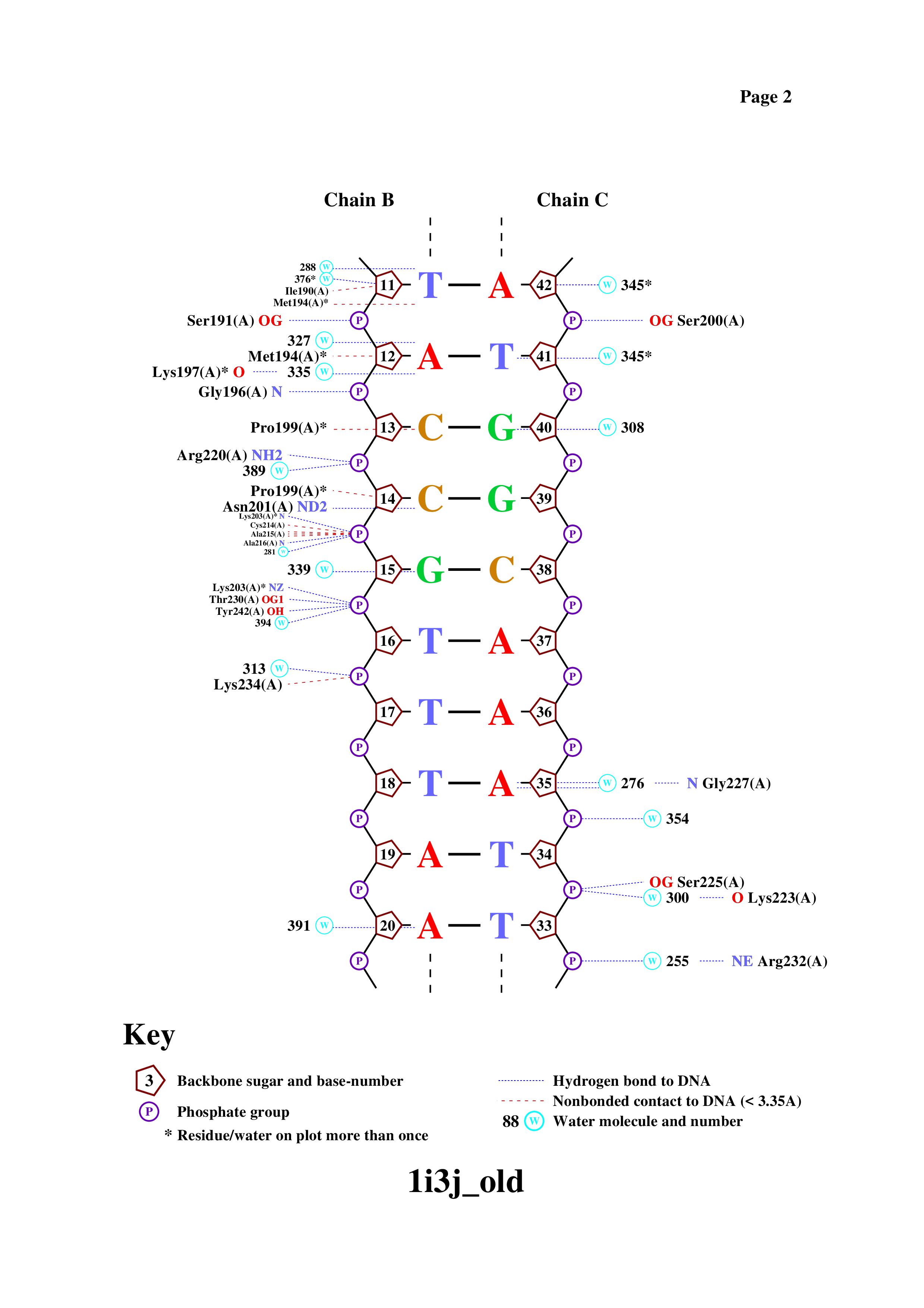
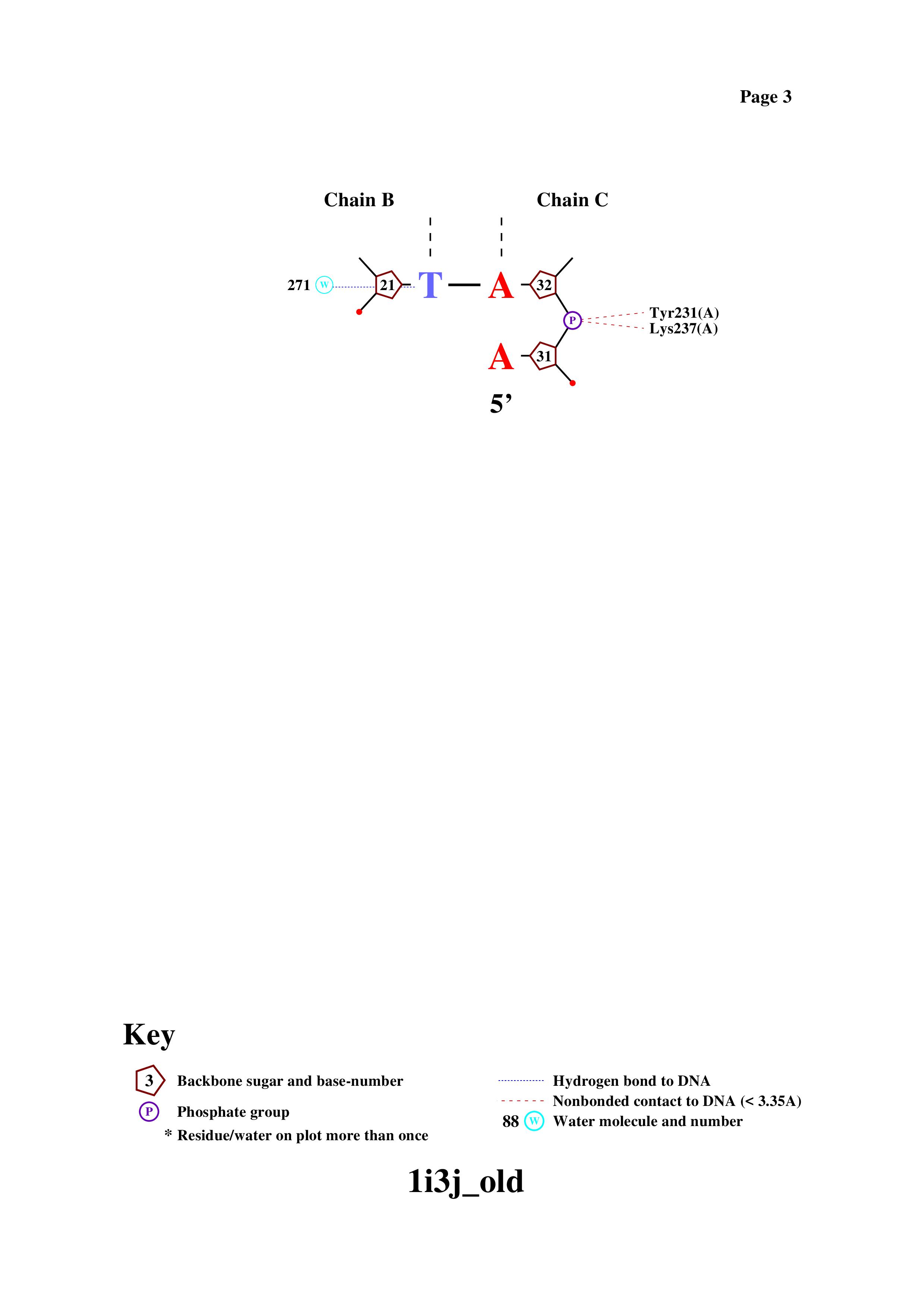
Упражнение 4
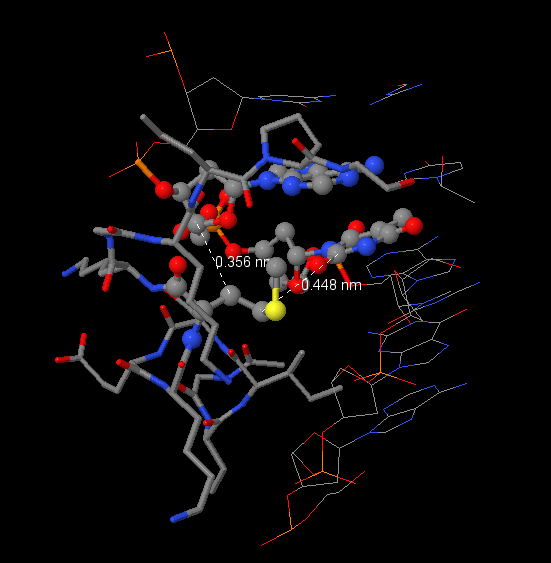
Для выполнения пункта а) я решил выбрать аминокислоту ARG168. Из рисунка 2 можно увидеть, что он имеет 3 контакта с 49 сахаром ДНК и 48 аденином. На рисунке 5 отображены эти контакты (по длинам они подходят под наше определение неполярного контакта).
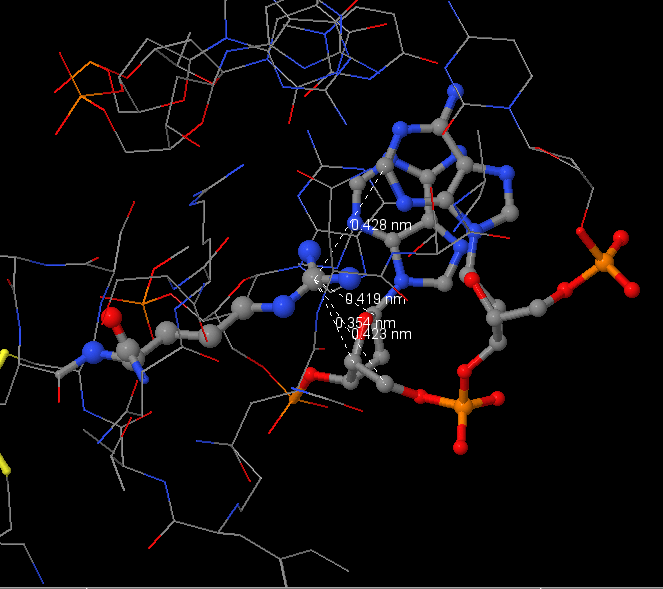
Для выполнения пункта б) я решил призадуматься. По-моему, логично предположить, что для хотя бы небольшой точности распознования необходимо подряд распозновать хотя бы от трех и более нуклеотидов. У меня на рисунке 3 как раз есть участок ДНК (TACC), из которых каждый нуклеотид имеет контакты с аминокислотами белка (помимо этого в этом участке также хорошо заметно распознавание, точнее контакты и с фосфатами между этими нуклеотидами). Так вот среди всех контактов с этим участком выделяется MET194, который контактирует и с нуклеотидом и сахаром. Поэтому, я думаю что эта аминокислота основопологающая.