Получение последовательности ДНК на основании данных, полученных из капиллярного секвенатора. Составление отчёта о проблемах при чтении хроматограмм
Ход работы
- С диска P из директории P:\y18\term3\block2\ab1_files\good были скачаны два файла: 37_F.ab1 и 37_R.ab1, который соответствуют прочтению прямой и обратной цепочки секвенируемой ДНК
- Для работы с хроматограммами была установлена программа GeneStudio и Chromas (т.к. по непонятной причине GeneStudio при выравнивании двух хромотограмм не рисовал до конца хромотограммы, но при этом писал последовательность). Первая программа умеет выравнивать и обрезать трэш-концы.
- Сначала прямая и обратная последовательности были открыты в GeneStudio, где они были выравнены и обрезаны (Chromas тоже умеет обрезать последовательности но с меньшей точностью, например, 37_F.ab1 он обрезал аж до 126 нуклеотида включительно, хотя последовательность уже с 49 нуклеотида выглядит хорошо)
- Хроматограммы были проанализированны на их качество
- Редактирование прямой и обратной последовательности (все исправления маленькой буквой)
- Выравнивание двух последовательностей и сохраниение выравнивания в формате fasta
- Приведены примеры нечитаемых фрагментов хроматограммы
- Создание скрипта для автоматической обработки двух хроматограмм
Пояснения и результаты
Задание 1
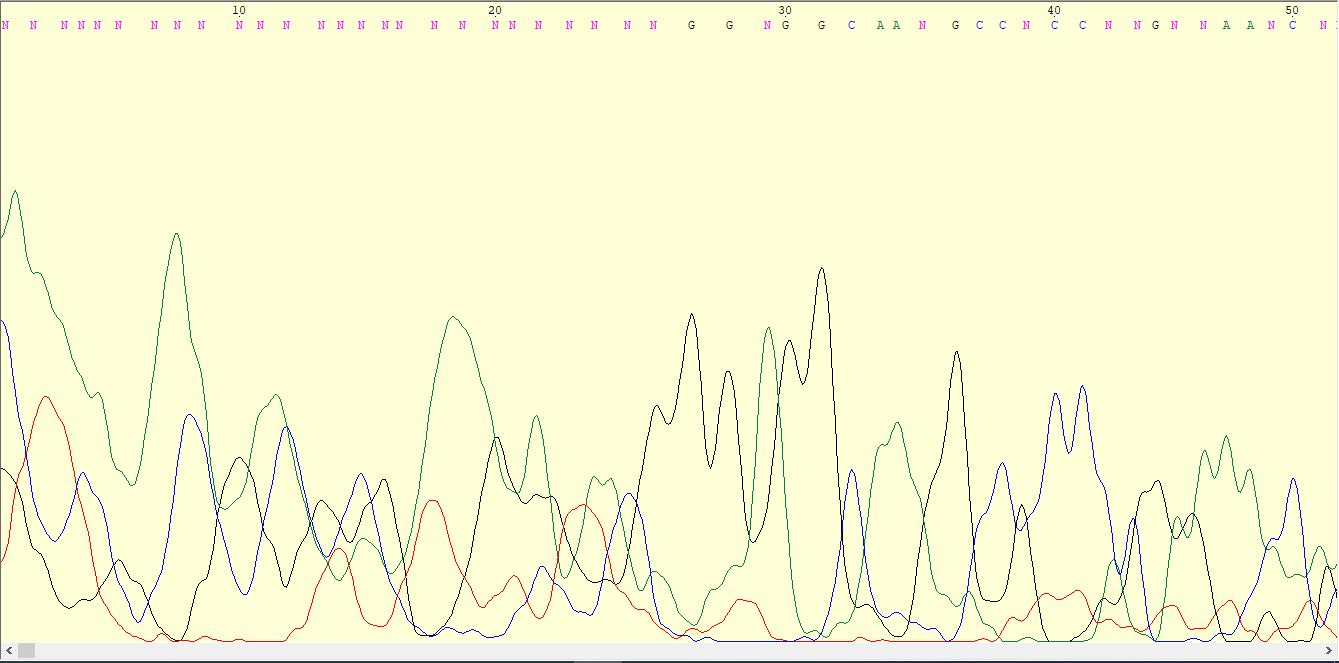
Файлы для анализа: 37_F.ab1 и 37_R.ab1. 37_F.ab1 был обрезан до 49 нуклеотида и отрезаны с 377 включительно (для конца последовательности обе программы дали одинаковый результат)
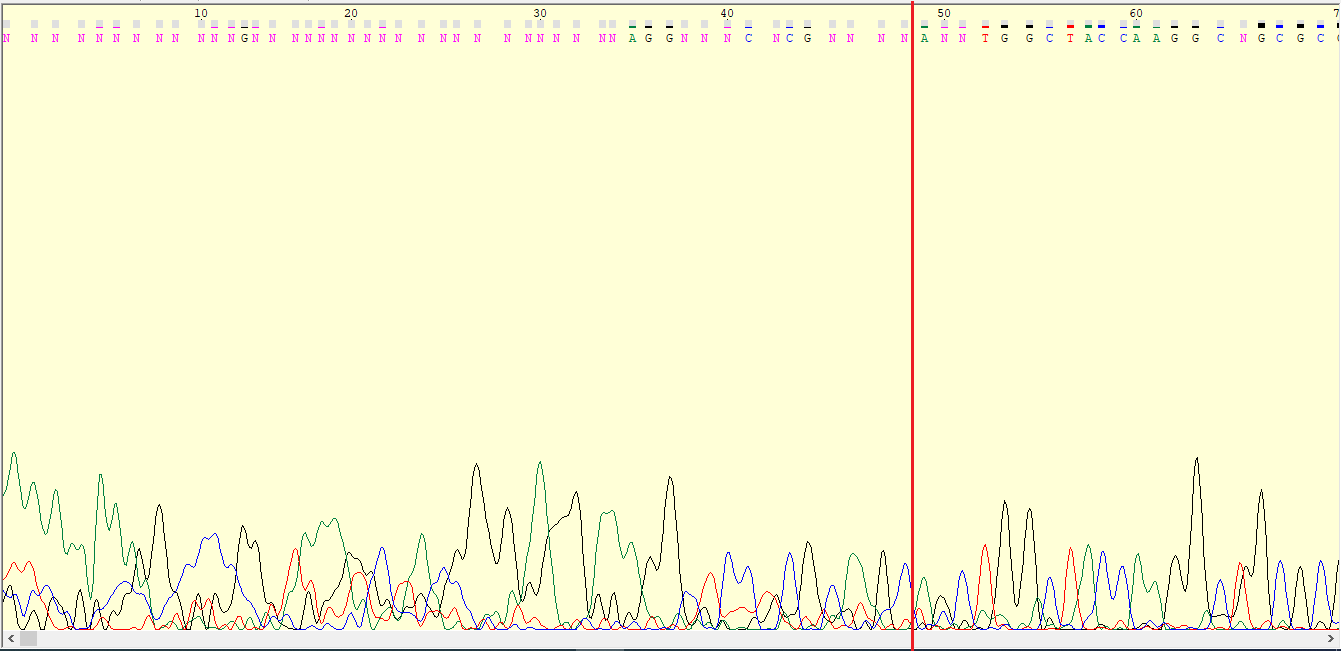
37_R.ab1 был обрезан до 3 (Chromas обрезал до 19) нуклеотида и отрезаны по моему мнению с 346 (Chromas предложил с 286, а GeneStudio с 326)
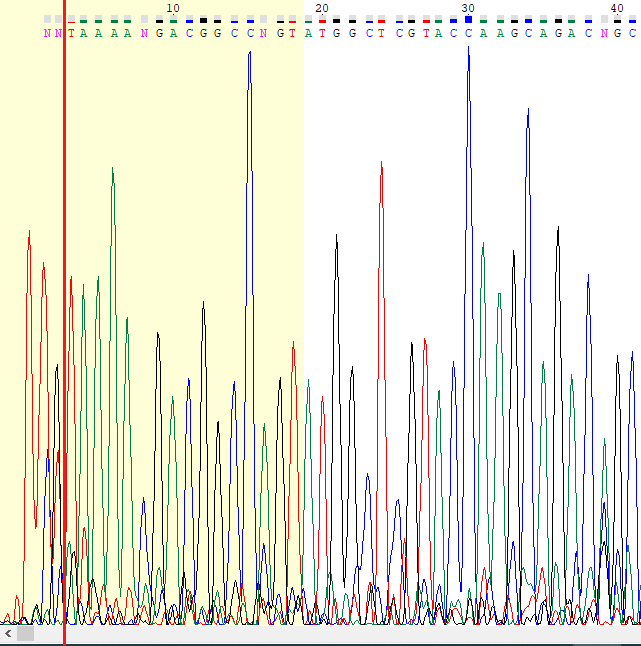
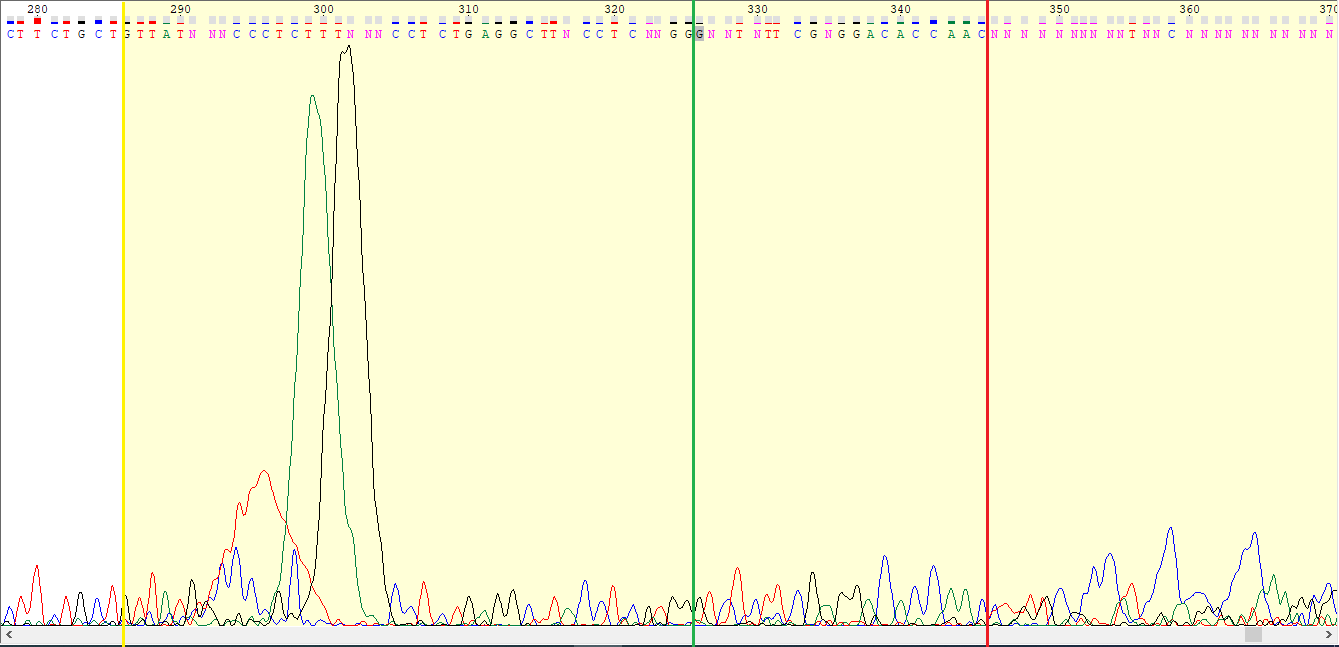
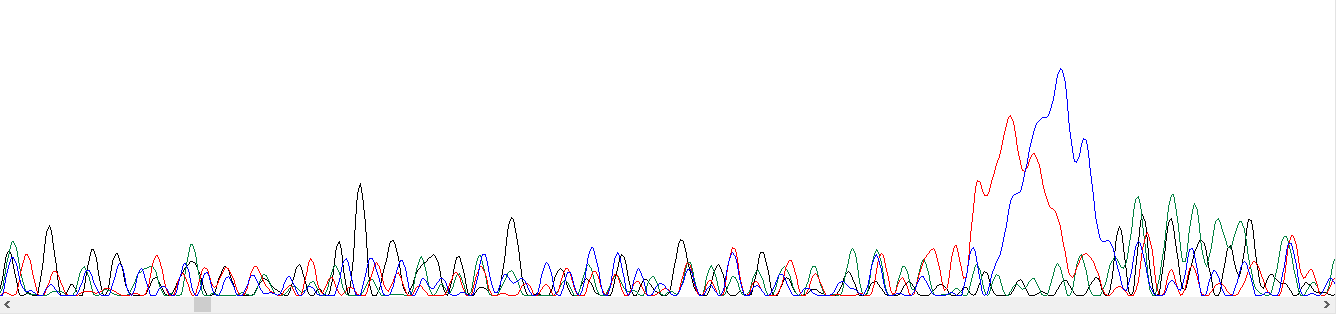
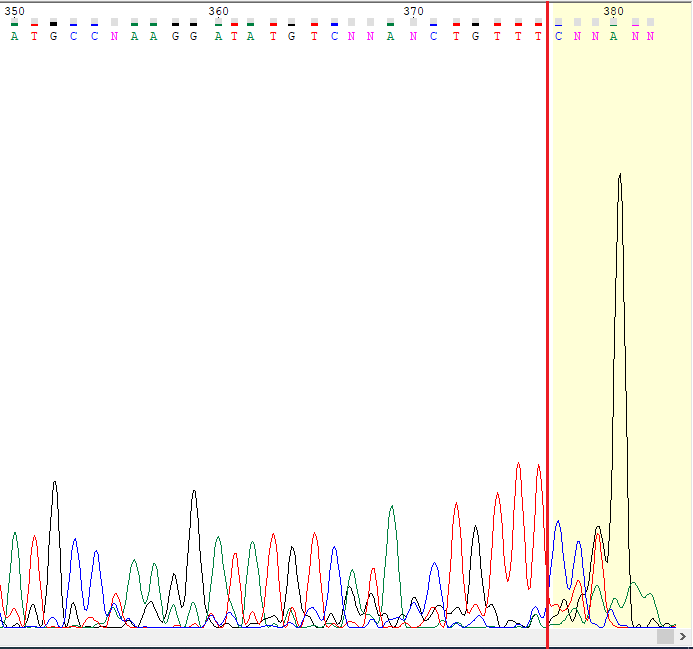
В дальнейшем хроматограммы были проанализированны на их качественность. При удалении концов с плохими пиками хроматограмма выглядит хорошо (низкие пики шумов) кроме некоторых выделяющихся мест, например, на рис.5 и 6.
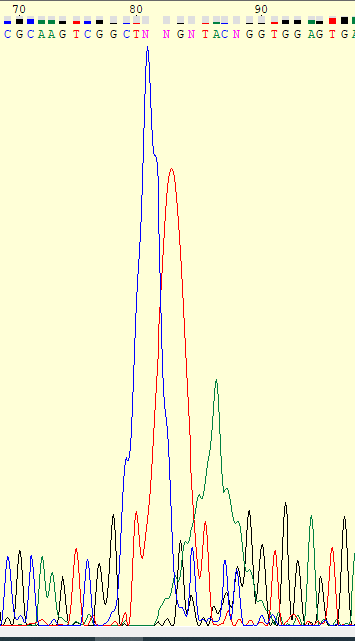
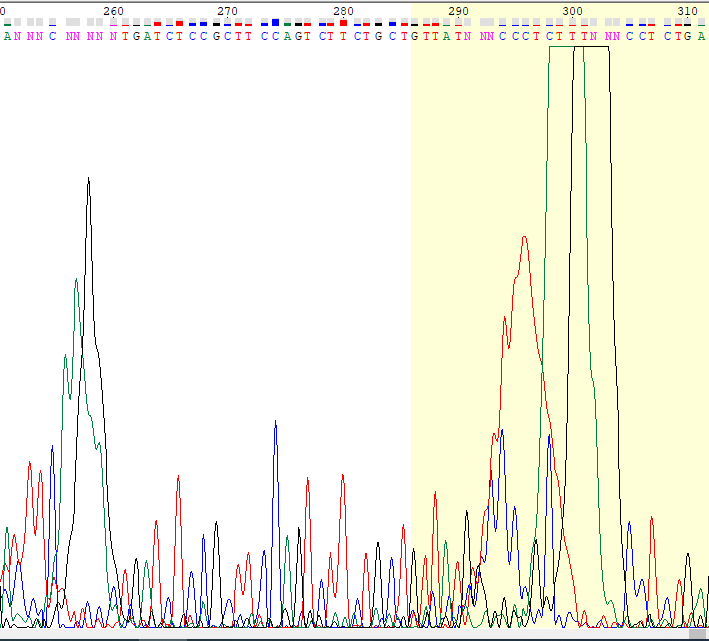
Автоматизацию процесса анализа качественности хроматограммы можно сделать по такому принципу: выделить все "хорошие" пики и посчитать их среднюю высоту, после чего сравнить ее со средней высотой пиков шумов, и, если второе не превышает первого, то такая хроматограмма хорошая, если превышает, то наоборот. Тоже самое можно проделать и с площадями.
Далее в программе GeneStudio было просмотренно выравнивание обрезанных последовательностей, для исправления каждой из них. При этом возникали и спорные моменты, которые приходилось проверять отдельно (GeneStudio автоматически создает из выравнивания прямой и обратной последовательности консенсусную последовательность, в которую уже вводит свои изменения, однако, на некоторых местах он вставлял ИЮПАКовские символы неопределенности, в моем случае Y - T или С, W - A или T, что продемонстрированно на рисунках ниже). К сожалению, последовательности не покрыли друг друга полностью, поэтому в них останутся неопределенные места.
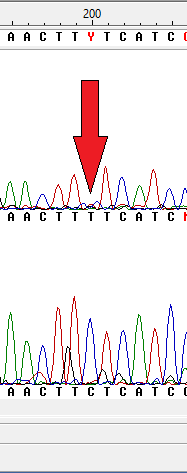
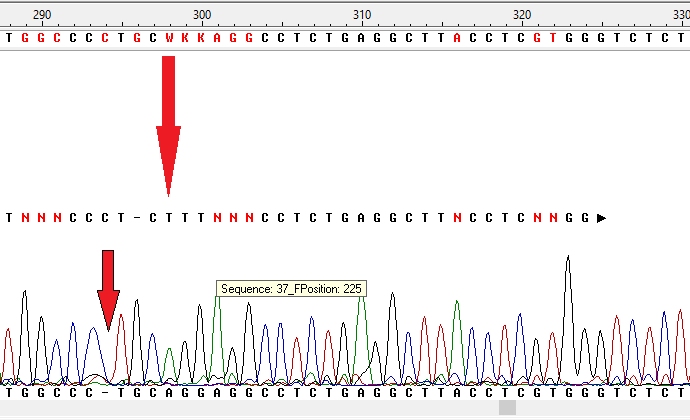
В итоге были сделаны fasta-файлы прямой и обратной последовательности, в которых отсутствуют трэш-концы и исправлены некоторые места в последовательности. Полученные последовательности были выравнены и само выравнивание было сохранено в fasta-формате.
Задание 2
На рисунках 5 и 6 указаны места в хроматограмме, которые похожи на пятна краски. Помимо этого, были просмотрены файлы NN_G10 и WSWS2950_H3_F_G09_2013-06-11-22-39-58 из дирректории bad. На первой хроматограмме помимо пятен краски, нет четко выраженных пиков, т.к. все они не отличимы от шума. На рисунке 9 видна эта проблема. На рисунке 10 хорошо отображена проблема с анализом начала хроматограмм.