Проведем итеративный поиск программой PSI-BLAST по банку Swiss-Prot (http://blast.ncbi.nlm.nih.gov/) для четырёх аминокислотных последовательностей: P18196, P0A832, P17265 и моего белка – P80643 (ACP_BACSU).
Сменим значение параметра "Max target sequences" с 500 на 5000, остальные параметры сохраним по умолчанию. Выполним до пяти итераций, пока появляются новые последовательности выше порога E-value 0,005.
Результаты занесем в таблицу:
| ID белка | AC белка | Число итераций | Для первой итерации | Для последней итерации | ||||
| Число находок выше порога (0,005) | Худшее E-value выше порога | Лучшее E-value ниже порога | Число находок выше порога (0,005) | Худшее E-value выше порога | Лучшее E-value ниже порога | |||
| MINC_ECOLI | P18196 | 6 | 165 | 0.004 | 0.005 | 995 | 9е-04 | 0.099 |
| SSRP_ECOLI | P0A832 | 2 | 514 | 3е-12 | 4.9 | 514 | 3е-38 | 0.42 |
| Y380_RHIME | P17265 | 4 | 15 | 7е-04 | 0.027 | 25 | 3е-18 | 0.024 |
| ACP_BACSU | P80643 | более 5 | 364 | 0.004 | 0.012 | 509 | 0.005 | 0.005 |
На примере этих четырех последовательностей можно сделать следующие замечания:
"разрыв" между значениями E-value у худшей находки выше порога и лучшей – ниже порога как правило
растет с количеством итераций,
но важнее, что итоговый "разрыв" должен быть значительным (чтобы не "подцепить"
новые последовательности).
если пороговый E-value подобран не правильно, то "разрывы" выше и ниже него минимальны, список не
стабилизируется.
Теперь подробнее рассмотрим каждый из случаев:
В первом поиске (P18196) "разрыв" то увеличивался, то уменьшался от итерации к итерации, удерживаясь на уровне 0.001-0.002 (то есть фактически отсутствовал) в запросах 1, 3, 4. В итерации 2 разрыв составил 0.062, итерации 5 он резко возрос до крайних значений 7е-04/0.023, что почти не отличается от итогового результата.
Во втором поиске (P0A832) "разрыв" сразу был очень большим и вырос еще больше (значения из таблицы).
В третьем поиске (P17265) "разрыв" сначала был большим (7е-04/0.027) и увеличивался (5е-09/0.011), затем после третьей итерации добавилась 1 последовательность, а разрыв упал (0.002/0.012), но поиск сразу прекратился, разрыв снова подскочил до пары 3е-18/0.024.
В четвертом поиске (P80643) разрыв почти не колебался, уменьшившись сразу с 0.008 до 0.001-0.003.
Этот список не стабилизировался даже на шестой итерации.
E-value лучшей находки во всех случаях ухудшался (рос) от итерации к итерации пропорционально количеству прибавленных последовательностей: в первом – от 8е-171 до 3е-82 (для шестой итерации), во втором – от 3е-116 до 1е-94, в третьем – от 7е-141 до 1е-71, а в последнем – от 4е-46 до 1е-23.
Также хочется отметить, что словосочетание "лучшая находка" не равносильно термину "исходная последовательность", хотя они почти всегда совпадают. Контрпримером является последний поиск, где уже после второй итерации последовательность P80643 (ACP_BACSU) уступила первую строчку другим последовательностям, E-value тоже был хуже. Это можно объяснить тем, что после каждой итерации создается новый профиль для выравнивания всех выбранных последовательностей, а в нем добавленные последовательности могут "задавить" исходную (но не очень сильно).
Об этом же говорит тот факт, что во всех случаях у случайно выбранной "средней" последовательности E-value, в целом значительно уменьшаясь (улучшаясь), колебался, так как добавляемые последовательности изменяли профиль выравнивания.
Для последовательностей, у которых список не стабилизировался после пятой итерации,
проведем поиск снова, изменив порог с 0,005 на 0,001 (как и рекомендуется, для разнообразия воспользуемся сервисом PSI-BLAST на EBI:
http://www.ebi.ac.uk/Tools/psiblast/,
где порог 0,001 стоит по умолчанию).
Сначала разберемся с последовательностью №1 (P18196).
Я провел параллельный поиск в PSI-BLAST на EBI и на NCBI. Поиск велся при одинаковых параметрах и в обоих случаях сошелся уже на третьей итерации. Однако при этом количество находок серьезно различалось: 264 на EBI против 188 на NCBI. Также различался и "разрыв": 1.0е-10/0.014 на EBI и 4е-12/0.001 на NCBI.
Скорее всего, причиной столь сильных различий в поиске является различие программы, создающей матрицу профиля и, таким образом, отсекающей лишние последовательности.
Стоит заметить также, что в PSI-BLAST на EBI все последовательности, оказавшиеся выше порогового
значения, имели общую часть названия: MINC_*, то есть относились к одной семье белков MinC family.
"Разрыв" E-value на этот раз не колебался, а сразу пошел вверх с пары 0.0010/0.0020 до 1.0е-6/0.025 и 1.0е-10/0.014
во второй и третьей итерациях.
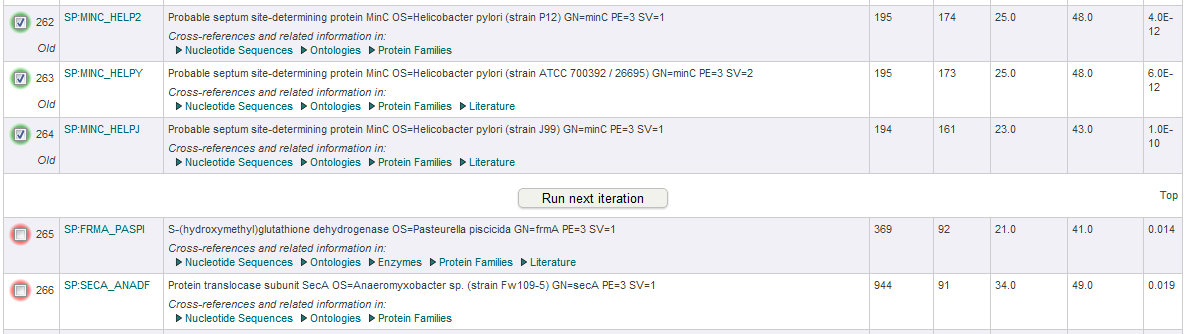
Так выглядит список поиска в месте "разрыва" на EBI.
 |
То, что список стабилизировался при пороге 0.001 и не стабилизировался при 0.005 объясняется тем, что на этот раз в одной из первых итераций была отсечена некоторая последовательность, учет которой при составлении матрицы профиля потянул за собой все новые и новые такие же "плохие" последовательности.
Скорее всего, максимальное пороговое значение, при котором итерации будут вести себя, как при пороге 0.001,
будет примерно 0.0019. Это значение обязано отсекать те же последовательности, что и порог в 0.001, а
минимальное значение E-value такой "нежелательной" последовательности 0.0020.
Итеративный поиск с последовательностью №4 (P80643, ACP_BACSU) доставил больше хлопот, так как значение порога
неоднократно приходилось уменьшать.
В конце концов при пороговом значении 0.00001 (что равносильно обозначению 1е-5) поиск сошелся на пятой итерации.
назад в проекты