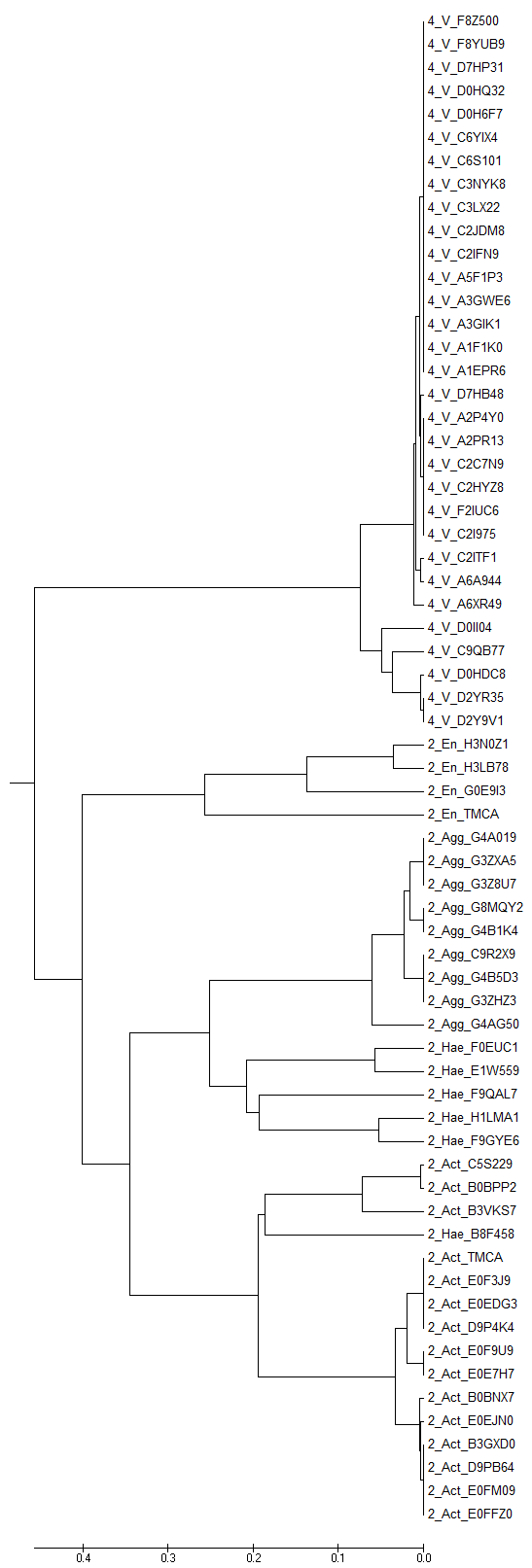
 UPGMA
UPGMA 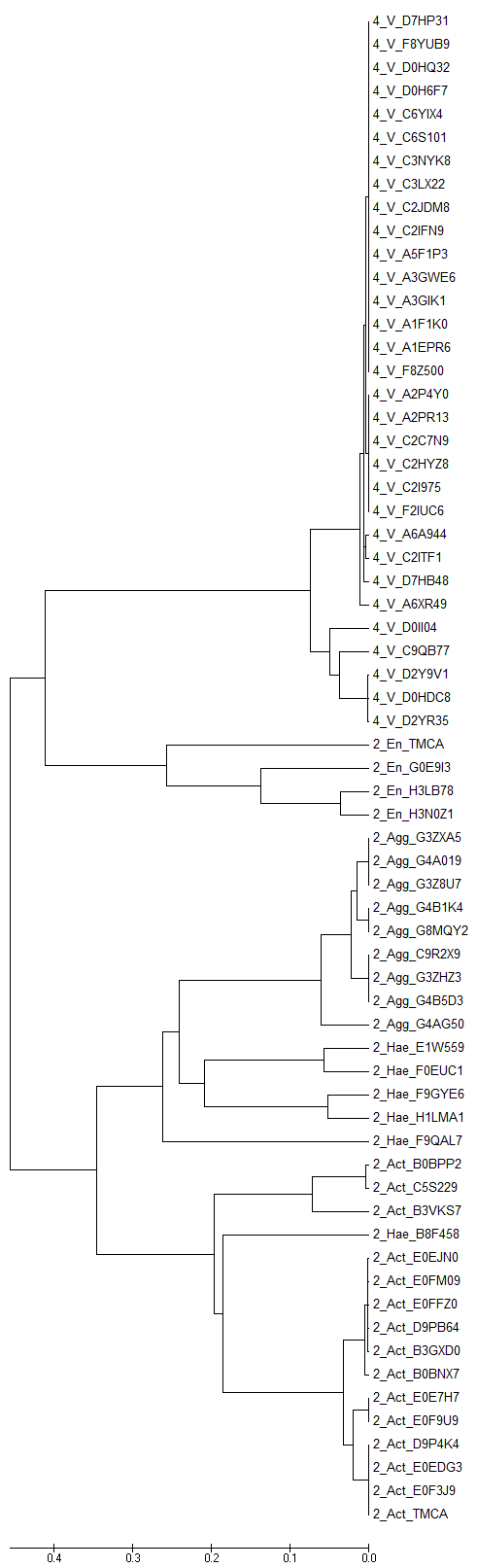 Neighbor-Joining
Neighbor-Joining 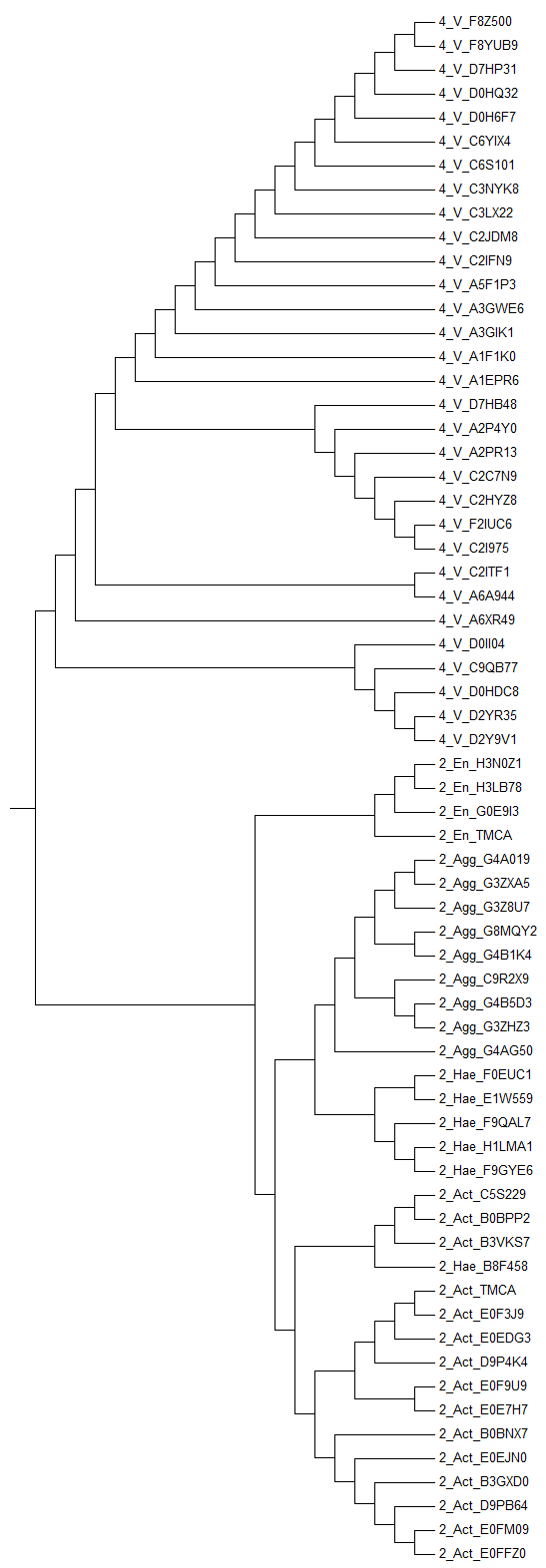 UPGMA (филогения)
UPGMA (филогения) 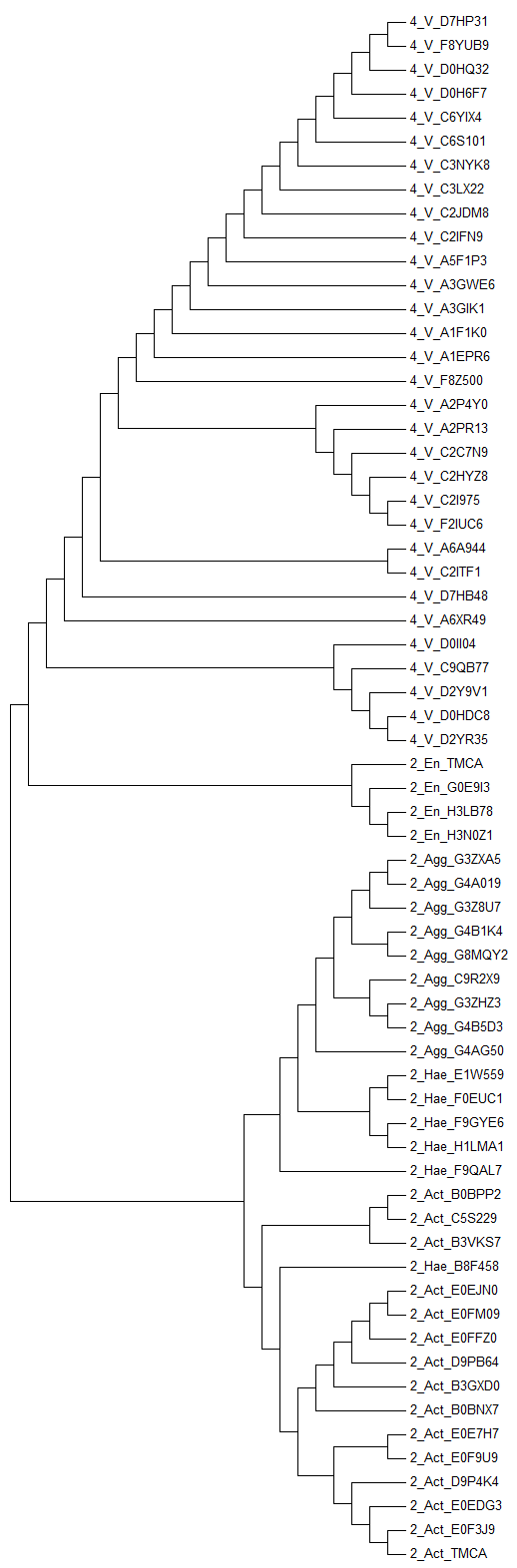 Neighbor-Joining (филогения)
Neighbor-Joining (филогения)  UPGMA UPGMA |
 Neighbor-Joining Neighbor-Joining |
 UPGMA (филогения) UPGMA (филогения) |
 Neighbor-Joining (филогения) Neighbor-Joining (филогения) |
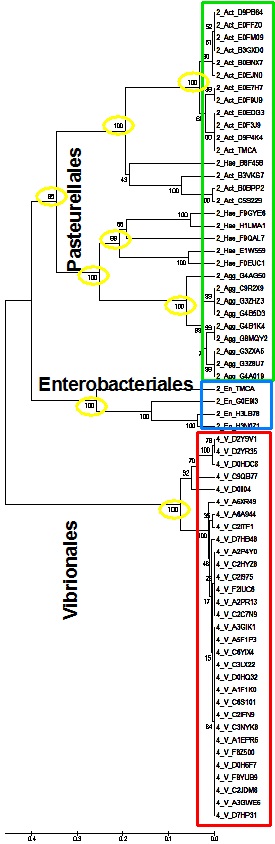 Boot-strep анализ.
Boot-strep анализ.