Профиль pftools. Занятие 11.
1. Построить филогенетическое дерево по выравниванию представителей домена, полученному в практикуме 9.
Из исходного выравнивания (такое же, как в прошлом практикуме) был получен файл .msf.
Jalview => seq_10.msf
Дальнейшую судьбу файла содержательно описывают строки ниже:
seqret seq_10.msf msf::seq_11.msf
затем, после ручной правки отступов,
noreturn -infile seq_11.msf -outfile seq_11_nrt.msf # убрали символы возврата строки
pfw seq_11_nrt.msf > seq1.msf # взвесили
pfmake seq1.msf /usr/share/pftools23/blosum62.cmp > my.prf # создали профиль
pfsearch -C 10 -f my.prf /srv/databases/uniprot/sprot_shuffled.fasta| sort -nr > scores.txt # поиск в "shuffled" SW
pfscale scores.txt my.prf > scaled.prf # нормализация
pfsearch -C 5.5 -f scaled.prf /srv/databases/uniprot/sprot.fasta| sort -nr > scores2.txt # поиск по нормализованному профилю
В результате был получен файл scores2.txt (прибл. 90мб => не даю ссылок) с находками по профилю в SwissProt с порогом 5,5.
По неизвестной причине, несмотря на нормализацию, веса находок в начале списка больше тысячи.
Вручную отсечем последовательности с порогом ниже 420 и будем проводить дальнейший анализ с оставшимися 597 последовательностями.
Сравним находки с наличием данного домена в них по аннотациям SwissProt.
Для этого используем функции Excel и данные, полученные в практикуме 9 о всех 1317 представителях семейства Helicase_RecD (PF05127).
Собственно,
файл Excel
содержит этих представителей (Лист 2), полученные последовательности и их обработку (Лист 1) и статистическую
обработку и графики (Лист 3).
После разметки найденных последовательностей, встретившихся в pfam (среди всех этих последовательностей подавляющее
большинство оказалось из TrEMBL, и лишь 30 - из SwissProt (можно проследить по ID)), была проведена статистическая обработка.
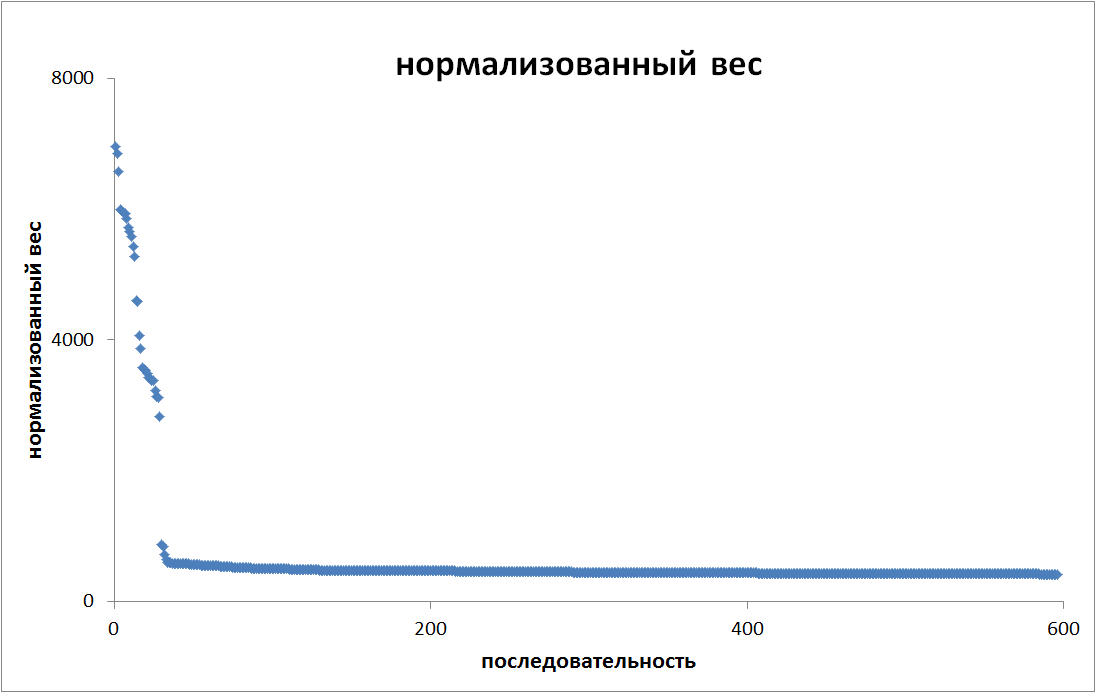
В Excel для полученного списка находок был построен график нормализованного веса находок,
«ступеньку» на котором можно интерпретировать как порог нормализованного веса для находок из семейства
(в данном случае равен 420):
Также были подсчитаны данные о специфичности и чувствительности поиска и составлена ROC-кривая.
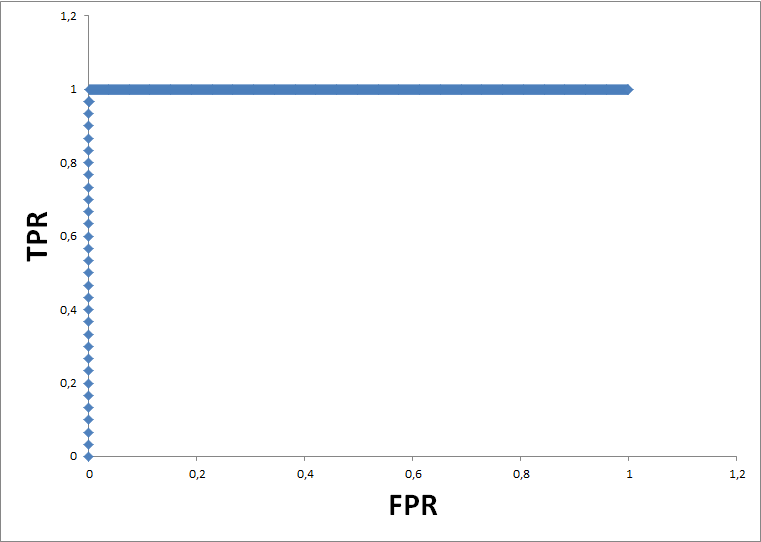
ROC-кривая представляет собой зависимость чувствительности алгоритма классификации
(т. е. true positive rate, TPR) от величины FPR (false positive rate), которую можно обозначить
как 1 – специфичность.
На основе данных, приведённых в файле cyclin_n_profiles.xlsx, можно установить
порог нормализованного веса, равный 840 (говоря строго, по полученным данным, он может варьироваться
от 718 до 849 без ограничений), который не даёт ошибок первого рода (это число
соответствует вероятности не определить последовательность, содержащую домен Cyclin_N согласно
Pfam) и 1 ошибку второго рода (0,17%; это значение соответствует вероятности определить
последовательность, не имеющую домена Helicase_RecD по Pfam, как принадлежащую семейству).
Можно сказать, что по итогам работы удалось создать профиль, позволяющий достоверно отличить заданные
группы последовательностей (в SwissProt). При этом можно хорошо "отловить" все искомые последовательности, почти
не захватив при этом лишнего (ошибки II рода). Также, как вариант для другой задачи, можно подобрать и другой
порог (см. файл), по которому можно отбросить все ложно определенные последовательности, допустив при этом
лишь одну ошибку 1 рода.
На страницу 4 семестра
© Aleshin Vasily

