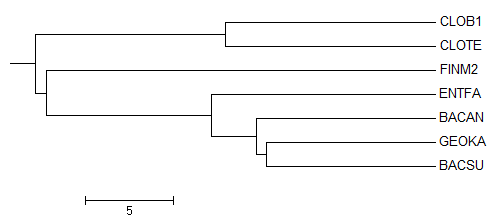
 Рис.3
Рис.3
Здесь отсутствует 1 ветвь (BACSU,BACAN vs CLOTE,CLOB1,FINM2,ENTFA,GEOKA).
Вместо нее есть ветвь (BACSU,GEOKA vs CLOTE,CLOB1,FINM2,ENTFA,BACAN).
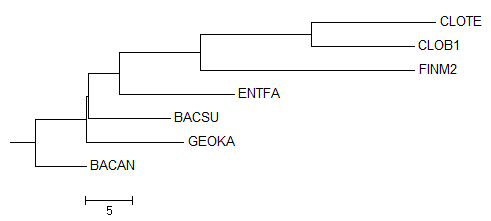 Рис.4
Рис.4
Здесь отсутствует 1 ветвь (BACSU,BACAN vs CLOTE,CLOB1,FINM2,ENTFA,GEOKA).
Вместо нее есть ветвь (BACAN,GEOKA vs CLOTE,CLOB1,FINM2,ENTFA,BACSU).
Данный метод создает неуорененные деревья.
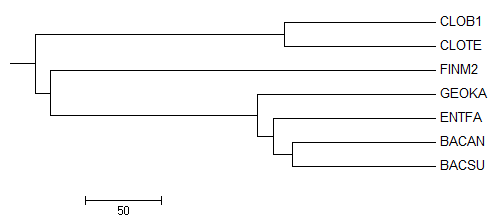 Рис.5
Рис.5
Здесь отсутствует 1 ветвь (BACSU,BACAN,GEOKA vs CLOTE,CLOB1,FINM2,ENTFA).
Вместо нее есть ветвь (BACSU,BACAN,ENTFA vs CLOTE,CLOB1,FINM2,GEOKA).
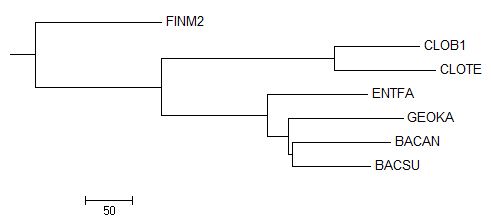 Рис.6
Рис.6
Все ветви составлены правильно, что здорово!
На картинке приведено неправильное укоренение, но данный метод дает неукорененное дерево, поэтому можно признать эту попытку полностью удачной.
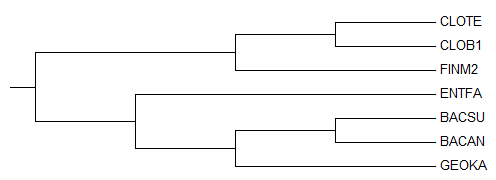 Рис.1
Рис.1
 Рис.2
Рис.2
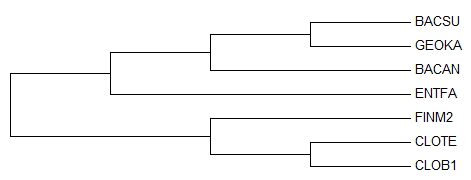 Рис.7
Рис.7