Занятие 2.
1. Восстановление кристалла.
а. Структура EHDD (Engrailed homeodomain DNA complex).
Для данного кристалла отсутствует файл с картой электронной
плотности на сервисе EDS => будем использовать построенную модель.
Покрасим в структуре 3HDD белковые цепи а и b в красный и синий соответственно, а цепи днк в оттенки желтого.
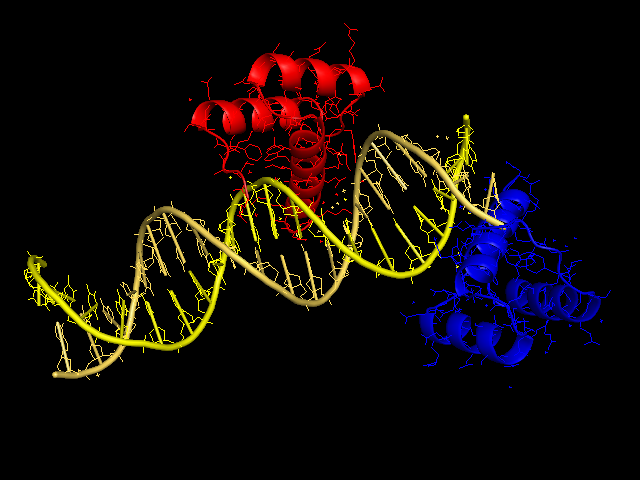
Рис. 1. Общий вид модели структуры 3HDD с раскраской по цепям.
Для объяснения направленных в пустоту остатков белка восстановим соседние с цепью b (Рис 1, синий) структуры.
Цепь b была выбрана, так как она находится на краю цепи ДНК, и наиболее доступна для связывания белками из
других ячеек.
symexp sym, 3hdd, chain b, 5
Эта команда достраивает объекты-реплики объекта 3hdd, называя их со слова "sym", находящиеся от цепи b структуры
менее чем в 5 ангстремах.
Новые структуры покрасим в серый цвет.
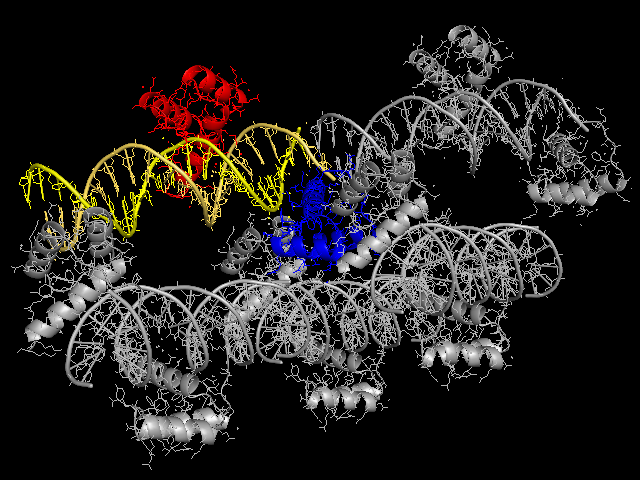
Рис. 2. Cтруктура 3HDD (цветовая схема сохранена) с добавленными соседними к цепи b репликами.
Для визуализации контактов между цепью b и ДНК из соседих ячеек сперва удалим воду:
remove solvent
затем командами
show sticks, byres (new_dna and (b_p around 3.5))
show sticks, byres (b_p and (new_dna around 3.5))
покажем в объектах "new_dna" и "b_p" (ДНК из других ячеек и исходная цепь b) остатки на расстоянии 3.5 ангстрема
между объектами.
Выделенные остатки можно выделить в отделные множества и покрасить поэлементно
(углероды белка зеленым, а ДНК - оранжевым) Рис. 3.
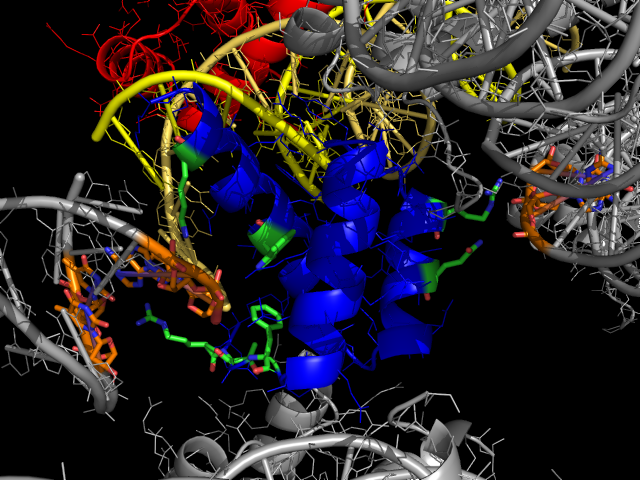
Рис. 3. Структура 3HDD, детали как в Рис. 2. Зеленым показаны атомы С остатков белка (цепь b),
контактирующие с ДНК из других ячеек (ее углероды из контактирующих остатков показаны оранжевым).
Можно заметить, что кроме водородных связей на расстоянии биже 3.5 ангстрем находятся и остатки, не образующие
водородных связей, например это сразу видно в случае фенилаланина.
Команда
distance hbonds, p_contact, dna_contact, 3.5, mode=2
Позволяет вывести такие случаи на чистую воду =) и выделить только "полярные связи" (за это отвечает mode=2).
Действительно, упомянутый выше фенилаланин, а также триптофан не образуют с ДНК водородных связей,
а просто близко расположены к ней, зато связи других остатков теперь видны явно (Рис. 4).
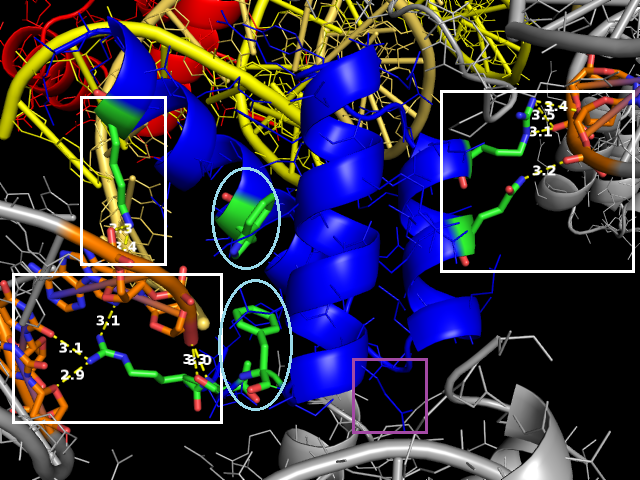
Рис. 4. Контакты между цепью b и ДНК из соседних ячеек. Водоробные связи и расстояния подписаны и выделены
трем белыми прямоугольниками (для удобства), в голубой овал заключены остатки, не образующие водородных
связей, но расположенные на расстоянии менее 3.5 ангстрем от ДНК. В фиолетовый прямоугольник заключен
остаток аргинина, расположенный на расстоянии более 3.5 ангстрем, но судя по структуре, все же взаимодейтвующий с
уже третьей достроенной цепью ДНК (для построения брали дистанцию 5 ангстрем).
Наконец, уберем все достроенные модели, которые не контактируют ДНК с исходной цепью b
(на основании предыдущих расчетов), и покажем контактирующие остатки в виде сфер (Рис. 5).
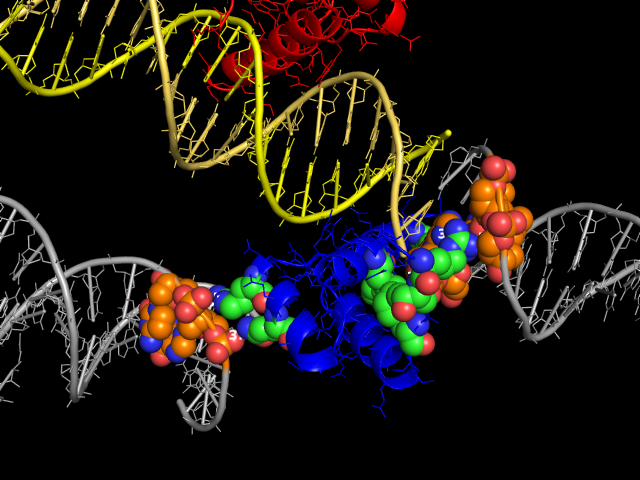
Рис. 5. Контакты между цепью b и ДНК из соседних ячеек в сферической модели.
Итак, наглядно показано, что в структуре 3HDD белок и ДНК образуют прочно взаимодействующие
контакты как внутри одной ячейки, так и между ячейками.
Можно было заметить, что в основном контакты образованы остатками лизина и аргинина, но также
встречаются остатки других гидрофильных аминокислот, например глутамин. Это элементарно объясняется большим
отрицательным зарядом ДНК, следовательно ДНК-связывающие белки должны быть положительно заряжены, а также обычно
они имеют специальные домены для укладки спирали в большую бороздку ДНК.
В данном белке это типичный гомеодомен.
b. Описание контактов молекул в структуре 1VI9.
По записи ниже можно получить информацию о кристалле белка:
CRYST1 53.383 92.592 111.237 90.00 99.42 90.00 P 1 21 1 8
Данные были внесены в таблицу.
Опишем контакты между молекулами этого белка.
Сперва достроим другие молекулы белка, расположенные вокруг выбранной структуры (в файле лежит модель для
двух структур, каждая по две цепи белка).
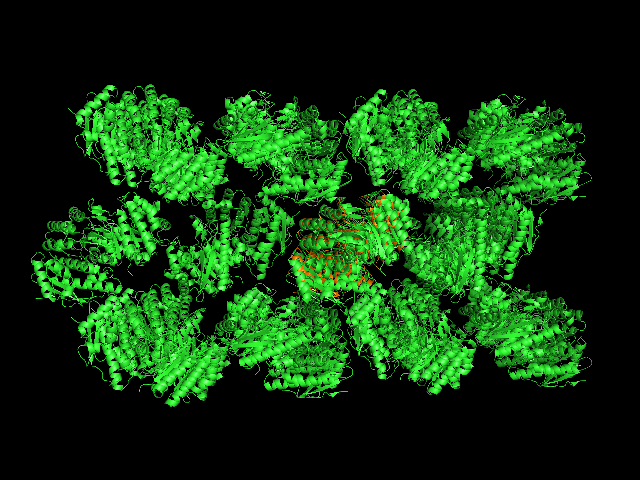
- Рис. 6. Модель белка 1vi9, в кристалле.
Белки показаны в палочной модели и занимают более одного объема элементарной ячейки.
- Рис. 7. То же с учетом вторичых структур.
- Рис. 8. То же в виде скелета.
Затем уберем молекулы растворителя, чтобы они не мешали искать контакты между непосредственно атомами белка.
Далее переименуем для удобства все контактирующие в кристалле димеры белка.
Всего получилось 8 контакирующих структур, включая и изначально имевшуюся в файле вторую модель
(результат переподгонки структуры авторами).
Все эти действия уже описаны выше, потому избавлю читателя от избыточного копирования информации.
А вот команду для создания поверхностей стоит записать:
surface, p1 within 3.5 of str2
distance hbonds, p1, str*, 3.5, mode=2
Первая строчка похволяет показать поверхность атомов из p1,
находящихся на рассоянии менее 3.5 ангстрем от структуры str2.
Вторая строка позволяет сразу задать все водородные связи (см. выше) для всех структур,
название которых начинается с str (str*).
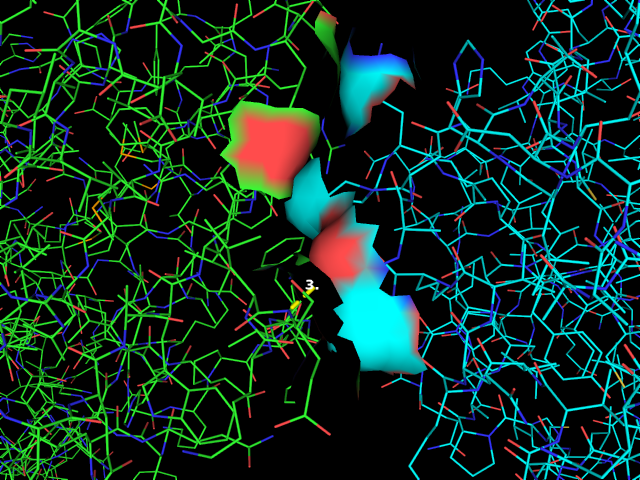
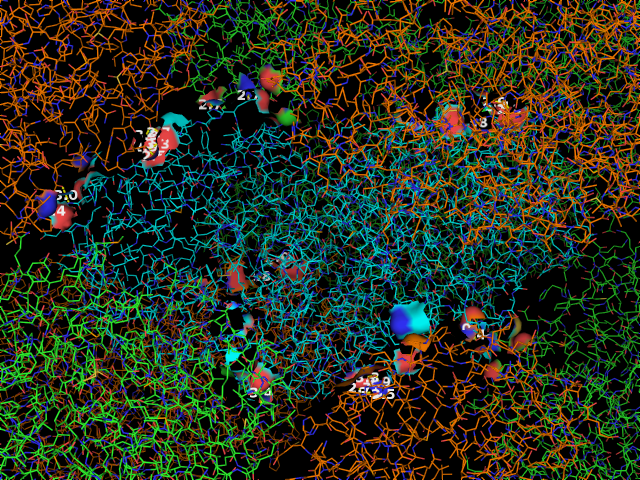
Рис. 9. Поверхности контакта и водородные связи на границе взаимодействия молекул белка в кристалле.
Такой же подход можно применить и к поверхностям, чтобы не рисовать их попарно.
В результате получаем картинку, где показаны все контакты (на обоих взаимодействующих структурах - Рис.10;
и только в одной целевой структуре "р1" -Рис.11).
- Рис. 10. Поверхности контакта и водородные связи на границе взаимодействия молекул белка в кристалле.
Структуры показаны разным цветом в виде линий.
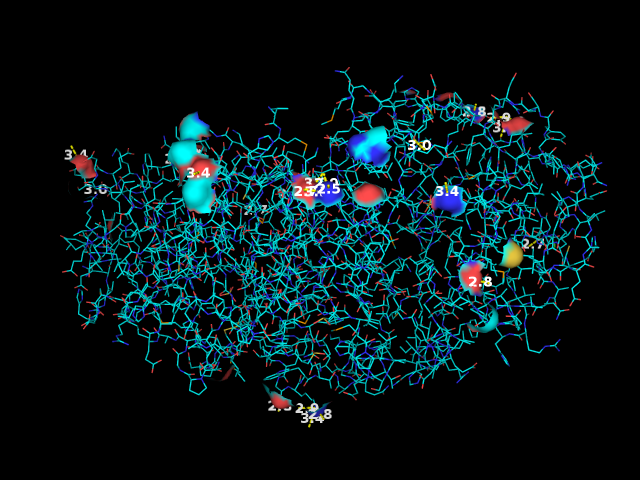
- Рис. 11. Поверхности контакта и водородные связи на границе взаимодействия лишь одной молекулы
белка в кристалле.
Отмеченные водородные связи, достаточно большая площадь поверхности взаимодействия (растояние 3.5 ангстрема),
большое количество самих контактов, и все это без учета взаимодействий через молекулы воды, которых,
судя по прошлому опыту работы с этой структурой, в районе контакта весьма много!
Итак, все это говорит о достаточно высокой степени взаимодействия молекул белка в кристалле, что неизбежно
влияет на полученные структуры, и можно с большой уверенностью предоложить, что структура белка в растворе,
направление и расположение а.к. остатков в растворе отличается от полученной модели.
Однако считается (и нет достаточных оснований для сомнений), что полученная модель отражает свойства белка
в достаточной степени.
На страницу 7 семестра
© Aleshin Vasily