ЯМР и Биологические единицы.
1. Запись PDB, полученная методом ЯМР.
В рамках данного задания необходимо было выбрать файл:
- полученный методом ЯМР
- с числом моделей более 2
- сделанный в 2014 году
- с длиной цепи более 60
- (плюс по заданию: в котором Arg взаимодействует с Asp или Glu)
Избежать ошибки и лишней траты времени помогает специализированный поиск по PDB, в котором для каждого из
условий можно было задать отдельное условие и посчитать, сколько структур ему удовлетворяет.
Например, файлов ЯМР на момент поиска насчиталось чуть более 10000, то есть 10% от вего PDB.
В результате поиска я остановился на структуре 2mh7, ферредоксина с 2Fe-2S кластером и длиной цепи 101 а.к.
В PyMol я пощелкал разные модели (всего их 15), запустил "видео", удивился, насколько высокая подвижность у
этого белка.
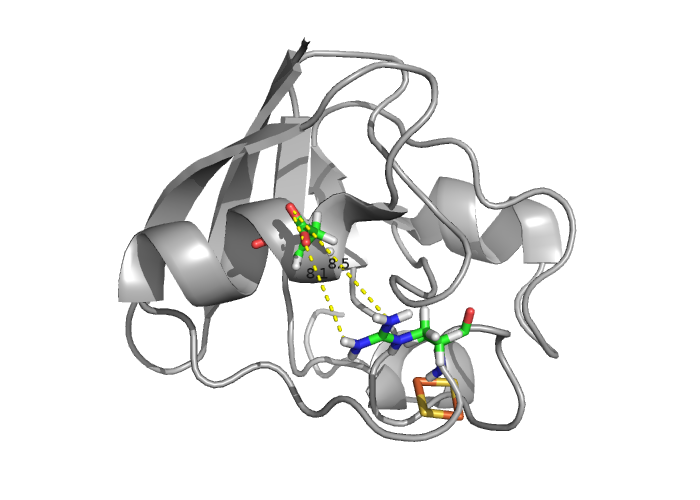
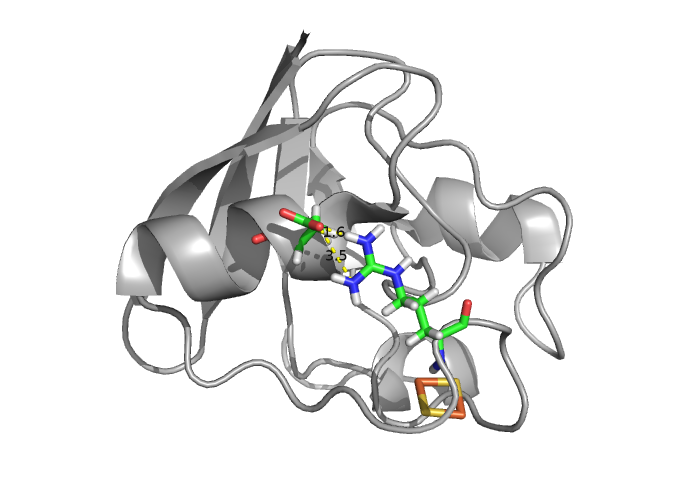
После этого я нашел единственный Arg в белке, и к счастью, пару к нему из Asp (См. Рис. 1 и 2).
- Рис. 1 и 2. Контакт между остатками Arg и Asp в структуре 2mh7. Расстояния указаны в Ангстремах.
Видно, что на рисунке 2 водородная связь присутствует в явном виде (1.6 Ангстрем), в то время как на рисунке 1 этой связи нет (более 8 Ангстрем).
При этом внешне не видно больших различий структуры, которые были видны при быстрой прокрутке моделей, но
то, что водородная связь в части моделей присутствует, а в части отсутствует, и расстояние
между остатками то 2, то 8 Ангстрем (в зависимости от модели), модет свидетельствовать о большей подвижности
молекулы при восстановлении ее структуры методом ЯМР в растворе по сравнению с РСА (вывод довольно очевидный).
В то же время нужно остородно относиться к подвижным участкам в моделях ЯМР, так как это лишь модель.
2. Биологические единицы.
В рамках данного задания необходимо было выбрать структуру, у которой:
- асимметрическая единица включает 2 цепи
- биологическая единица состоит из 4 цепей
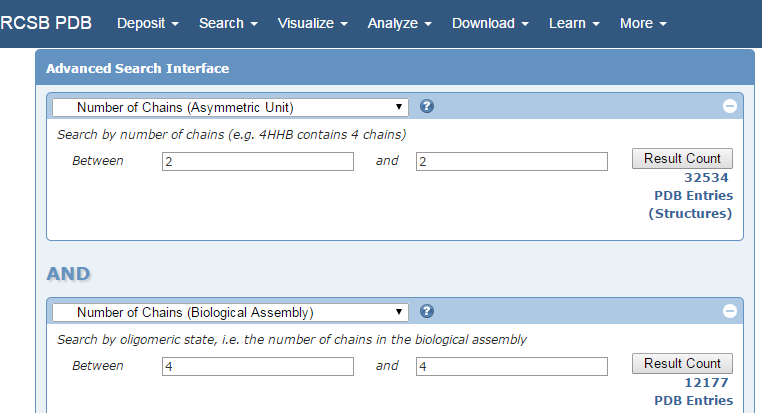
- Рис. 3. Поиск в PDB по критериям задания 2.
В результате несложного запроса (скриншот на Рис. 3) было найдено несколько тысяч структур, и выбрана
пируваткиназа (структура 4ks0 2014 года), так как сам фермент мне знаком и интересен.
В норме это тетрамер,
а в асимметрической ячейке лежал димер, что было исправлено командами
split_states 4ks0
distance hbonds, 4ks0_0001, 4ks0_0002, 2.7, mode=2
первая из которых переводила два "состояния" в разные объекты, водородные сязи между которыми и искала
вторая команда.
Далее была выбрана одна из связей, удобная для визуализации. Так совпало, что это была не связь N...O, а связь O...O,
между серином и аспартатом.
Расстояние между атомами кислорода 2.5 Ангстрем (Рис. 4).
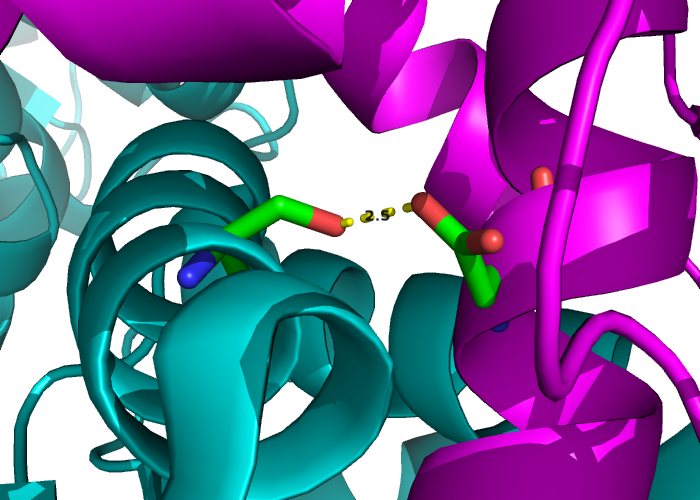
- Рис. 4. Одна из водородных связей между белковыми цепями тетрамера пируваткиназы, лежавшими в разных "состояниях" (показаны цветом) биологической единицы структуры 4ks0.
Стоит подчеркнуть, что общее колиество водородных связей между субъединицами было очень большим,
значительно больше чем в аналогичном задании по поиску водородых связей в местах контакта белков в кристалле.
Представленная на рисунке водородная связь лишь одна из многих.
На страницу 7 семестра
© Aleshin Vasily