Умения. d4. Построение и раскраска поверхностей.
7. Построение поверхностей.
Для комплекса пуринового репрессора 1bdh была получена биологическая единица.
В рамках данного задания все задачи включали использование множеств из белка, ДНК, мономеров белка, которые без труда выбираются по цепям.
В дальнейшем заданые множества prot, p1, p2 (белковые цепи) и dna использовались для определения
множеств контактирующих остатков командами
sel contacts1, (p1 and (p2 around 5))
sel contacts2, (p2 and (p1 around 5))
sel contacts3, (prot and (dna around 5))
sel contacts4, (dna and (prot around 5))
По Далее несложно было получить требуемые изображения (Рис. 1-4).
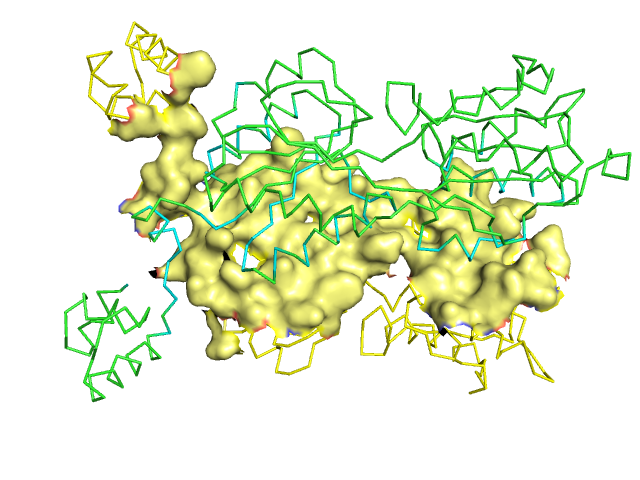
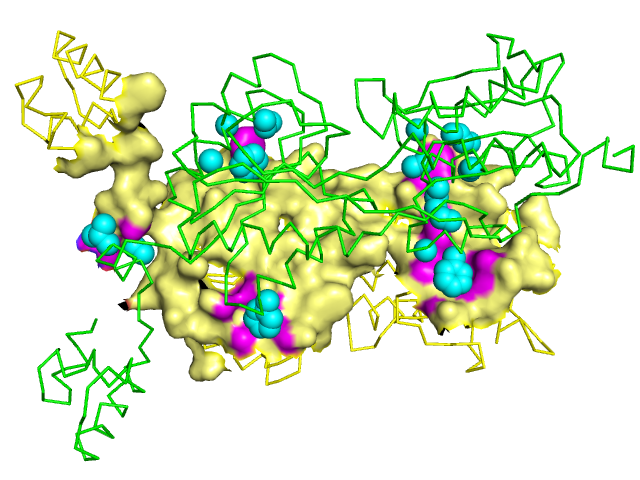
- Рисунок 1. Изображение поверхности контата мономера белка с симметричным мономером на фоне остовной модели белка. Циановым показаны остатки, относящиеся к поверхности контакта со стороны второго мономера.
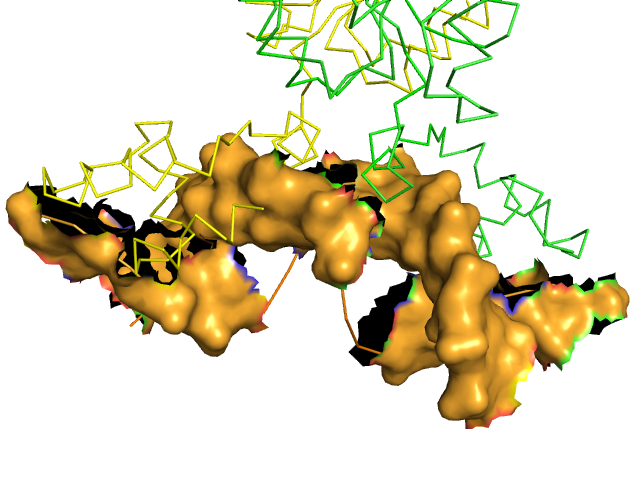
- Рисунок 2. Изображения поверхности контакта димера белков с ДНК на фоне остовной модели остатков белка, вовлеченных во взаимодействие.
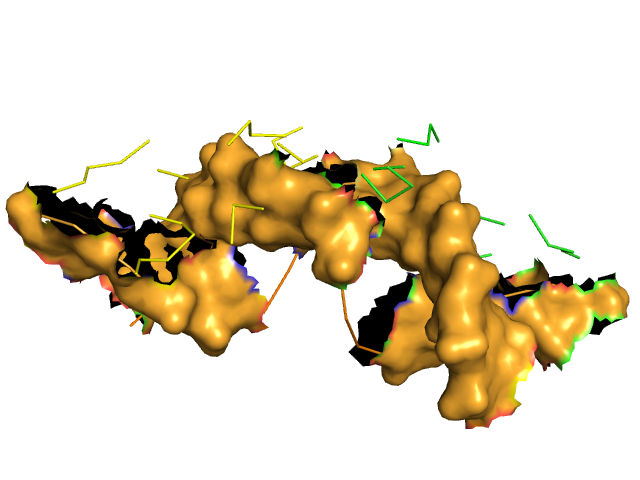
- Рисунок 3. Изображения поверхности контакта димера белков с ДНК на фоне остовной модели остатков димера белка.
На рисунке 2 и 3 видно, насколько плотно взамодействуют остатки белка с ДНК. Некоторые цепи прямо, как ключ к замку соответствуют
поверхности ДНК. Удивительно!
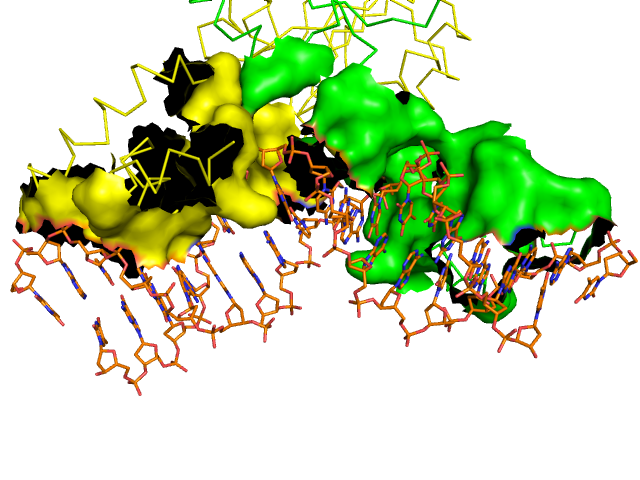
- Рисунок 4. Изображение поверхности контата ДНК с димером белков на фон проволочной (stick_radius 0.17) модели ДНК.
На данном рисунке наглядно показано, насколько важно учитывать димеризацию белка (биологичекую единицу).
8. Изображение поверхности и раскраска ее участка, соответствующего гидрофобному кластеру CluD.
Сперва необходимо было как-то подать CluD димер белка, с чем я справился, создав собственый файл белка в формате pdb,
который CluD смог принять и обработать.
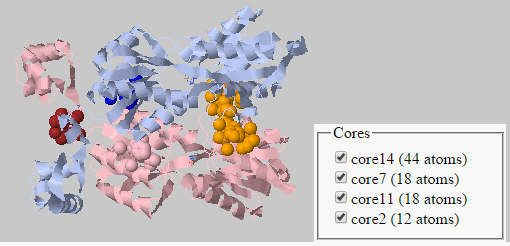
Было получено 4 кластера с оговоренными параметрами: расстояние 5.0, минимум 10 атомов в кластере (Рис. 5).
- Рисунок 5. Изображение межсубъединичных кластеров CluD.
Далее необходимо было как-то получить сами атомы, их однозначное определение. Для этой цели я сперва попробовал использовать Jmol, и весьма продвинулся, как я считаю, для первого раза.
Однако на этапе раскраски поверхностей, относящихся к разным множествам я застопорился, и решил, что легче будет вытащить данные из
текстовой выдачи CluD, с чем я успешно справился, получив команду для PyMol
sel contp2_2, p2 and (////50/CG2 or ////50/CB or ////54/CG or ////54/CD1 or ////54/CB or ////54/CD2 or ////107/CE2 or ////110/SD
or ////107/CD1 or ////107/CZ or ////107/CE1 or ////107/CD2 or ////107/CG or ////106/CB or ////103/CG or ////103/CD or ////103/CB
or ////86/CE2 or ////86/CE1 or ////86/CD1 or ////86/CZ or ////86/CD2 or ////86/CG or ////86/CB or ////282/CE2 or ////282/CZ
or ////282/CD1 or ////282/CE1 or ////282/CD2 or ////282/CG or ////282/CB or ////283/CD1 or ////252/CE or ////283/CE1 or ////252/SD
or ////252/CG or ////223/CB or ////224/CB or ////227/CD1 or ////256/SG or ////227/CE1 or ////256/CB or ////223/CG or ////227/CG
or ////227/CB or ////279/CB)
(немного громоздко, но автоматически).
А вот сам исходный a href="files/prot.txt">выходной файл CluD в котором я оставил лишь нужные нам остатки.
В результате получилось изображение Рис 6.
- Рисунок 6. Полученное изображение, аналогичное рисунку 1, но при этом розовым показаны участки поверхости, соответствующие гидрофобным кластерам CluD, а голубым показаны атомы другого мономера белка, к этим гидрофобным кластерам.
Гидрофобные контакты соответствуют рисунку 5, как мы видим.
txt-файл от PDBeFold
На страницу 7 семестра
© Aleshin Vasily