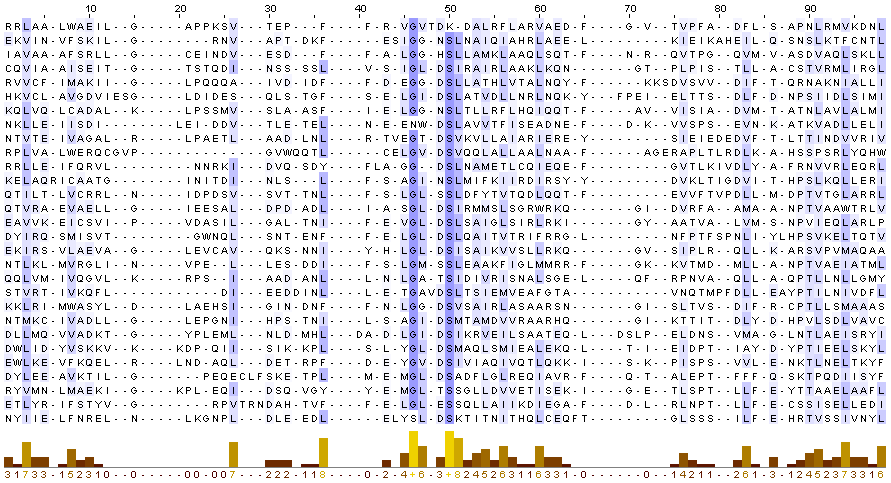
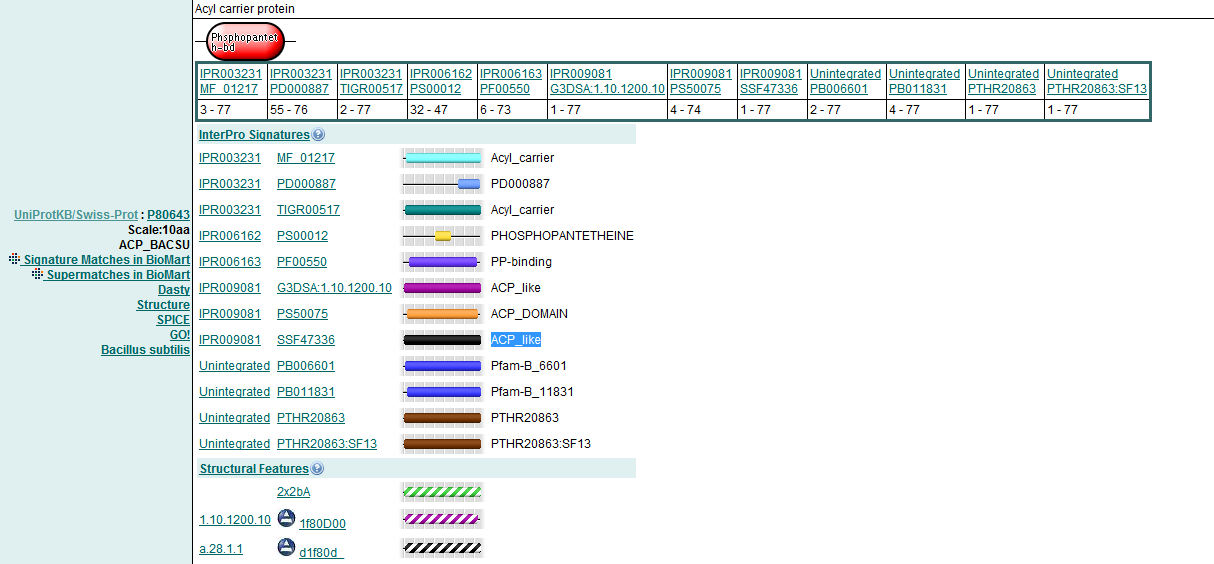
Доменная структура белка ACP_BACSU по данным Pfam

4'-фосфопантеиновая связующая ("протезная") группа прикрепляется через а.о. серина.
Она действует как "качающийся рычаг", помогая связыванию активированных жирных кислот с аминокислотными группами.
Этот домен образует пучок из четырех спиралей.
Cемейство включает в себя белки, не входящие в Prosite, на основании анализа последовательностей и функциональных особенностях.
В родственном домене P19828 связующий серин замещен на аланин.