 |
 |
 |
 |
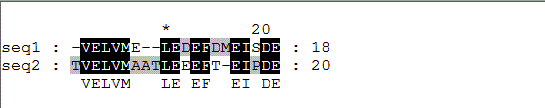
| белок (ID) | ACP_BACSU | ACP_BREBN |
| организм | Bacillus subtilis (Сенная палочка) | Brevibacillus brevis |
| идентичность,% | 81 | 81 |
| сходство,% | 91 | 91 |
| число ГЭПов (символов разрыва) | 0 | 0 |
| Число идущих подряд гэпов | 0 | 0 |
| Суммарное число гэповых колонок | 0 | 0 |
| Координаты выровненных участков | последовательность выровнена целиком, с 1 по 77 остатки | последовательность выровнена целиком, с 1 по 77 остатки |
 |
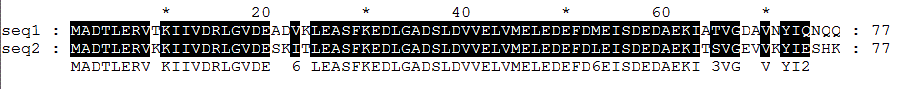 |
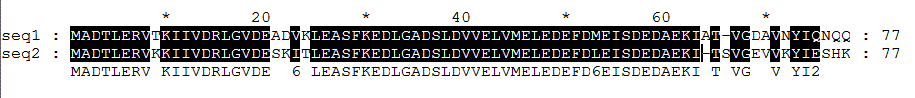 |