Опиcание доменной архитектуры белка YOAJ_BACSU в соответствии с банком Pfam.
С помощью банка Pfam была получена схема доменной архитектуры белка YOAJ_BACSU. Она показана на рисунке 1. Пояснения к схеме представлены в таблице 1.

Рис.1. Схема доменной архитектуры белка YOAJ_BACSU. Зеленым цветом показан домен DPBB_1.
Таблица 1. Пояснения к схеме доменой архитектуры белка YOAJ_BACSU.
| № | Pfam AC | Pfam ID | Полнное название семейства доменов | Положение в последовательности белка | Клан |
| 1 | PF03330 | DPBB_1 | Дубль-psi beta-бочонок, характерный для белков Rare lipoprotein A. У разных белков может выполнять разные функции, может выступать в роли ферментативного домена. | 56 - 127 | Клан DPBB (CL0199) содержит 5 достаточно разнообразных семейств доменов, выполняющих в основном каталитические функции. |
Кроме домена DPBB_1 белок YOAJ_BACSU содержит сигнальный пептид (sig_p, на схеме оранжевый) в положении 1 - 25 и участок низкой сложности в положении 33 - 46 (low_complexity, на схеме голубой).
Информация о домене DPBB_1
Домен DPBB_1 входит в 43 различные архитектуры. Известна последовательность 5162 белков и определена пространственная структура трех различных белков, содержащих данный домен.
Для фрагментов белков, соответствующих домену DPBB_1, было получено множественное выравнивание в формате msf. Файл с выравниванием можно получить пройдя по ссылке. На рисунке 2 представлено изображение выравнивания.

Рис.2. Множественное выравнивание фрагментов белков, соответствующих домену DPBB_1.
На изображении выравнивания видно, что некоторые консервативные участки сохраняются по всей его длине. Исходя из этого, можно сделать вывод, что выравнивание достаточно достоверно.
Анализ встречаемости доменов PF00759 и PF03330 в организмах разных таксонов
Для анализа встречаемости доменов в организмах взята архитектура E4XG49_OIKDI, содержащая домены PF00759 (Glyco_hydro_9) и PF03330 (DPBB_1). Её изображение показано на рисунке 3. Домен PF00759 (Glyco_hydro_9) характерен для семейства гликозил-гидролаз 9. Это широко распространенная группа ферментов, гидролизующих гликозидные связи в молекулах углеводов. Для обоих доменов были получены таксономические деревья с указанием числа доменов для каждого таксона. Результаты анализа представлены в таблице 2.

Рис.3. Схема архитектуры E4XG49_OIKDI. Красным цветом показан домен PF03330, а зеленым PF00759.
Таблица 2. Представленность доменов PF00759 и PF03330 в организмах разных таксонов.
| Таксон | Количество белков с доменом PF00759 | Количество белков с доменом PF03330 | |
| Эукариоты | Зеленые растения | 697 | 1623 |
| Грибы | 28 | 317 | |
| Животные | 229 | 127 | |
| Остальные эукариоты | 35 | 75 | |
| Археи | 3 | 0 | |
| Бактерии | 836 | 3011 | |
| Вирусы | 0 | 3 | |
В результате анализа выяснилось, что оба домена наиболее широко представлены среди зеленых растений и бактерий. Такое распределение встречаемости доменов согласуется с их функциями, так как для мноих белков, содержащих эти домены, характерна эндоглюканазная активность, а способностью расщеплять полисахариды в основном обладают растения и бактерии.
Сравние описания мотивов в разных банках семейств, по данным InterPro для белка YOAJ_BACSU
InterPro - это база данных, в которой хранится информация о различных семействах белков, доменах, сайтах и мотивах. Она объеиняет различные базы данных, такие как SUPERFAMILY, CATH-Gene3D, PRINTS, PANTHER, Pfam. В результате выполнения работы было получено изображение со сравнением особенностей последовательности YOAJ_BACSU, из баз данных SUPERFAMILY, CATH-Gene3D и Pfam. Его можно увидеть на рисунке 4.
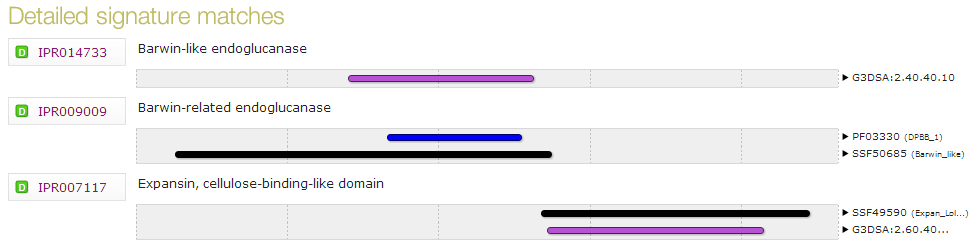
Рис.4. Изображение доменов последовательности YOAJ_BACSU из различных баз данных. Домены SUPERFAMILY показаны коричневым, Gene3D - розовым, Pfam - синим.
Самый короткий мотив называется PF03330 (DPBB_1), он описан в банке Pfam. SSF50685 (Barwin_like) - самый длинный мотив он описан в банке SUPERFAMILY. Оба они относятся к группе доменов Barwin-related endoglucanase (IPR009009). В InterPro приведены две структурные подписи для белка YOAJ_BACSU. Одна из них из банка PDB с достаточной достоверностью описывает его структруру, её положение 26 - 232. Другая из банка MODBASE имеет предположительный харатер, она начинается с 17 и заканчивается 211 аминокислотным остатком. Эти подписи не интегрированы в InterPro (для них не указан InterPro ID). Домен, описанный в Pfam, имеет положение 56 - 127, он находится в гранцах описанных выше структурных подписей.