Последовательность белка - дело случая?
Чтобы показать, что последовательности белков не возникают исключительно случайным путем, можно определить частоту встречаемости какого-нибудь слова в последовательностях. Это слово должно быть составлено из букв, совпадающих с буквами, которые обозначают аминокислоты. Затем эту частоту нужно сравнить с реальной частотой встречаемости такого слова в последовательностях белков с помощью банка Prosite .
Для того, чтобы теоретически определить количество слов "align", встречающихся в банке SwissProt, нужно знать частоты встречаемости нужных аминокислотных остатков (A = 8.25%, L = 9.66%, I = 5.96%, G = 7.07%, N = 4.06%) и их общее количество в банке (191670831). Далее частоты нужно перемножить между собой. Получится частота встречаемости слова, которая умножается на общее количество букв в банке.
8.25*9.66*5.96*7.07*4.06*10(-10)*191670831 = 261
Поиск в Prosite показал, что слово "align" встречается в SwissProt 129 раз, и 58 из них определил как случайные. Различие теоретической и реальной величины говорит о том, что количество и порядок аминокислот в различных белках не всегда являются делом случая. Для слова "align" реальное количество меньше, чем теоретическое, из чего можно сделать вывод, что это не часто встречающееся слово, и доля частот встречаемости его букв приходится на более распространенные слова, содержащие эти же буквы.
Поиск вероятных гомологов белка YOAJ_BACSU в банке SwissProt с помощью паттернов
Паттерн можно описать как запись участка последовательности с различными степенями консервативности общую для какого-либо множественного выравнивания. С помощью составления паттернов можно искать гомологичные белки и посредством регуляции "строгости" паттерна получать выборки с той или иной степенью гомологии. На основе множественного выравнивания, изображение которого представлено на рисунке 1, были составлены три паттерна различной силы для выполнения поиска. Результаты поиска представлены в таблице 1.
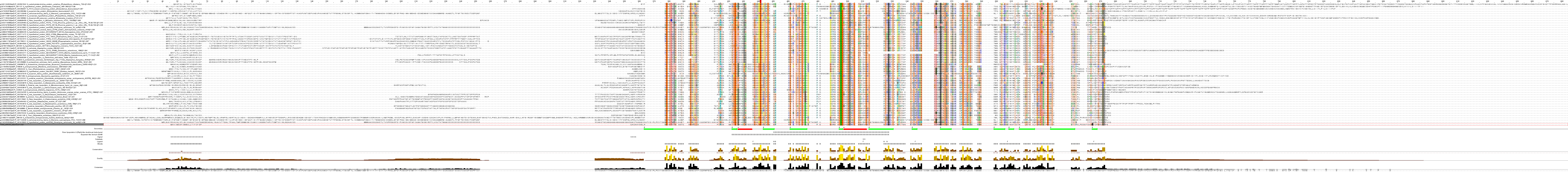
Рис.1. Множественное выравнивание эукариотических и прокариотических гомологов белка YOAJ_BACSU.
Таблица 1. Результаты поиска вероятных гомологов белка YOAJ_BACSU в банке SwissProt с помощью паттернов.
| Характеристика паттерна | Паттерн | В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? | Количество последовательностей из выравнивания |
| Сильный | [ILVF]-D-L-[FYSNAH]-X(2)-A-F-X(2)-[ILV]-X(0,2)-[AGDENT]-X(4)-[GS]-X-[IVLFTY] | 14 | 1 |
| Средний | X-D-L-X(3)-A-F-X(2)-[ILV]-X(0,2)-[GSADE]-X(4)-G-X-[ILV] | 35 | 1 |
| Слабый | [PG]-X(1,8)-[TGACIVLS]-[AG]-[ILMV]-[NGSA]-X(3)-[WYFL]-X(2,6)-[AGS]-X(2)-[ACS]-G | 2052 | 1 |
Как и следовало ожидать, с "ослаблением" паттерна растет число находок. Однако, во всех трех случаях была найдена всего одна последовательность из выравнивания - последовательность исследуемого белка YOAJ_BACSU. Такой результат нетрудно объяснить. Выравнивание проводилось для последовательностей из банка Refseq, который содержит белки из разных источников, соответственно в нем их намного больше, чем в SwissProt, для которого производился поиск. Более того, SwissProt содержит аннотированные последовательности, и чаще это будут белки из значимых для человека организмов. Например, как в полученном мной результате, из дикого лимона, риса, резуховидки Таля. Белки этих организмов могли не попасть в выравнивание из-за слишком низкого сходства в других участках последовательности.
Поиск мотивов PROSITE в последовательности белка YOAJ_BACSU
Мотивы - это определенные высоко консервативные участки последовательности белка, имеющие какую-либо функцию. В банке Prosite содержится информация о различных мотивах, и можно организовать поиск по банку, чтобы обнаружить мотивы в интересующем нас белке.
Результаты поиска представлены в таблице 2. Следует отметить, что специфичных мотивов для белка обнаружено не было.
Таблица 2. Результаты поиска мотивов в последовательности белка YOAJ_BACSU.
| Идентификатор документа Prosite (AC) | Название мотива | Краткое описание мотива | Тип подписи | Паттерн | Специфична ли подпись? | Сколько мотивов нашлось в белке? |
| PS00008 | MYRISTYL | Сайт N-миристоилирования | Паттерн | G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} | неспецифична | 5 |
| PS00006 | CK2_PHOSPHO_SITE | Сайт фосфорилирования казеинкиназы II | Паттерн | [ST]-x(2)-[DE] | неспецифична | 2 |
| PS00001 | ASN_GLYCOSYLATION | Сайт N-гликозилирования | Паттерн | N-{P}-[ST]-{P} | неспецифична | 1 |
| PS00005 | PKC_PHOSPHO_SITE | Сайт фосфорилирования протеинкиназы С | Паттерн | [ST]-x-[RK] | неспецифична | 4 |