Поиск ДНК-белковых контактов в структуре 1HDD
С помощью команды define в Jmol были заданы следующие множества атомов:
- атомы кислорода 2'-дезоксирибозы (set1);
- атомы кислорода в остатке фосфорной кислоты (set2);
- атомы азота в азотистых основаниях (set3).
Пройдя по ссылкам, можно загрузить
скрипт-файл с определениями этих множеств и
скрипт-файл вызов которого в JMol даст последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3.
Изображение структуры 1HDD показано на рисунке 1, изображение молекулы ДНК из структуры 1HDD в проволочной модели показано на рисунке 2, а на рисунках 3, 4 и 5 выделены множества атомов, определенные ранее.
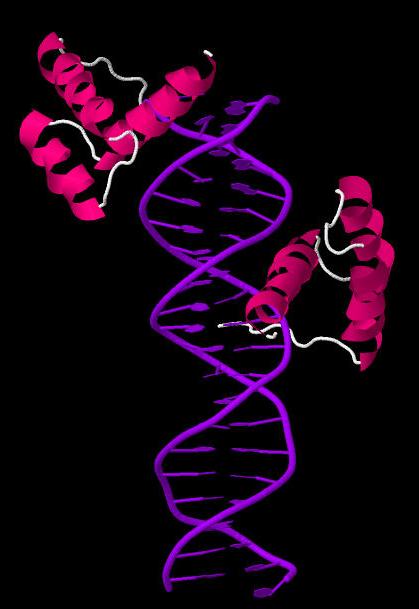
Рис.1. Структура 1HDD. |
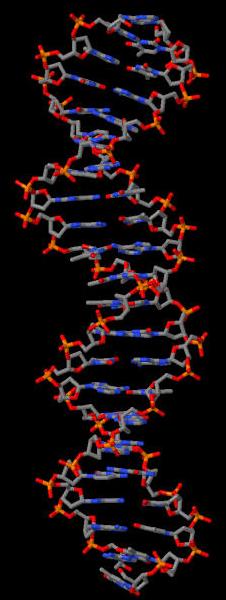
Рис.2. Молекула ДНК из структуры 1HDD в проволочной модели. |

Рис.3. ДНК из структуры 1HDD. Выделено множество set1. |
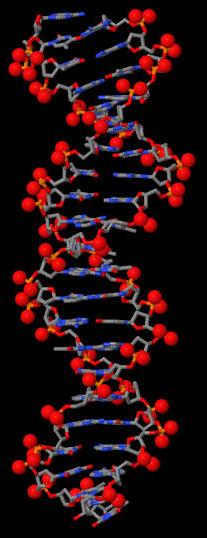
Рис.4. ДНК из структуры 1HDD. Выделено множество set2. |
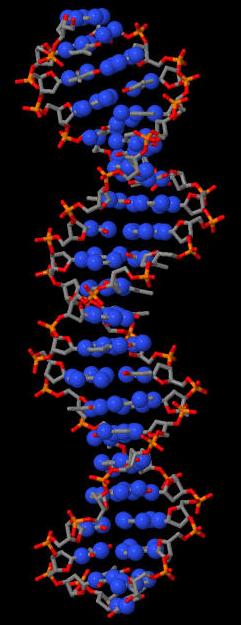
Рис.5. ДНК из структуры 1HDD. Выделено множество set3. |
Для описания ДНК-белковых контактов были заданы следующие множества атомов:
- атомы кислорода 2'-дезоксирибозы (set1p);
- атомы углерода 2'-дезоксирибозы (set1n);
- атомы кислорода в остатке фосфорной кислоты (set2p);
- атомы фосфора в остатке фосфорной кислоты (set2n);
- полярные атомы в азотистых основаниях, направленные в сторону большой бороздки (set4p);
- неполярные атомы в азотистых основаниях, направленные в сторону большой бороздки (set4n);
- полярные атомы в азотистых основаниях, направленные в сторону малой бороздки (set5p);
- неполярные атомы в азотистых основаниях, направленные в сторону малой бороздки (set5n);
- полярные атомы в белке (set6p);
- атомы углерода в белке (set6n).
Полярными будем считать атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы.
Полярным контактом будем считать ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5А. Неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5А. В результате выполнения скрипта была создана таблица 1.
Таблица 1. Количество контактов ДНК и белка.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 4 | 13 | 17 |
| остатками фосфорной кислоты | 7 | 0 | 7 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 8 | 10 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 1 | 2 |
С помощью программы nuclplot было получено изображение контактов ДНК с белком (рис.6,7,8).
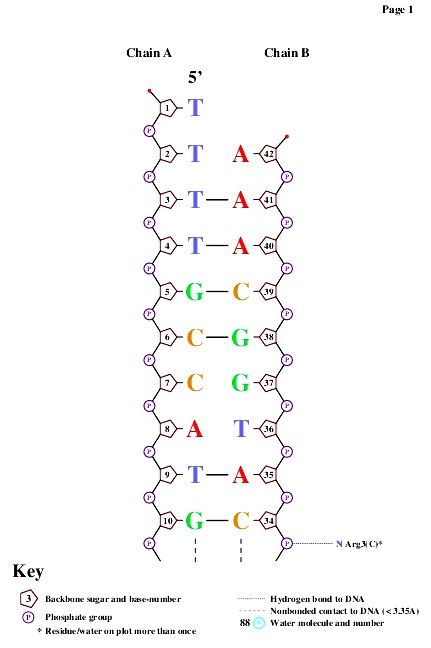
Рис.6. Связи ДНК с белком в структуре 1HDD.
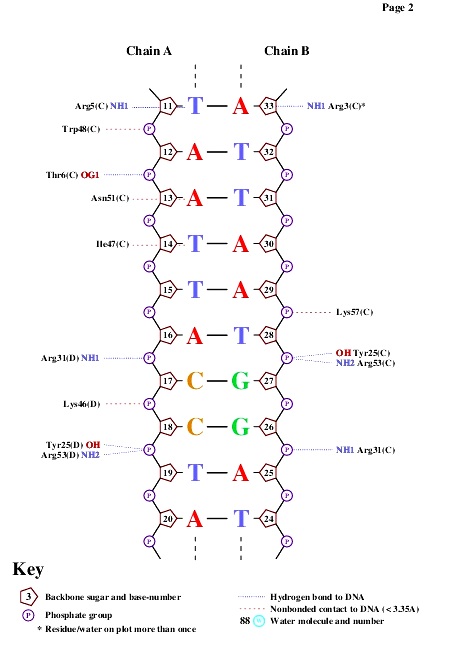
Рис.7. Связи ДНК с белком в структуре 1HDD.
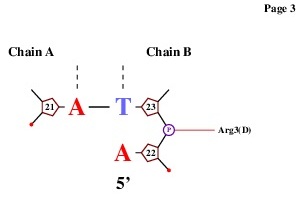
Рис.8. Связи ДНК с белком в структуре 1HDD.
На изображениях видно, что каждый аминокислотный остаток образует только одну связь. Для распознавания последовательности ДНК по моему мнению наиболее важны Asn51, Ile 47 и Arg5, так как только они образуют связи с азотистыми основаниями. В описании структуры 1HDD в PBD сказано, что Arg3 и Arg5 формируют сайт связывания автономно реплицирующейся последовательности (ARS), а точнее ARS-кора (последовательность из 11 пар оснований 5'-(А/Т)ТТТАТ(А/G)ТТТ(А/Т)-3') вблизи 5'-конца одной из цепей ДНК. Также IIe47 и Asn51 образуют связи вблизи 3'-конца ТААТ сайта. Исходя из этих данных, можно сделать вывод, что предположение о роли аминокислотных остатков в распознавании последовательности ДНК было сделано верно. Далее рассмотрим остатки Arg5 и Asn51. Их взаимодействия с ДНК показаны на рисунках 9 и 10.
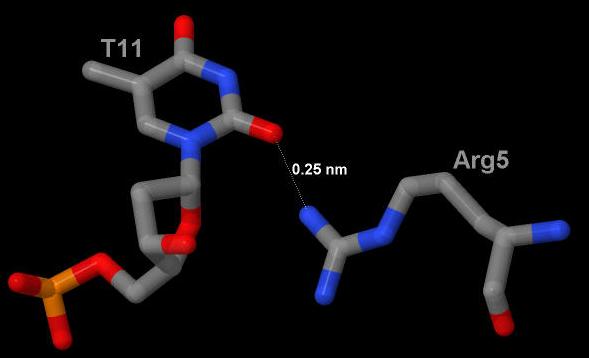
Рис.9. Связь атома азота из Arg5 с атомом кислорода из тимина-11. Эта связь является полярной. Её длина составляет 0.25 нм.
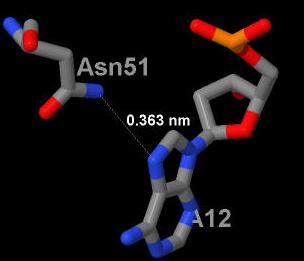
Рис.10. Рис.9. Связь между атомами азота из Asn51 и аденина-12. По определению полярной связи, введенному выше, эта связь полярная. Её длина составляет 0.363 нм.
Предсказание вторичной структуры заданной тРНК
Предсказание вторичной структуры тРНК с PDB-кодом 2DLC путем поиска инвертированных повторов
Для поиска инвертированных повторов использовалась программа einverted из пакета EMBOSS:
einverted 2DLC-2.fasta Find inverted repeats in nucleotide sequences Gap penalty [12]: Minimum score threshold [50]: 15 Match score [3]: Mismatch score [-4]: Sanger Centre program inverted output file [sequence.inv]: File for sequence of regions of inverted repeats. [sequence.fasta]:
Был получен файл sequence.inv, в котором показаны следующие инвертированные повторы:
SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps 27 caaga 31 ||||| 43 gttct 39 SEQUENCE: Score 15: 5/5 (100%) matches, 0 gaps 49 gggcg 53 ||||| 65 cccgc 61
Предсказание вторичной структуры тРНК по алгоритму Зукера.
С помощью онлайн версии программы mfold The mfold Web Server было построено изображение предположительной вторичной стурктуры тРНК. С параметром P=15% (параметр означает, на сколькопроцентов выдаваемое предсказание структуры может отличаться по своей вычисленной энергии от оптимального) было получено пять стурктур, из них была выбрана одна наиболее подходящая.
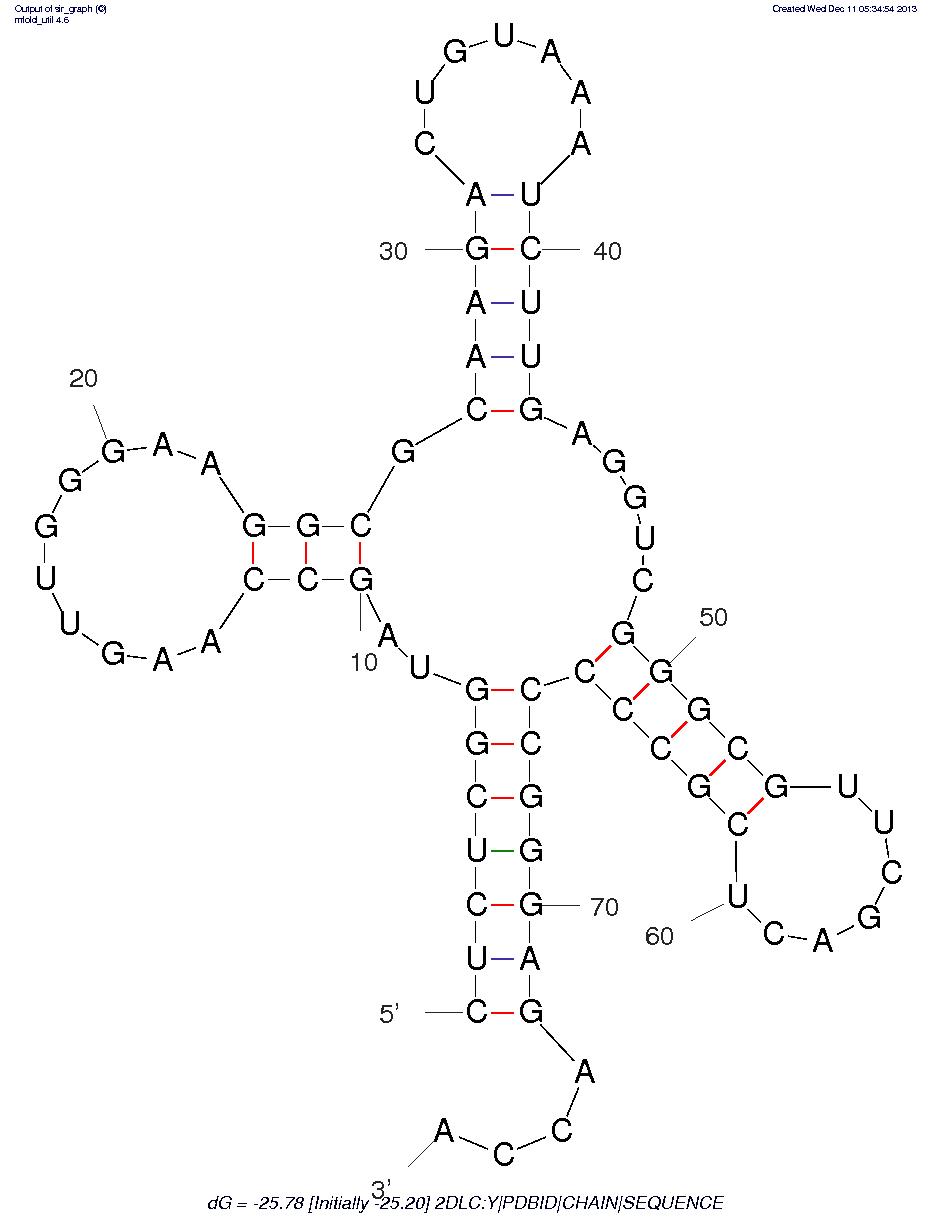
Рисунок 11. Предсказание вторичной структуры тРНК 2DLC, полученное с помощью алгоритма Зукера.
Таблица 2. Сравнение реальной и предсказанной вторичной структуры тРНК из файла 2DLC.pdb
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-501-507-3' 5'-566-572-3' всего 7 пар | - | 5'-501-507-3' 5'-566-572-3' всего 7 пар |
| D-стебель | 5'-510-513-3' 5'-522-525-3' всего 4 пары | - | 5'-510-512-3' 5'-523-525-3' всего 3 пары |
| T-стебель | 5'-549-553-3' 5'-561-565-3' всего 5 пар | 5'-549-553-3' 5'-561-565-3' всего 5 пар | 5'-549-553-3' 5'-561-565-3' всего 5 пар |
| Антикодоновый стебель | 5'-526-528-3' 5'-542-544-3' всего 5 пар | 5'-527-531-3' 5'-539-543-3' всего 5 пар | 5'-527-531-3' 5'-539-543-3' всего 5 пар |
| Общее число канонических пар нуклеотидов | 21 | 10 | 20 |
Как видно из таблицы, с помощью алгоритма Зукера можно получить результат очень близкий к настоящему. Предсказание с помощью инвертированных повторов не дало хороших результатов.