Программа getorf пакета EMBOSS
Был создан файл с записью D89965 банка EMBL:
entret embl:d89965
Далее была запущена программа getorf так, чтобы получить набор трансляций всех открытых рамок данной последовательности длиной не менее 30 аминокислотных остатков, которые определены при использовании стандартного кода и начинаются со старт-кодона (или начала последовательности) и заканчиваются стоп-кодоном (или концом последовательности):
getorf -minsize 90 -find 1 -table 0
Был получен файл d89965.orf с последовательностью пяти рамок считывания. В поле FT файла d89965.entret приведена третья кодирующая последовательность (CDS). Далее был создан файл hslv_ecoli.fasta с последовательностью записи SwissProt, на которую ссылается данная запись EMBL:
seqret hslv_ecoli.entret hslv_ecoli.fasta
Далее была определена рамка, которой соответствует эта последовательность:
blastp -query hslv_ecoli.fasta -subject d89965.orf -out hslv.blastp
Это рамка считывания под номером пять.
Можно заметить несоответствие.
Аннотация в embl указывает на запись в банке SwissProt. Согласно ей обсуждаемый белок закодирован на прямой третьей рамке считывания и является белком бактерии E.coli P0A7B8 (HSLV_ECOLI). Однако, при получении всех возможных рамок считывания из гена Rattus norvegicus, этот белок выравнивается с обратной пятой рамкой, причем не целиком. Как такое могло произойти?
Предположительно, исследователи искали ген определенного белка, о котором они знали такие данные как массу и заряд. Препарат мРНК, который использовался для определения белка, был получен из желудка крысы. Препарат был отсеквенирован, и была определена подходящая рамка считывания из генома крысы. Однако в препарате, скорее всего, оказалась бактериальная ДНК, и на самом деле этот белок принадлежит не крысе, как хотели показать авторы, а E.coli. Для такого белка, принадлежащего крысе, нет записи в SwissProt. Поэтому была сделана ссылка на единственную существующую запись, подходящую к указанной рамке считывания. То есть третья рамка Rattus norvegicus оказалась некодирующей, зато обнаружилась пятая рамка E.coli на которой на самом деле закодирован этот белок. Такие ошибки встречаются достаточно редко, потому что для этого нужно существование больших некодирующих рамок считывания, а средняя длина рамки между стоп кодонами примерно 21 кодон. В записи P0A7B8 SwissProt есть ссылка на обсуждаемую запись в EMBL с указанием на ошибку аннотации.
Файлы-списки
Для создания файла-списка нужных последовательностей белков в файл adh.fasta в fasta-формате были скопированы все доступные в Swissprot последовательности алкогольдегидрогеназ:
seqret sw:adh*_* adh.fasta
Затем был получен файл с универсальными адресами (USA) этих последовательностей:
infoseq -only -usa > listfile.txt
Из этого файла-списка был получен другой, меньший, с адресами последовательностей, взятых из группы организмов №3 (файл list.txt):
grep -f list.txt listfile.txt > listfile2.txt
На основе нового файла-списка (listfile2.txt) был получен fasta-файл с последовательностями дегидрогеназ организмов группы №3:
seqret @listfile2.txt listfile2.fasta
Ссылка на файл с последовательностями алкогольдегидрогеназ орагнизмов из группы №3.
EnsEMBL
Изучение портала EnsEMBL было проведено на примере гена, кодирующего белок ACHA9_HUMAN Neuronal acetylcholine receptor subunit alpha-9. По записи в SwissProt был определен AC в банке EMBL. Была получена запись из банка EMBL. Согласно ей, этот ген содержит 4 экзона, поэтому для дальнейшей работы использовалась вся кодирующая последовательность гена aam74523.fasta:
entret sw:ACHA9_HUMAN entret embl:AAM74523.1 seqret aam74523.entret
В выдаче "BLAST/BLAT" с параметром Search Tool "BLAT" показаны результаты выравнивания запроса с человеческим геномом, в том числе его положение на хромосоме (в данном случае, это хромосома 4).
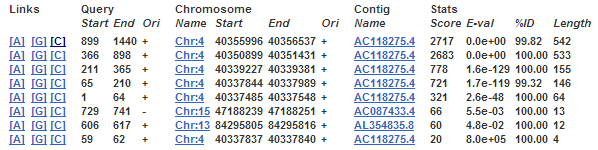
Рисунок 1.
На рисунке 1 показаны результаты выравнивания. 5 участков последовательно образуют хорошее выравнивание на четвертой хромосоме. Ссылка "Contig view" отправляет нас страницу с детальным описанием участка хромосомы, с которым проведено выравнивание. На схеме показана разметка хромосомы (номера пар оснований), названия контигов и закодированные гены с указанием их типа. Ниже показано выравнивание запроса с участком хромосомы, также приведены кДНК, кодирующие участки генов, processed transcript - транскрипт, который не содержит ORF и количество и расположение constrained elements - участков множественного выравнивания, обладающих высокой консервативностью. Также показан график кодирующего потенциала. Прямо на этой странице можно выбрать организим или группу организмов и посмотреть выравнивание с их геномами, а также сравнить результаты. Изображение показано на рисунке 2.

Рисунок 2.
Сбоку страницы есть меню Location-based displays, с его помощью можно перейти к различным другим отображениям выравнивания, к текстовой выдаче.