Построение моделей структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA
Аббривиатура 3DNA расшифовывается как 3-Dimensional Nucleic Acid. Этот пакет программ применяется для анализа, моделирования и визуализации структур нуклеиновых кислот. Он работает под операционной системой LINUX, поэтому с помощью программы Рutty, используя протокол ssh, подсоединяемся к серверу kodomo.cmm.msu.ru, заходим в рабочую директорию и указываем путь к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
Затем, используя программу fiber пакета 3DNA, строим A- и B- формы ДНК из 20 пар оснований так, чтобы они представляли собой повторы последовательности из четырех разных нуклеотидов, и Z-форму из 10 повторов GC. Ссылки на полученные файлы:
A-форма ДНК
B-форма ДНК
Z-форма ДНК
Работа со структурами нуклеиновых кислот в JMol
Выделение разных атомов и химических группировок, используя предопределенные множества JMol
На рисунках 1, 2, 3 и 4 представлены результаты выполнения задания на примере A-формы ДНК gcta_a.pdb.
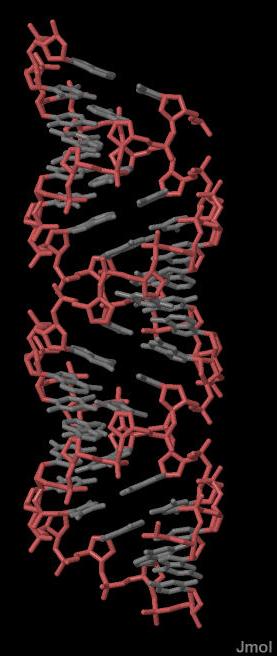
Рис.1. Сахарофосфатный остов выделен розовым. Ссылка на скрипт. |
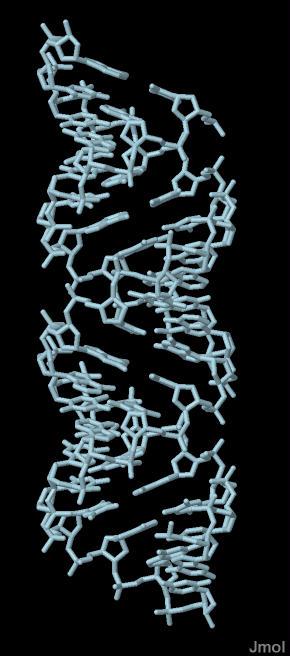
Рис.2. Голубым цветом выделены все нуклеотиды, но так как эта молекула состоит только из нуклеотидов, она окрашивается целиком. Ссылка на скрипт. |
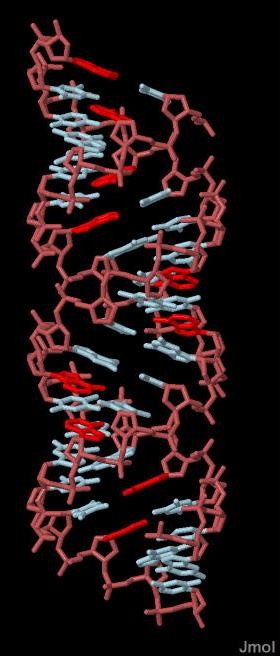
Рис.3. Красным выделены аденины. Ссылка на скрипт. |
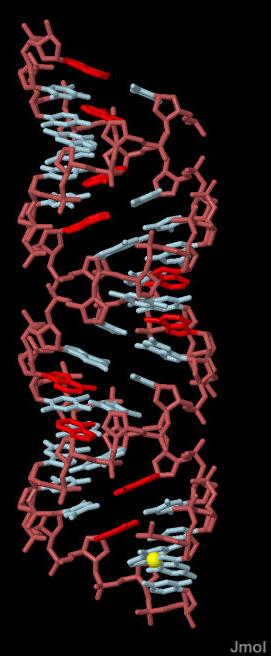
Рис.4. Атом N7 первого гуанина в последовательности выделен желтым и представлен в виде шарика. Ссылка на скрипт. |
Работа со структурами комплексов нуклеиновых кислот и белков, полученных в PDB.
Из PDB в рабочую директорию были скопированы структуры комплексов тРНК-белок 2DLC и ДНК-белок 1HDD. Для проверки цепей ДНК и РНК на наличие разрывов обе структуры были рассмтрены в Jmolв видк проволочной модели. На рисунках 5 и 6 можно увидеть, что разрывов в структурах нет. Однако для структуры тРНК наблюдаются разрывы остова молекулы, которые видно при представлении её с помощью команд backbone или cartoon. На рисунке 7, представленном в PDB, эти разрывы также видны. Такая ситуация объясняется наличием модифицированных нуклеотидов, включение которых нарушает структуру нарушает сахарофосфатного остова.
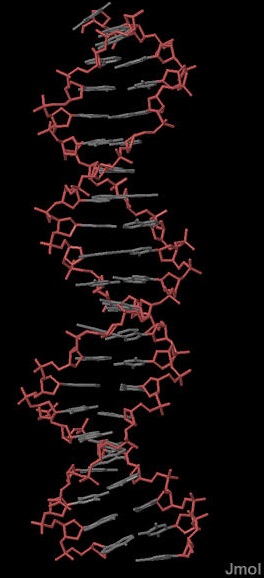
Рис.5. ДНК из структуры 1HDD в проволочной модели. Розовым выделен сахарофосфатный остов |
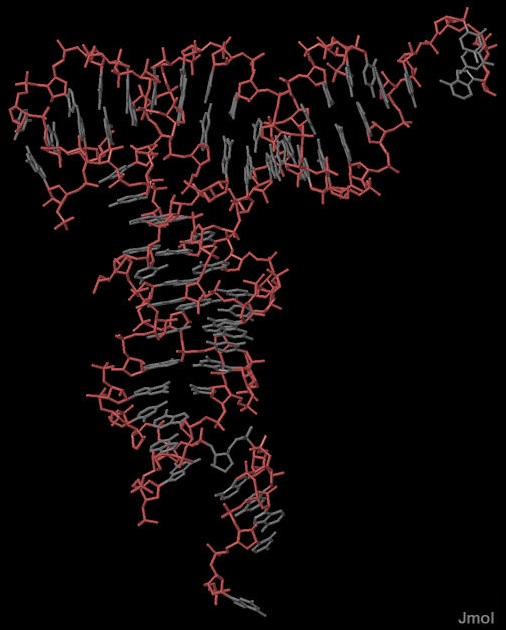
Рис.6. тРНК из структуры 2DLC в проволочной модели. Розовым выделен сахарофосфатный остов |
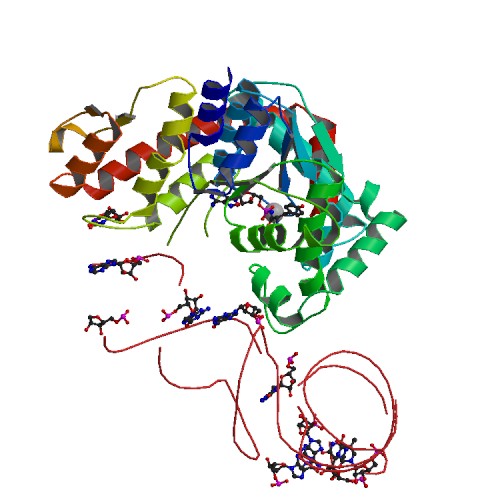
Рис.7. Изображение структуры 2DLC, приведеннное в PDB. Видно, что остов молекулы тРНК прерывается модифицированными нуклеотидами.
Координаты атомов только ДНК и РНК из данных стуктур были найдены с помощью команды grep. Ссылки:
Координаты атомов ДНККоординаты атомов РНК
Сравнение структур трех форм ДНК с помощью средств JMol
Большие и малые бороздки ДНК
На рисунке 8 представлена B-форма ДНК gcta_b.pdb. Рассмотрим тимин. Атомы С6, C5, C5M, C4, O4 выделены красным, видно, что они обращены в сторону большой бороздки. Атомы N1, C2, O2 выделены синим, они обращены в сторону маленькой бороздки.
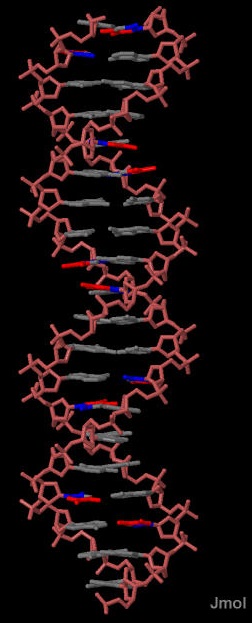
Рис.8. B-форма ДНК. Атомы С6, C5, C5M, C4, O4 тимина выделены красным, а N1, C2, O2 - синим.
В случае A- и Z-форм ДНК ориентация этих атомов другая. В A-форме ДНК атомы тимина, которые в B-форме были напралены в большую бороздку направляются в центр молекулы, а те, которые были направлены в малую бороздку - наружу, это показано на рисунке 9. В Z-форме тимина нет, зато есть цитозин. В B-форме его атомы C6, C5, C4, N4 направлены в большую бороздку, выделим их красным, а атомы N1, C2, O2 - в маленькую, выделим их синим. На рисунке 10 показано, что в Z-форме ДНК "красные" атомы направлены наружу структуры, а "синие" внутрь.

Рис.9. A-форма ДНК. Атомы С6, C5, C5M, C4, O4 тимина выделены красным, а N1, C2, O2 - синим. |
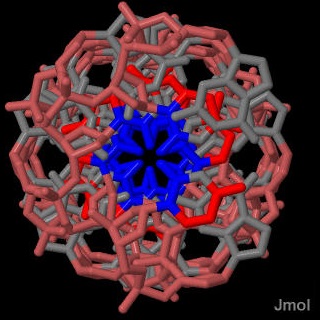
Рис.10. Z-форма ДНК. Атомы C6, C5, C4, N4 цитозина выделены красным, а N1, C2, O2 - синим. |
Далее показаны полученные в ChemSketch изображения тимина и цитозина с выделением атомов, смотрящих в сторону большой бороздки красным, а в сторону малой - синим.
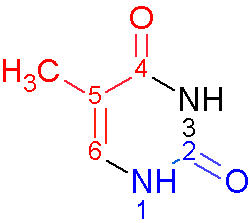
Рис.11. Тимин. |
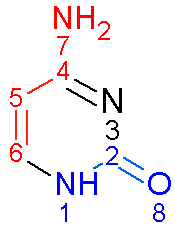
Рис.12. Цитозин. |
Для тимина:
- в сторону малой бороздки обращены атомы: С6, C5, C5M, C4, O4;
- в сторону малой бороздки обращены атомы: N1, C2, O2;
- остальные атомы: N3.
Сравнение основных спиральных параметров разных форм ДНК
Таблица 1. Сравнение основных спиральных параметров разных форм ДНК.
| A-форма | B-форма | *Z-форма | |
| Тип спирали (правая или левая) | Правая | Правая | Левая |
| Шаг спирали (A) | 28.03 | 33.75 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки, нм | 1.681 ([A]30:B.P #595 - [G]13:A.P #247) | 1.721 ([T]33:B.P #657 - [G]5:A.P #83) | 1.83 ([C]12:A.P #228 - [C]26:B.P #515) |
| Ширина малой бороздки, нм | 0.798 ([A]4:A.P #62 - [G]31:B.P #616) | 1.169 ([C]14:A.P #269 - [G]31:B.P #616) | 0.868([G]35:B.P #698 - [G]11:A.P #206) |
Сравнение торсионных углов для А- и В- форм ДНК
В таблице 2 приведены значения торсионных углов для А- и В- форм ДНК на примере тимина. Нужно отметить, что для других аминокислот они почти не будут отличаться. Звездочкой отмечены атомы предыдущих и следующих нуклеотидов.
Таблица 2. Сравнение торсионных углов для А- и В- форм ДНК.
| alpha(O3'*-P-O5'-C5') | beta (P-O5'-C5'-C4') | gamma (O5'-C5'-C4'-C3') | delta (C5'-C4'-C3'-O3') | epsilon (C4'-C3'-O3-P'*) | xi (C3'-O3-P'*-O5'*) | chi (C2-N1-C4'-O4') | |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
Таблица 3. Сравнение торсионных углов для А- и В- форм ДНК, данные приведенные в презентации.
| alpha(O3'*-P-O5'-C5') | beta (P-O5'-C5'-C4') | gamma (O5'-C5'-C4'-C3') | delta (C5'-C4'-C3'-O3') | epsilon (C4'-C3'-O3-P'*) | xi (C3'-O3-P'*-O5'*) | chi (C2-N1-C4'-O4') | |
| A-форма | 62 | 173 | 52 | 88/3 | 178 | -50 | -160 |
| B-форма | 63 | 171 | 54 | 123/131 | 155 | -90 | -117 |
Значения торсионных углов, полученные из сгенерированных программой fiber структур нуклеиновых кислот, в большинстве своём отличаются от значений из презентации. Однако некоторые углы, например beta для А-формы, совпадают. Это может быть связано с тем, что для измерения углов были выбраны другие атомы, или с различными условиями получения этих углов. Ведь в первом случае углы измерялись для ненастоящих структур, возможно что в презентации приведены данные измеренные на реальной молекуле.
Значения торсионных углов для структуры тРНК 2DLC
В таблице 4 показаны средние значения торсионных углов для структуры тРНК 2DLC. Стоит отметить, что из-за нерегулярной структуры тРНК значения торсионных углов разных нуклеотидов очень сильно варьируются, поэтому по среднему значению нелья делать вывод о возможной структуре. Значения некоторых углов (zeta, chi, alpha и gamma) очень похожи на значения для А-формы.
Таблица 4. Средние значения торсионных углов для структуры тРНК 2DLC.
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| Strand I | -34.34 | 52.63 | 49.89 | 87.29 | -115.62 | -56.13 | -162.15 |
| Strand II | -39.87 | 86.18 | 48.24 | 89.80 | -103.44 | -53.02 | -157.30 |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Определение торсионных углов в структуре ДНК 1HDD
Таблица 5. Значения торсионных углов для структуры ДНК 1HDD.
| Strand I | Strand II | |||||||||||||||||
| base | alpha | beta | gamma | delta | epsilon | zeta | chi | base | alpha | beta | gamma | delta | epsilon | zeta | chi | |||
| 1 | T | --- | -164.6 | 60.4 | 117.3 | -170.7 | -89.7 | -122.3 | 1 | A | -68.4 | 177.6 | 52.1 | 147.7 | --- | --- | -105.4 | |
| 2 | T | -80.4 | 176.3 | 61.3 | 160.8 | -156.2 | -174.9 | -110.5 | 2 | A | -74.7 | -176.3 | 53.8 | 140.7 | -171.1 | -128.9 | -92.2 | |
| 3 | T | -16.8 | 133.1 | 29 | 156.1 | -52.3 | 141.5 | -104.6 | 3 | A | 44 | 164.2 | -50.8 | 158.7 | 164.5 | -81.4 | -86.7 | |
| 4 | G | -32.2 | 123.5 | -21.6 | 142.9 | -168.8 | -132.4 | -86.6 | 4 | C | -64.1 | -163 | 40 | 142.7 | 169.8 | -108.5 | -94.9 | |
| 5 | C | -75.8 | 142.1 | 100.4 | 110.6 | -125.6 | -111.9 | -150.7 | 5 | G | 0.8 | 124.3 | 13 | 155 | -156.1 | -129.5 | -105 | |
| 6 | C | -55.9 | 105.4 | 99.7 | 83.2 | -142 | -116.9 | -159 | 6 | G | -66.3 | -174 | 42.5 | 156 | -157.6 | -169.1 | -98.3 | |
| 7 | A | -25.6 | 141.5 | 43.6 | 138.1 | -147.9 | -116.5 | -93.3 | 7 | T | -63.8 | -176.7 | 50.1 | 101.9 | -167.8 | -103.4 | -120.2 | |
| 8 | T | -48.3 | 145 | 56.1 | 101 | -173.2 | -92.3 | -142.8 | 8 | A | 0.2 | 80.3 | 34.3 | 119.4 | 168.8 | -80.4 | -94.3 | |
| 9 | G | -51.3 | -144.9 | 19.8 | 143.4 | 81.6 | 11.6 | -88.2 | 9 | C | -42.7 | 173.3 | 19.4 | 150.4 | -94.9 | 126.3 | -65.6 | |
| 10 | T | -155.8 | -150.5 | 71.4 | 148.5 | -102.9 | 165.1 | -93.9 | 10 | A | -57.5 | 169.6 | 33.2 | 159.7 | -162.2 | -128.7 | -85.1 | |
| 11 | A | -53.3 | 136.1 | 30.3 | 145.5 | -169.7 | -121.6 | -97.4 | 11 | T | 2.1 | 143.6 | 17.1 | 164.5 | -139.7 | -142.8 | -88.4 | |
| 12 | A | -23.7 | 177.1 | 4.5 | 171.8 | -164.8 | -143.7 | -95.7 | 12 | T | -77.8 | 140.3 | 100 | 86.2 | -154.6 | -113.5 | -151.5 | |
| 13 | T | -29 | 179.1 | 17.5 | 160.3 | -141 | -158.1 | -83.5 | 13 | A | -53.3 | 170.3 | 34.6 | 131.3 | -169.5 | -97.5 | -121.8 | |
| 14 | T | -36.2 | 133.6 | 36.3 | 110.1 | -159.1 | -131 | -127.3 | 14 | A | -100.8 | -168.3 | 55.4 | 153.1 | -155.5 | -126.5 | -109.9 | |
| 15 | A | -13.9 | 152.6 | 11.5 | 154.9 | -152.8 | -135.2 | -84.3 | 15 | T | -93.7 | -157.8 | 79.3 | 115.9 | 172 | -56.2 | -139.6 | |
| 16 | C | -52.4 | 155.7 | 39.3 | 109.4 | 161.6 | -81.8 | -117.8 | 16 | G | -58.1 | 164 | 26.2 | 103.2 | 166.4 | -72.3 | -143.9 | |
| 17 | C | -69.2 | -178.2 | 45.9 | 79.1 | 178 | -56.3 | -158.7 | 17 | G | -62.1 | 151.4 | 34 | 159.5 | -141.8 | -164.6 | -84.8 | |
| 18 | T | -100.5 | -161.7 | 90.3 | 139.2 | -169.5 | -90.6 | -109.8 | 18 | A | -31.9 | 157 | 18.7 | 159.5 | -131 | -148 | -86.9 | |
| 19 | A | -63.8 | -173 | 22.3 | 99.7 | 146.5 | -95.4 | -106.2 | 19 | T | -25.7 | 160.3 | 15.4 | 142.5 | -148.3 | -147.8 | -92.5 | |
| 20 | A | 147.1 | -133.1 | -174.4 | 153.2 | --- | --- | -133.1 | 20 | T | --- | -156 | 47.8 | 154.3 | -157.6 | -137 | -100.1 |
Таблица 6. Средние значения торсионных углов для структуры ДНК 1HDD.
| alpha | beta | gamma | delta | epsilon | zeta | chi |
| -50.3 | 52.1 | 38.2 | 134.9 | -71.3 | -92.0 | -107.6 |
Структура 1HDD мало похожа как на А-, так и на В-формы ДНК. Как видно в таблице 4, значения углов для разных нуклеотидов в ней сильно различаются.
Для определения наиболее отклоняющегося нуклеотида были вычислены модули разностей значений всех торсионных углов и средних значений, а номера нуклеотидов расположены по их убыванию. Получилась таблица 6. Среди первых двух строчек наиболее часто встречается номер 17. Его и определим как самый "деформированный".
Таблица 7. Номера нуклеотидов, расположенные по убыванию "отклонения" значений торсионных углов для структуры ДНК 1HDD.
| alpha | beta | gamma | delta | epsilon | zeta | chi |
| 10 | 17 | 5 | 17 | 2 | 10 | 6 |
| 18 | 19 | 6 | 6 | 17 | 3 | 17 |
| 15 | 18 | 4 | 12 | 16 | 9 | 5 |
| 3 | 10 | 18 | 19 | 19 | 2 | 8 |
| 2 | 9 | 12 | 8 | 9 | 13 | 13 |
| 12 | 13 | 10 | 2 | 8 | 12 | 15 |
| 5 | 12 | 15 | 16 | 11 | 15 | 4 |
| 7 | 2 | 2 | 13 | 18 | 4 | 14 |
| 13 | 16 | 13 | 14 | 4 | 14 | 9 |
| 17 | 15 | 9 | 5 | 12 | 17 | 7 |
| 4 | 8 | 8 | 3 | 14 | 11 | 10 |
| 14 | 5 | 19 | 15 | 15 | 6 | 12 |
| 19 | 7 | 3 | 10 | 7 | 7 | 16 |
| 6 | 11 | 11 | 11 | 6 | 5 | 11 |
| 11 | 14 | 17 | 9 | 13 | 16 | 3 |
| 16 | 3 | 7 | 4 | 5 | 19 | 2 |
| 8 | 4 | 14 | 18 | 10 | 18 | 18 |
| 9 | 6 | 16 | 7 | 3 | 8 | 19 |
Водородные связи в структуре РНК 2DLC.
Стебли в структуре образуют нуклеотиды с номерами 501-508, 510-515, 522-528, 542-544, 548-556, 558, 561-572. На рисунке 13 они выделены розовым.
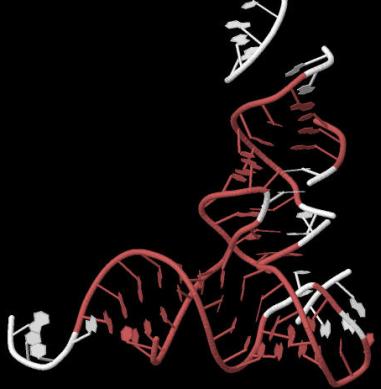
Рис.13. Стебли в структуре РНК 2DLC.
В структуре 2DLC присутсвуют неканонические пары оснований. Они представлены на рисунке 14.

Рис.14. Неканонические пары оснований в структуре РНК 2DLC.
В рассматриваемой тРНК есть дополнительные водородные связи, стабилизирующие ее третичную структуру. Они представлены на рисунке 15. Можно заметить, что четыре из пяти этих связей образованы неканоническими парами оснований.

Рис.15. Дополнительные водородные связи в структуре РНК 2DLC.
Стекинг-взаимодействия в структуре тРНК 2DLC
В файле 2DLC_old.out находим данные о величине площади "перекрывания" 2-х последовательных пар азотистых оснований. Среди них наибольшее значение определяется для пар 13 uP/ga, можно заметить, что она образована модифицированными основаниями. В разделе файла 2DLC_old.out с описанием водородных связей находим эти пары. Определяем, что u - это 5-метилуридин, a - это 6-гидро-1-метиладенозин, P - это псевдоуридин, а g - это О2'-метилгуанозин. Изображение этих пар представлено на рисунке 16.
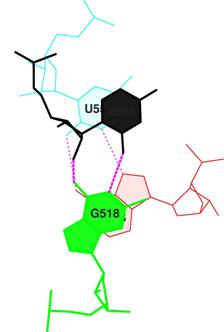
Рис.16. Пары оснований uP/ga.
Следующее по величине значение площади "перекрывания" определяется для пар 10 GC/GC, образованнной немодифицированными основаниями. Их изображение можно увидеть на рисунке 17. Значение для этих пар приводится для сравнения, чтобы не создавалось впечатления, что модифицированные остатки имеют необычно большие площади "перекрывания".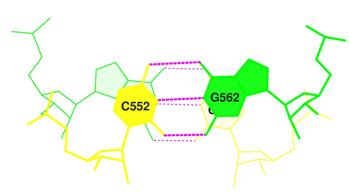
Рис.17. Пары оснований GC/GC.
В качестве примера для пар с минимальным перекрыванием были взяты пары 23 GG/Cc, где с - это 5-метилцитидин, и 14 PU/Ag, где P - это псевдоуридин, а g - это О2'-метилгуанозин. Для них указано значение 0.00. Их изоборажения показаны на рисунках 18 и 19.
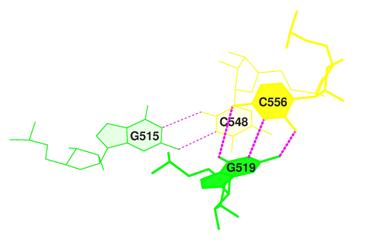
Рис.18. Пары оснований GG/Cc. |
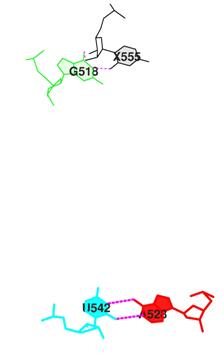
Рис.19. Пары оснований PU/Ag. |
Для получения изображений в файле staking.pdb находим номера структур, описывающих координаты искомых нуклеотидных остатков, а затем вырезаем эти структуры в отдельный файл с помощью команды
ex_str -n stacking.pdb step_n.pdb, где n - это номер структуры.
Затем, с помощью команды
stack2img -cdolt step_n.pdb step_n.ps
строим изображение.
Номера структур совпадают с номерами пар в файле 2DLC_old.out.