Эволюционные домены (part I)
Описание домена P_proprotein (PF01483)
Для изучения непрерывной эволюции доменов был выбран домен P_proprotein (PF01483). Для него известно:
- 112 архитектур;
- 1577 последовательностей;
- 2 взаимодействия;
- 646 видов;
-15 структур.
Информация о домене представлена в таблице 1.
Таблица 1.Описание домена P_proprotein (PF01483).
| AC | ID | Функция домена | Ссылка на страницу домена в Pfam |
| PF01483 | P_proprotein | Необходим для поддержания структуры и активности каталитического домена некоторых эндопептидаз. Возможны и другие функции, так как домен недостаточно изучен. | http://pfam.xfam.org//family/PF01483 |
В программе JalView было построено выравнивание последовательностей данного домена. Последовательность белка KEX2_YEAST была связана с его структурой (PDB-код: 1OT5). Последовательности раскрашены по консервативности ClustalX с порогом 20%.
Информация об архитектурах
В рабочей директории был запущен скрипт
python swisspfam_to_xls.py -p PF01483 -i /srv/databases/pfam/swisspfam.gz -z -o arch_text.txt
для получения информации об архитектуре всех последовательностей, содержащих выбранный домен. Был получен файл arch_text.txt. Затем этот файл был переведен в формат книги Excel, сделана сводная таблица, отражающая количество архитектур определенного типа в белках. Таксономия организмов, которым принадлежат белки была получена с использованием Uniprot и скрипта
python uniprot_to_taxonomy.py -i 20140517919A5DXIPB.txt -o arch_tax.txt
Посмотреть примечание
В таблицу также была добавлена колонка с длиной выбранного домена из каждой последовательности. Таблицу можно скачать по ссылке. Список архитектур с из визуализацией можно посмотреть в Pfam, пройдя по ссылке. Для дальнейшей работы были выбраны две архитектуры:

Рисунок 1. Схема архитектуры Peptidase_S8 (PF00082), P_proprotein (PF01483). Всего в Pfam найдено 778 последовательностей с данной архитектурой.

Рисунок 2. Схема архитектуры Peptidase_S8 (PF00082), P_proprotein (PF01483), GF_recep_IV (PF14843). Всего в Pfam найдено 74 последовательности с данной архитектурой.
Далее архитектуру Peptidase_S8, P_proprotein будем называть архитектурой 1, а Peptidase_S8, P_proprotein, GF_recep_IV архитектурой 2. Домен Peptidase_S8 характерен для семейства Subtilase сериновых протеаз, а GF_recep_IV - это внеклеточный домен рецепторов-тирозинкиназ. Совместное расположение доменов Peptidase_S8 и P_proprotein можно объяснить с точки зрения функции домена P_proprotein. В его описании утверждается, что он необходим для поддердания работы каталитического домена эндопептидаз. Соответственно, они должны находиться рядом.
Таксономия последовательностей для обсуждаемых архитектур вынесена на соответствующий лист таблицы.
Выбор таксона и подтаксонов
Так как выбранные архитектуры в основном представлены в эукариотических организмах, а именно в животных, в качестве таксона было выбрано царство Metazoa, а в качестве подтаксонов тип Chordata и клада Ecdysozoa. Последовательности доменов из данных архитектур были разбиты на соответствующие группы, и было сделано выравнивание. Каждая группа раскрашена по консервативности ClustalX с порогом 20%. Отдельно на выравнивании выделена последовательность домена, к которой прикреплена структура. Проект в формате .jar можно скачать по ссылке. Изображение выравнивания показано на рисунке 3.

Рисунок 3. Выравнивание последовательностей доменов из указанных архитектур. Сверху вниз: последовательность домена, к которой прикреплена структурапоследовательность домена, к которой прикреплена структура, группа последовательностей из архитектуры 1 и группа последовательностей из архитектуры 2.
При составлении выравнивания были удалены все пустые колонки и выступающие С-концевые участки. На выравнивании четко прослеживается консервативность во многих колонках.
Эволюционные домены (part II)
Построение дерева
Так как для работы были выбраны две архитектуры в таксонах Chordata (обозначение С) и Ecdysozoa (обозначение Е), названия последвательностей были изменены по типу: номер архитектуры_таксон_ID(белка). Номера архитектур Peptidase_S8, P_proprotein и Peptidase_S8, P_proprotein, GF_recep_IV 1 и 2 соответственно.
Таблица 1. Названия последовательностей.
| Название последовательностей | Расшифровка |
| 1_E_ID(белка) | Архитектура Peptidase_S8, P_proprotein таксон Ecdysozoa |
| 1_С_ID(белка) | Архитектура Peptidase_S8, P_proprotein таксон Chordata |
| 2_E_ID(белка) | Архитектура Peptidase_S8, P_proprotein, GF_recep_IV таксон Ecdysozoa |
| 2_C_ID(белка) | Архитектура Peptidase_S8, P_proprotein, GF_recep_IV таксон Chordata |
Далее по выравниванию было построено дерево. Для построения дерева был выбран метод Maximum Likelihood в программе Mega. Построение дерева выполнялось с использованием bootstrap. Метод Maximum Likelihood не предполагает молекулярные часы. В результате было получено дерево, показанное на рисунке 1:

Рисунок 1. Дерево, полученное с bootstrap. Описание в тексте.
По умолчанию программа укореняет дерево в среднюю точку. Положение корня дерева было изменено. Полученный результат можно увидеть на рисунке 2.
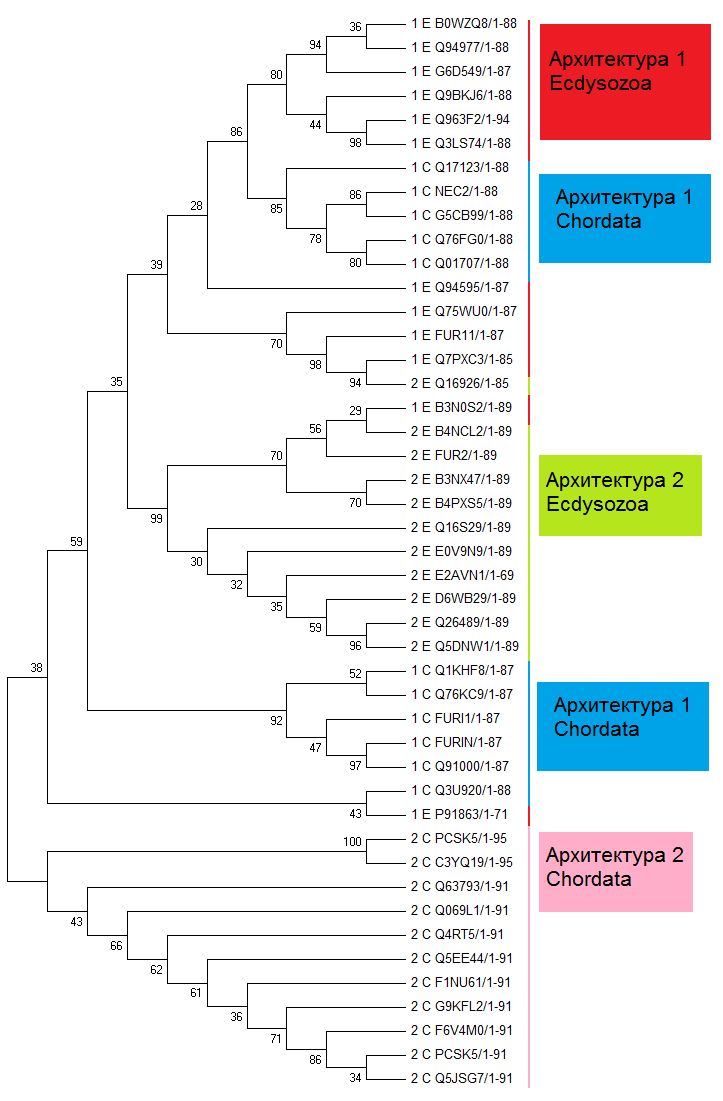
Рисунок 2. Переукорененное дерево.
Выводы
Начиная рассматривать дерево с корня, можно заметить, что оно разделяется на две ветви: одна с архитектурой 1 (Peptidase_S8, P_proprotein), а вторая затем тоже делится на домены из архитектуры 2 (Chordata), и другую ветвь (архитектура 2 Ecdysoszoa, архитектура 1 Chordata и Ecdysoszoa). Получается, что сначала эволюция архитектуры 1 пошла по двум путям. В одном из этих путей к ней прибавился домен GF_recep_IV. Далее эта архитектура 2 эволюционировала независимо от архитектуры 1. Причем, можно заметить, что архитектура 2 возникала потом еще один раз (архитектура 2 Ecdysoszoa).