Описание трансмембранных белков с известной 3D структурой
В базе данных Orientations of Proteins in Membranes были выбраны три белка с трансмембранными участками, представленными альфа-спиралями, и три белка с трансмембранными бета-бочонками. Характеристика структур представлена в таблице 1.
Таблица 1. Описание трансмембранных белков с известной 3D структурой.
| PDB код | Тип | Какая мембрана | Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 3a7k | Спираль | Клеточная мембрана археи Natronomonas pharaonis | 33.8 ± 1.4 A | 21 |
| 4iar | Спираль | Клеточная мембрана эукариотической клетки (Homo sapiens) | 33.6 ± 1.8 A | 24 |
| 2zd9 | Спираль | Внутренняя мембрана Грам-отрицательной бактерии Rhizobium loti | 31.6 ± 1.2 A | 20 |
| 2mpr | Бета-бочонок | Внешняя мембрана Грам-отрицательной бактерии Salmonella enterica | 24.5 ± 1.0 A | 10 |
| 4bum | Бета-бочонок | Внешняя мембрана митохондрии (Danio rerio) | 23.4 ± 0.9 A | 7 |
| 3b07 | Бета-бочонок | Клеточная мембрана Грам-положительной бактерии Staphylococcus aureus (секретируемый белок) | 21.4 ± 1.0 A | 7 |
Как видно из таблицы, среднее число остатков, приходящихся на трансмембранный сегмент спиралей, примерно в два раза больше, чем для бета-бочонков. Однако, все структуры с бочонками были рассмотрены в мембранах с толщиной около 20 A, а все спирали - около 30 A. Исходя из полученных данных можно утверждать, что количество остатков в трансмембранном сегменте увеличивается с увеличением толщины мембраны (стоит отметить, что во всех рассмотренных структурах трансмембранный участок полностью пронизывает мембрану, поэтому такой вывод достаточно очевиден. Если бы были рассмотрены белки, трансмембранный участок в которых занимает лишь часть толщины мембраны, такой связи бы не наблюдалось). Также можно заметить, что клеточные мембраны архей, эукариот и внутренняя мембрана рассмотренной Грам отрицательной бактерии толще, чем мембраны Грам-положительных бактерий, митохондрий и чем внешняя мембрана рассмотренной Грам-отрицательной бактерии.
Отбор гомологов белка Phospho-N-acetylmuramoyl-pentapeptide-transferase (PDB-код 4J72)
Поиск гомологов проводился с использованием BLAST по белкам (БД refseq). Так как белок принадлежит бактерии Aquifex aeolicus для поиска были исключены белки бактерий этого типа. С порогом Expect threshold = 1 были отобраны следующие гомологи. Все они принадлежат бактериям и имеют достаточно высокое сходство. Также был осуществлен поиск по всем организмам, кроме бактерий, и отобрана другая группа гомологов. Все гомологи из этой группы не имеют полного "перекрытия" по последовательностям и в основном принадлежат археям и растениям. Предсказанные белки не включались в выборки.
Анализ структуры белка Phospho-N-acetylmuramoyl-pentapeptide-transferase (PDB-код 4J72)
Анализ структуры представлен в таблице 2.
Таблица 2.Описание структуры трансмембранного белка Phospho-N-acetylmuramoyl-pentapeptide-transferase (PDB-код 4J72, цепь A (цепь B идентична, так как белок - гомодимер).
| PDB код | Организм | Тип мембраны | TC-код | Угол наклона спиралей к нормали | Количество трансмембранных спиралей |
| 4j72 | Aquifex aeolicus | - | Внутренняя мембрана Грам-отрицательной бактерии | 0 ± 0° | 20 |
К сожалению, структура 4j72 не была найдена в БД TCBD. Для определения кода был осуществлен поиск структур гомологов с помощью BLAST, однако такие структуры не были обнаружены. Поиск по БД PDB по запросу "Phospho-N-acetylmuramoyl-pentapeptide-transferase" также не дал положительных результатов. Таким образом, структур, гомологичных данной обнаружено не было, и определить TC-код не удалось.
Анализ множественного выравнивания трансмембранных белков
Так как гомологи из первой выборки очень похожи друг на други и анализировать их выравнивание не имеет смысла (показано на рисунке 1), был проведен анализ выравнивания для второй выборки (рисунок 2). Выравнивание было построено программой Muscle. Последовательности окрашены с учетом гидрофобности остатков (Hydrophobicity), с порогом по консервативности 25%. В аннотации TM_REAL указаны альфа-спирали, предсказанные для белка с последовательностью ref|YP_002049050.1|[Paulinella chromatophora]. Изображение предсказания, полученное в программе TMHMM показано на рисунке 3. Аннотация TM_PREDICTED отражает положение альфа-спиралей в исследуемом белке (Phospho-N-acetylmuramoyl-pentapeptide-transferase (PDB-код 4J72)). Номера остатков, образующих спирали, получены из БД Uniprot.

Рисунок 1. Множественное выравнивание бактериальных гомологов белка Phospho-N-acetylmuramoyl-pentapeptide-transferase (PDB-код 4J72).

Рисунок 2. Множественное выравнивание гомологов белка Phospho-N-acetylmuramoyl-pentapeptide-transferase (PDB-код 4J72) из архей и растений. Пояснения в тексте.
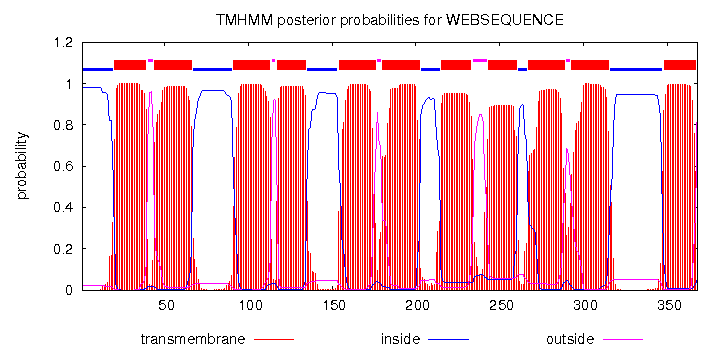
Рисунок 3. Предсказание расположения фрагментов белка с последовательностью ref|YP_002049050.1|[Paulinella chromatophora] относительно мембраны.

Рисунок 4. Изображение структуры белка Phospho-N-acetylmuramoyl-pentapeptide-transferase (PDB-код 4J72). Показано расположение белка в мембране. Один из двух идентичных мономеров, составляющих белок окрашен согласно выравниванию.
Описание полученных результатов
На рисунке 2 видно, что трансмембранные участки достаточно консервативны, в отличие от участков между спиралями. В трансмембранных участках среди консервативных остатков встречаются как полярные (например, S, T, Q), так и заряженные (например, E, D). В положении 541 на выравнивании есть достаточно консервативный столбец из остатков E, D и S, а в положении 506 есть столбец из остатков D и K. Однако, таких мест в выравнивании совсем немного, в основном консервативные столбцы представлены остатками G, L, I и другими неполярными и ароматическими. Наличие заряженных и полярных остатков в трансмембранных участках можно объяснить их стабилизацией за счет взаимодействия с соседними остатками, а также наличием у этих остатков важных функций. Например, если белок осуществляет транспорт через мембрану, они могут быть нужны в канале для взаимодействия с транспортируемыми молекулами.
Положение предсказанных альфа-спиралей на выравнивании почти полностью совпадает с реальными, отмеченными для исследуемого белка. Лишь одна спираль является лишней. Она находится в самом начале выравнивания в неконсервативной области. Возможно, она определена ошибочно. Однако, нужно отметить, что последовательность белка, для которго предсказана эта спираль в данном участке состоит почти полностью из гидрофобных остатков, не считая одного S. У других белков на этом участке такого не наблюдается. Возможно, что данный участок погружен в мембрану только у этого белка.