Таксономия бактерий из задания 1
| Название | Мнемоника | Таксономия |
| Bacillus anthracis | BACAN | Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| Clostridium botulinum | CLOB1 | Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Lactococcus lactis | LACLM | Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Clostridium tetani | CLOTE | Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Streptococcus pneumoniae | STRPN | Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
| Geobacillus kaustophilus | GEOKA | Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Staphylococcus aureus | STAA1 | Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Staphylococcus epidermidis | STAES | Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
На дереве отобранных бактерий ветви, выделяющие следующие таксоны: классы Clostridia и Bacilli, порядки Bacillales и Lactobacillales, семейства Bacillaceae и Staphylococcaceae. Соответствующие ветви показаны на рисунке 1.
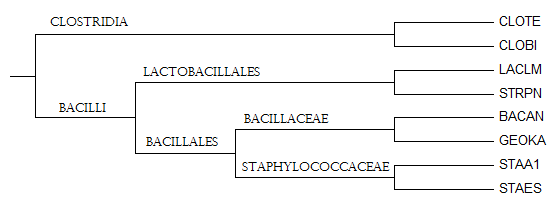
Рисунок 1. Филогенетическое дерево, указанных выше бактерий.
Реконструкция филогенетического дерева
Для реконструкции были выбраны белки семейства "Рибосомный белок S2" (RS2). Идентификаторы белков в SwissProt будут иметь вид RS2_XXXXX, где XXXXX - это мнемоника бактерии. Был получен fasta-файл с указанными последовательностями и сделано множественное выравнивание программой muscle. Выравнивание представлено на рисунке 2. Цветами отмечены возможные диагностические позиции выравнивания: зеленым - порядок Lactobacillales, желтым - семейство Staphylococcaceae, красным - класс Clostridia.
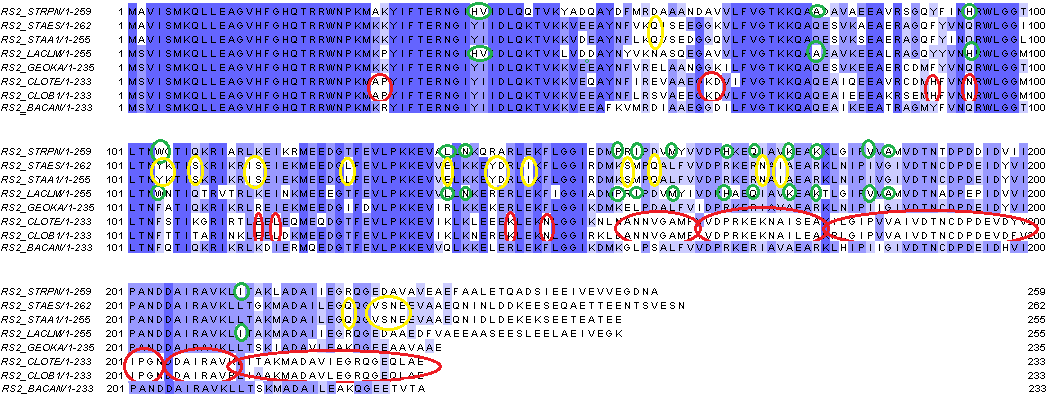
Рисунок 2. Выравнивание последовательностей белков семейства "Рибосомный белок S2", принадлежащих указанным выше бактериям. Цветами отмечены возможные диагностические позиции выравнивания. Пояснения в тексте.
По данному выравниванию с использованием различных алгоритмов было построено 4 филогенетических дерева. Их изображения представлены ниже.
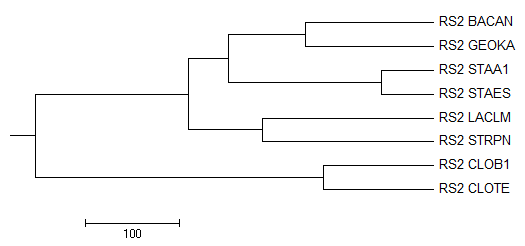
Рисунок 3. Филогенетическое дерево построенное алгоритмом Average distance using % identity.
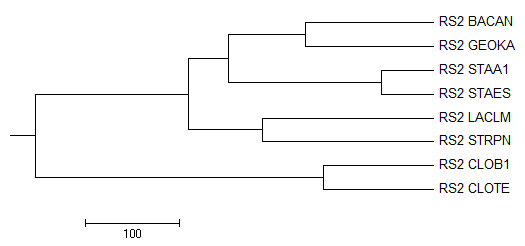
Рисунок 4. Филогенетическое дерево построенное алгоритмом Average distance using blosum62.
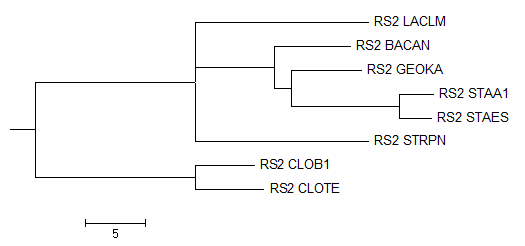
Рисунок 5. Филогенетическое дерево построенное алгоритмом Neighbour joining tree using % identity.
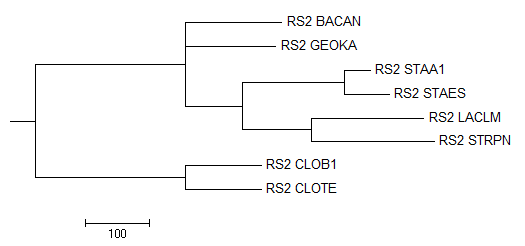
Рисунок 6. Филогенетическое дерево построенное алгоритмом Neighbour joining tree using blosum62.
Деревья полученные с помощью "Average distance" совпадают с правильным деревом.
В дереве, постороенном с помощью "Neighbour joining tree using % identity" правильно определены ветви классов Clostridia и Bacilli, однако в классе Bacilli есть несовпадения с правильным вариантом. Состав ветви Bacillales определен верно, однако члены семейства Bacillaceae не объединены. Ветвь Staphylococcaceae определена верно. Таким образом, в этом дереве отсутствуют ветви Lactobacillales и Bacillaceae и появилась ветвь {GEOKA, STAA1, STAES}.
В дереве, постороенном с помощью "Neighbour joining tree using blosum62" есть правильные ветви Clostridia и Bacilli, Lactobacillales и Staphylococcaceae. Однако ветвь Lactobacillales ошибочно отнесена к неправильной ветви {STAA1, STAES, LACLM, STRPN}, отсутствует ветвь Bacillales.
Таким образом, лучшме деревья получились с помощью алгоритмов "Average distance".
Далее с помощью программы Mega методом "Maximum Parsimony" был построен еще один вариант филогенетического дерева. Корень был определен вручную. В дереве, постороенном с помощью "Neighbour joining tree using % identity"
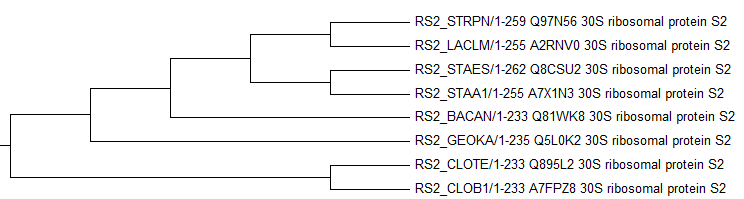
Рисунок 7. Филогенетическое дерево построенное с помощью программы Mega методом "Maximum Parsimony".
На этом дереве, как и в остальных случаях, правильно определены ветви классов Clostridia и Bacilli. Появилась ветвь {STAA1, STAES, LACLM, STRPN, BACAN} и {STAA1, STAES, LACLM, STRPN}. Отсутствуют ветви Bacillaceae и Bacillales. Это дерево больше всего похоже на построенное алгоритмом Neighbour joining tree using blosum62.