Построение дерева по нуклеотидным последовательностям
По инструкции, указанной в задании, были получены координаты генов РНК 16S рибосомной субъединицы в геноме изучаемых бактерий. Нужно отметить, что в геномах бактерий существует по несколько генов pРНК 16S. Гены для выравнивания были выбраны случайно. РНК 16S рибосомной субъединицы непроаннотирована для бактерии Streptococcus pneumoniae. Чтобы определить её координаты, было сделано выравнивание полного генома бактерии S.pneumoniae с геном РНК 16S рибосомной субъединицы бактерии Lactococcus lactis. Эта бактерия имеет наибольшее родство с S.pneumoniae среди изучаемых.
blastn -query laclm.fasta -subject strpn.fasta>align.txt
Был получен файл с тремя выравниваниями одинакового качества. Для определения координат предполагаемого гена РНК 16S было выбрано одно из них.
| Бактерия | Мнемоника | AC в EMBL | Координаты | Направление |
| Staphylococcus epidermidis | STAES | AE015929 | 1598006..1599559 | - |
| Staphylococcus aureus | STAA1 | AP009324 | 531922..533476 | + |
| Lactococcus lactis | LACLM | AM406671 | 511423..512971 | + |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421..11973 | + |
| Clostridium tetani | CLOTE | AE015927 | 8715..10223 | - |
| Clostridium botulinum | CLOB1 | CP000726 | 9282..10783 | + |
| Bacillus anthracis | BACAN | AE017334 | 9335..10841 | + |
| Streptococcus pneumoniae | STRPN | AE005672 | 15353..16895 | + |
Далее с помощью программы seqret были получены последовательности генов РНК 16S рибосомной субъединицы. Затем они были выровнены программой muscle. Выравнивание записано в файл al16s_sequences.fasta. Для построения дерева, показанного ниже на рисунке 1, использовался метод Maximum likelihood. Методы Neighbor-Joining, Minimum evolution привели к такому же результату. Это дерево не является правильным. Для сравнения на рисунке 2 указано правильное дерево. В полученном дереве нет ветви bacillaceae.
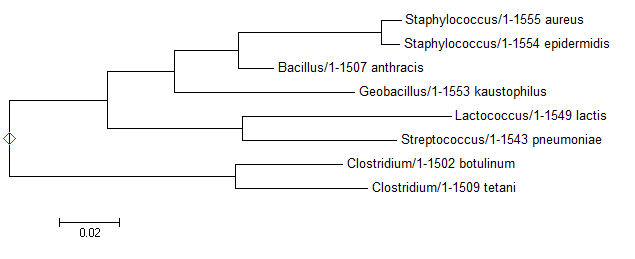
Рисунок 1. Филогенетическое дерево построенное алгоритмом Maximum likelihood по последовательностям генов РНК 16S рибосомной субъединицы.
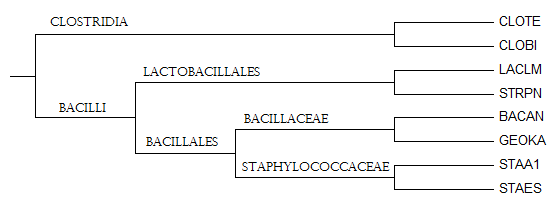
Рисунок 2. Филогенетическое дерево указанных бактерий.
Построение и анализ дерева, содержащего паралоги
Для построения дерева, содержащего паралоги, были найдены последовательности гомологов белка CLPX_BACSU среди изучаемых бактерий. Гомологи были найдены с помощью программы blastp с использованием файла с последовательностями белков бактерий в качестве базы данных.
blastp -task blastp -query clpx_bacsu.fasta -db proteo.fasta -evalue 0.001
Последовательности гомологов были определены в результате поиска по банкам данных, а также с использованием программы seqret. По полученным последовательностя алгоритмом Muscle было построено выравнивание. А по нему с поомощью программы Mega алгоритмом максимального правдоподобия было построено дерево, которое показано на рисунке 3. Файл с выравниванием, по которому было построено дерево, можно посмотреть по ссылке.

Рисунок 3. Дерево гомологов белка CLPX_BACSU среди изучаемых бактерий.
Гомологичные последовательности называют ортологами, если они произошли в результате видообразования. Паралоги - гомологи, которые произошли в результате дупликации. На данном дереве можно найти много примеров ортологов и паралогов. Вот некоторые из них.
Ортологи: CLPX_STAES и CLPX_LАCLM, CLPX_CLOTE и CLPX_CLOB1, Q899V4_CLOTE и Q97SK0_STRPN.
Паралоги: Q99XR9_STRP1 и J7MBF9_STRP1, Q899H3_CLOTE Q891B9_CLOTE. Интересная картина наблюдается для белков J7M6I1_STRP1, Q9A200_STRP1, FTSH_STRPN и B1SHF4_BACAN, C2NBK7_BACAN, B0AWL5_BACAN. Как видно на дереве, до образования этих шести белков, гомологи для CLOTE и CLOB1 шли парами, то есть до этого произошла дупликаци. Предположительно, перед разделением ветвей {J7M6I1_STRP1, Q9A200_STRP1, FTSH_STRPN} и {B1SHF4_BACAN, C2NBK7_BACAN, B0AWL5_BACAN} произошла еще одна дупликация. Белки J7M6I1_STRP1 и Q9A200_STRP1 произошли в результате неё. Они паралоги. Для BACAN из данного дерева нельзя определить какие белки появились раньше, а какие позже.