Совмещение структуры 3D30 и четырех гомологичнх структур
С помощью PDBeFold были найдены структуры, гомологичные 3D30, четыре из них (4js7, 1n10, 2hcz, 4jjo) были выбраны для пространственного совмещения. Выдача PDBeFold для этих структур доступна по ссылке.
Далее было сделано пространственное совмещение структур в Jmol. Результат показан на рисунке 1.
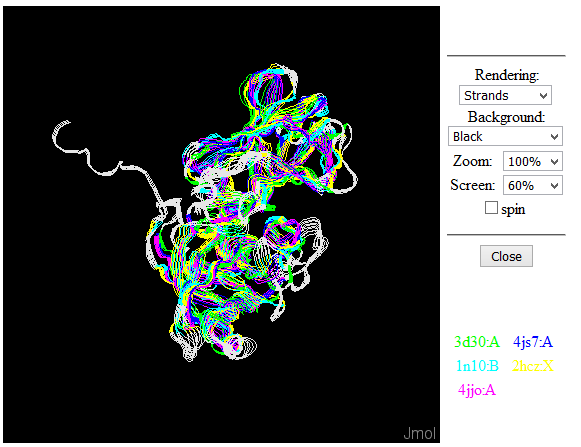
Рисунок 1. Пространственное совмещение структур 3d30 и 4js7, 1n10, 2hcz, 4jjo в Jmol.
Можно заметить, что структуры хорошо совмещены, хотя последовательности белков не идетничны (примерно 70-90% I).
Далее с помощью программы T-coffee было получено множественное выравнивание последовательностей указанных белков. Также было получено выравнивание последовательностей по структуре. Один их участок показан на рисунке 2. Сверху выравнивание T-coffee, а снизу по структуре. Проект JalView в выравниваниями можно получить по ссылке.

Рисунок 2. Участок выравнивания последовательностей структур 3d30 и 4js7, 1n10, 2hcz, 4jjo. Сверху выравнивание T-coffee, а снизу по структуре.
На рисунке 2 видно, что эти выравнивания местами не совпадают. Видно, что возникает различие в связи с двумя последовательностями, которые имеют вставку. Выравнивания различаются сдвигом других последовательностей относительно этой вставки, и сложно понять, какое из них правильное. К сожалению, несмотря на наличие подсказок, не получилось скачать файл Jmol (так, как указано в подсказках, сделать не получается), поэтому точно и красиво визуализировать участок не удалось. Однако этот участок в совмещении Jmol выглядит выровненным хорошо, может быть, что выравнивание по структурам лучше. Стоит также отметить, что и выравнивание без использовани структур выглядит неплохо. Оно "ровнее", то есть там меньше пропусков.
Поиск по сходству структур в PDBeFold
Для поиска была выбрана структура 1adj домен С, остатки 326-419. Всего 292 находки. В отсортированных по RMSD результатах такой домен не найден, вероятно потому что по умолчанию в параметрах PDBeFold необходимо совпадение последовательностей 70%, как от длины находки, так и от длины запроса. Указанный кусочек просто меньше 70% от домена.
Поиск по сходству структур в PDBeFold
В формате PDB были сохранены структуры константного домена T-клеточного рецептора из цепочки альфа (1kgc region d:118-206) и из бета (1mi5 region e:119-247). С помощью Sheep были построены карты бета-листов для этих структур. Карты, соответствующие указанным участкам показаны на рисунках 3 и 4. Видно, что карты sht_0, map_0 для 1kgc (альфа) и sht_0, map_0 для 1mi5 (бета) соответствуют друг другу. Консервативные остатки цистеина Cys139 для 1kgc и Cys148 для 1ьш5.
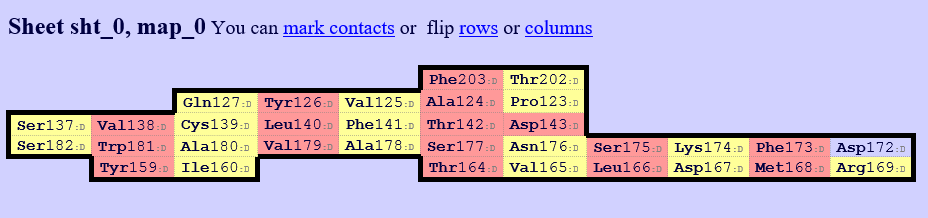
Рисунок 3. Карты бета-листов для структуры 1kgc region d:118-206.

Рисунок 4. Карты бета-листов для структуры 1mi5 region e:119-247.
Далее было сделано пространственное совмещение структур 1kgc (красный) и 1mi5 (розовый). Результат на рисунках 5 и 6.
PyMOL>select 1kgc, //pdb1kgc/D* and resi 118-206 PyMOL>select 1mi5, /pdb1mi5//E* and resi 119-247 PyMOL>select sht1kgc, 1kgc and name CA and resi 124-127+139-142+177-180 PyMOL>select sht1mi5, 1mi5 and name CA and resi 130-133+145-148+195-198 PyMOL>pair_fit sht1mi5, sht1kgc
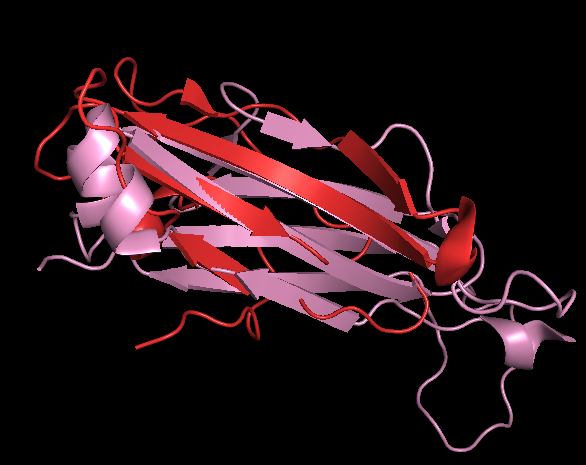
Рисунок 5. Пространственное совмещение структур 1kgc и 1mi5. Показаны только домен D структуры 1kgc (красный) и домен E структуры 1mi5 (розовый).
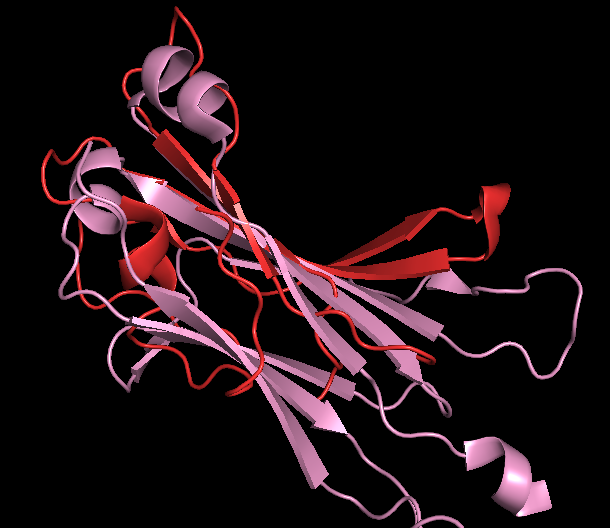
Рисунок 6. Пространственное совмещение структур 1kgc и 1mi5. Показаны только домен D структуры 1kgc (красный) и домен E структуры 1mi5 (розовый).
Как видно из рисунков 5 и 6 направление, положение тяжей похожи у двух доменов, кажется, что топология бета-листа совпадает. Однако есть сдвиги листов в пространстве (вдоль одной линии) и средний тяж короче у структуры 1kgc короче, чем у 1mi5.