Построение изображений для комплекса димера пуринового репрессора с ДНК 1JFT (PURINE REPRESSOR MUTANT-HYPOXANTHINE-PURF OPERATOR COMPLEX)
Выбранный димер показан на рисунке 1. С помощью PyMOL были посторены изображения различных поверхностей димера пуринового репрессора с ДНК 1JFT. Они показаны на рисунках 2 - 4.
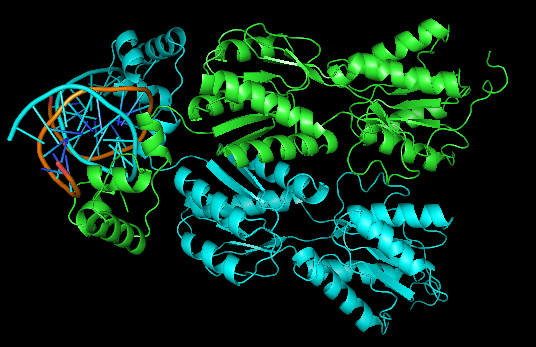
Рисунок 1. Комплекс димера пуринового репрессора с ДНК 1JFT. Мономеры показаны разными цветами
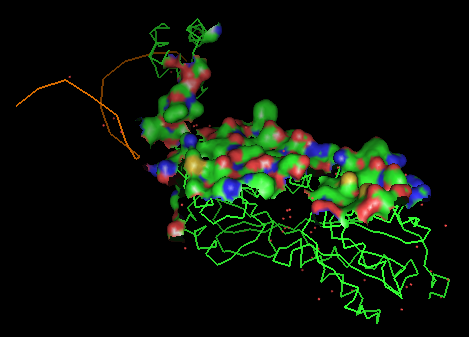
Рисунок 2. Поверхность контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера. Второй мономер не показан (что и требуется, если я правильно поняла задание). Поверхность раскрашена в соотвествии с типом атомов.
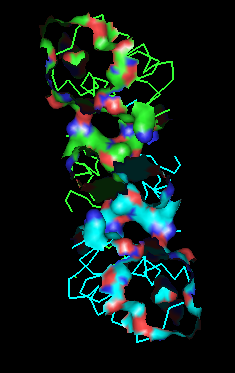
Рисунок 3. Поверхность контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт. Поверхность раскрашена в соотвествии с типом атомов.
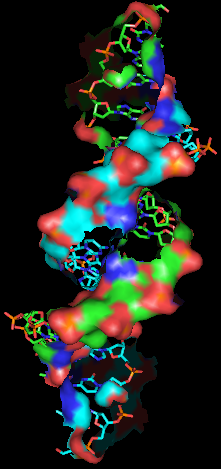
Рисунок 4. Поверхность контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали. Поверхность раскрашена в соотвествии с типом атомов.
С помощью сервиса CluD, были определены гидрофобные кластеры объёмом не менее 10 атомов на интерфейсе мономеров белка в том же комплексе (порог 4.4 Å). Были выбраны кластеры, которые находятся близко к участку взаимодействия мономеров. Они показаны на рисунке 5. Далее с помощью PyMOL было получено изображение поверхности контакта мономеров, на котором участки поверхности контакта, обращенные к выбранным кластерам показаны красным цветом (рисунок 6). Поверхность показана голубым. Сами атомы, входящие в кластеры также показаны на рисунке (синие шарики).
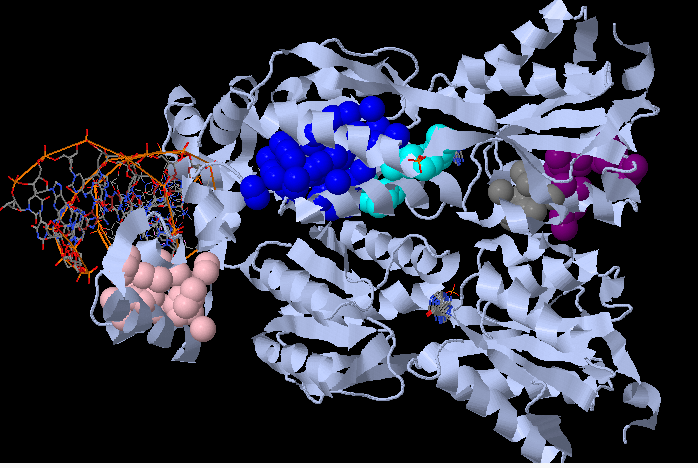
Рисунок 5. Комплекс димера пуринового репрессора с ДНК 1JFT. Гидрофобные кластеры, которые находятся близко к участку взаимодействия мономеров показаны шариками.
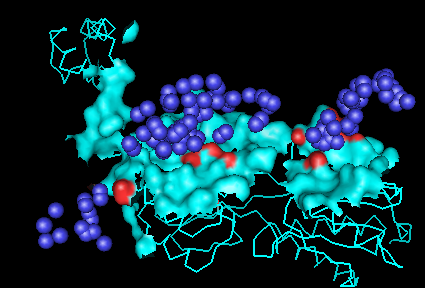
Рисунок 6. Поверхность контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера. Участки поверхности контакта, обращенные к выбранным кластерам показаны красным цветом, поверхность показана голубым (и сам мономер тоже). Атомы, входящие в кластеры показаны на рисунке (синие шарики).