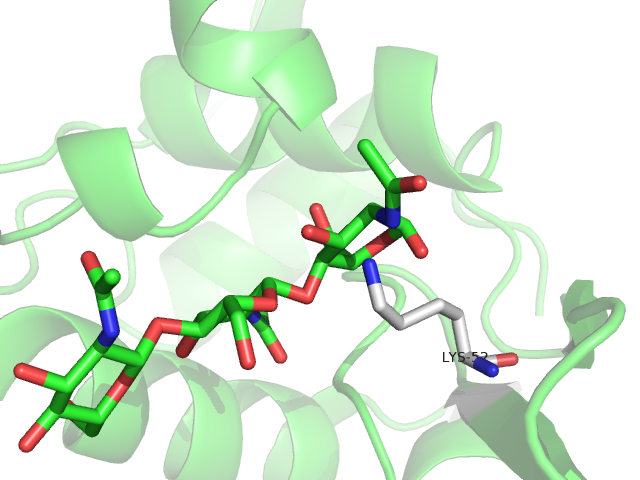
Внесение в структуру белка мутации, которая должна нарушить взаимодействие с лигандом.
Визуализация зоны контакта лиганда с белком была сделана с использованием iPython Notebook. К сожалению, дальше с ним возникли проблемы, и задание было выполнено локально. Asp52 был заменён на Lys52. Вероятно, что противоположный заряд и длинный радикал будут мешать взаимодействию лиганда с белком.
Далее был сделан анимационный ролик (mpeg) где происходит совмещение белков и показывается место мутации (скрипт). Видимо из-за близкого расположения атомов не удалось сохранить молекулу с мутацией в изначальном виде, как на первой картинке. PyMol упорно добавляет связи между мутантным остатком и лигандом.
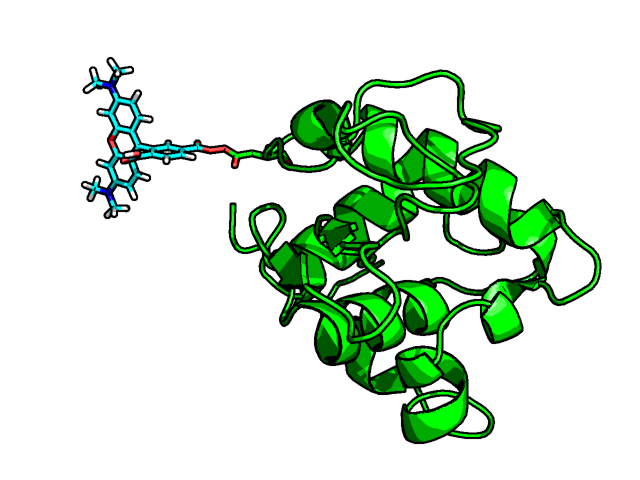
Приседенение флуорусцентной метки TAMRA к белку через сложноэфирную связь
Для присоединения метки был выбран Asp86, находящийся на поверхности структуры. Использовались команды fuse и torsion.

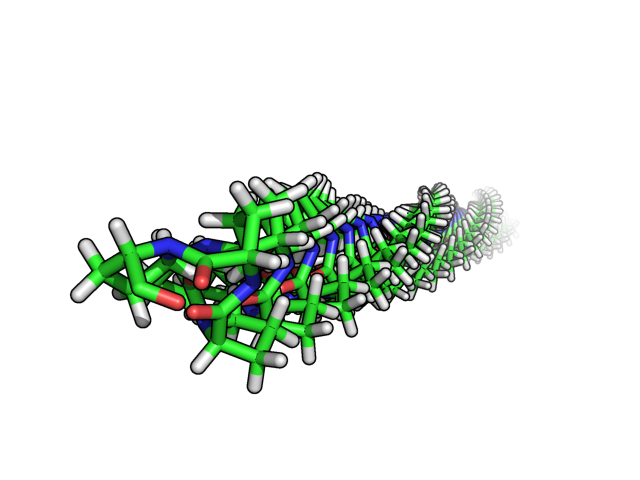
Построение поли-пролиновой спирали
Был написан скрипт, который был запущен через PyMol (File -> Run...)

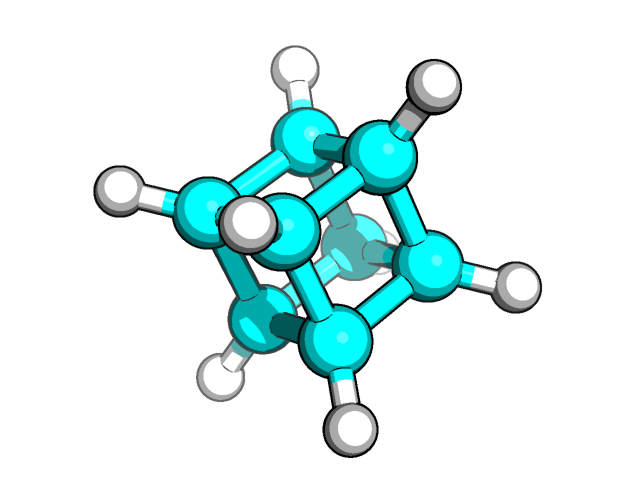
Построение третичной структуры вещества на основе SMILE нотации
На kodomo воспользовалась программой babel:
echo C12C3C4C1C5C2C3C45 > cubane.smi obgen cubane.smi > cubane.mol
Затем открыла cubane.mol через PyMol и получила картинку.