Значение кода заданного фермента
Найдем EC код белка ACON2_ECOLI и по нему определим функцию заданного фермента.Для этого обратимся к странице БД Uniprot с описанием ACON2_ECOLI. Итак, ЕС=4.2.1.3.
Изучим подробнее каждый пункт кода.
ЕС=4.2.1.3. Класс фермента имеет номер 4, что соответствует классу "лиазы" (Lyases). Ферменты этого класса катализируют реакции негидролитического отщепления и/или присоединения функциональной группы к субстрату. При этом может разрезаться С-С, C-N, C-O, C-S и другие связи.
EC=4.2.1.3. Подкласс фермента имеет номер 2 - углерод-кислородные лиазы (Carbon-Oxygen Lyases). Лиазы этого подкласса образуют и/или расщепляют С-О связь субстрата.
EC=4.2.1.3. Подподкласс фермента имеет номер 1 - гидролиазы (Hydro-Lyases). Ферменты этого подподкласса отщепляют и/или добавляют к субстрату воду.
EC=4.2.1.3. Порядковый номер фермента - 3 - аконитат-гидратаза (aconitate hydratase).
Реакция, катализируемая этим ферментом, выглядит так:
Reaction: citrate = isocitrate (1a) citrate = cis-aconitate + H2O (1b) cis-aconitate + H2O = isocitrateИли по-русски:
Реакция: цитрат = изоцитрат (1a) цитрат = цис-аконитат + H2O (1b) цис-аконитат + H2O = изоцитратКак видно из уравнения реакции, ACON2_ECOLI катализирует реакции взаимного превращения цитрата в изоцитрат путем расщепления С-О связи в одной части молекулы и дальнейшего образования ее в другой части. Графически реакция выглядит следующим образом:
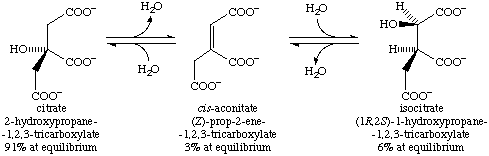
Промежуточное соединение реакции - цис-аконитат. В зависимости от того, к какому углероду, образующему двойную связь, присоединится гидроксильная группа, получится цитрат или изоцитрат. Механизм реакции выглядит так:
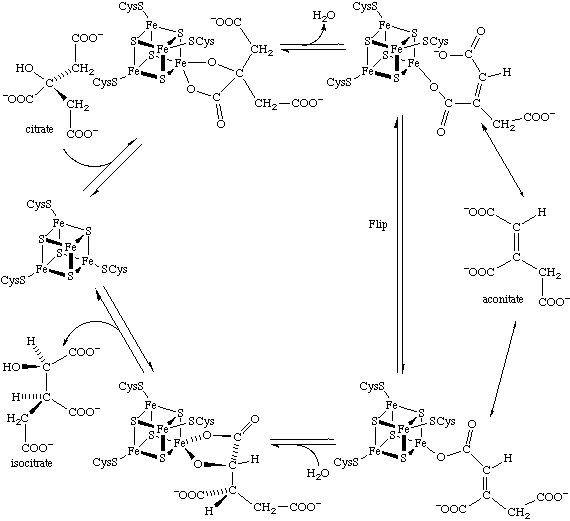
В катализе реакции основную роль выполняет Fe-S кластер, координирующий одну из двух карбоксильных групп и, в зависимости от этого, определяющий присоединение гидроксильной группы к одному из углеродов двойной связи (смена координируемой карбоксильной группы осуществляется "прыжком" (flip), изображенным в правой части механизма). Важно, что цис-аконитат остается связанным с ферментом все время, пока к нему вновь в то или иное место не присоединится гидроксильная группа.
Метаболические пути, в которых работает изучаемый фермент
В документе Uniprot с описанием белка ACON2_ECOLI (см. пункт 1) найдем имя локуса его гена. Имя локуса - b0118. Проведем поиск гена по названию локуса на главной странице БД KEGG. На выходе получаем документ с описанием гена и кодируемого им фермента ACON2_ECOLI. В частности, в нем описаны 3 метаболических путя, ассоциированных с изучаемым геном:- eco00020 - Цитратный цикл (цикл трикарбоновых кислот) (Citrate cycle (TCA cycle));
- eco00630 - Метаболизм глиоксилата и дикарбоксилата (Glyoxylate and dicarboxylate metabolism);
- eco01100 - Метаболические пути (Metabolic pathways).
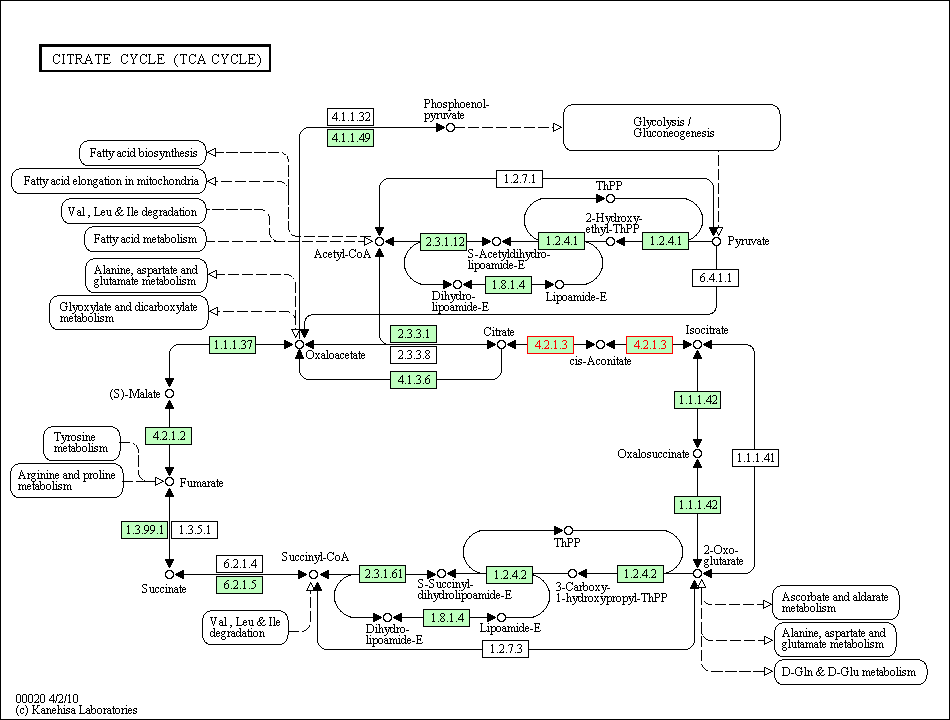
Работа фермента аконитазы (аконитат-гидратазы) выделена красной рамочкой. Почти все реакции в цикле обратимы. Более того, прямая и обратная реакция катализируются чаще всего одним и тем же ферментом. В их числе и реакция, катализируемая ферментом ACON2_ECOLI. В схеме видна связь между цитратным циклом и метаболизмом глиоксилата и дикарбоксилата (пунктирная стрелка в левой части схемы), где изучаемый фермент тоже принимает участие.
Структурные формулы заданных соединений в KEGG
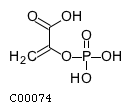
Изучим в KEGG структурные формулы фосфоенолпирувата и фенилаланина. Для этого откроем страницу оглавления KEGG. Перейдем оттуда к базе данных химических соединений KEGG LIGAND. Теперь проведем поиск по каждому из названий веществ.Фосфоенолпируват (Phosphoenolpyruvate, или Phosphoenolpyruvic acid, или PEP) имеет идентификатор C00074. Его стуктурная формула выглядит так:
Фенилаланин (Phenylalanine) имеет 3 записи в KEGG LIGAND:
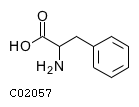
- Фенилаланин (Phenylalanine, или alpha-Amino-beta-phenylpropionic acid) имеет идентификатор C02057 и структурную формулу:
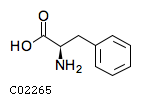
- D-Фенилаланин (D-Phenylalanine, или D-alpha-Amino-beta-phenylpropionic acid) имеет идентификатор C02265 и структурную формулу:
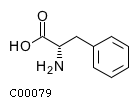
- L-Фенилаланин (L-Phenylalanine, или (S)-alpha-Amino-beta-phenylpropionic acid) имеет идентификатор C00079 и структурную формулу:
Метаболический путь от одного заданного соединения к другому
Обратимся к БД KEGG Pathway. Используем инструмент "Color Objects in KEGG Pathways". Путей, в которых участвуют и фосфоенолпируват, и L-фенилаланин, нашлось 5. Это:- Метаболические пути (Metabolic pathways) - ko01100;
- Биосинтез фенилпропаноидов (Biosynthesis of phenylpropanoids) - ko01061;
- Биосинтез алкалоидов, полученных из метаболического путя шикимата (Biosynthesis of alkaloids derived from shikimate pathway) - ko01063;
- Биосинтез фенилаланина, тирозина и триптофана (Phenylalanine, tyrosine and tryptophan biosynthesis) - ko00400;
- Биосинтез растительных гормонов (Biosynthesis of plant hormones) - ko01070.
К сожалению, на выбранной карте нет пути образования фосфоенолпирувата из L-фенилаланина, зато есть путь образования L-фенилаланина из фосфоенолпирувата. Я выбрал один из самых коротких возможных путей, состоящий всего из 10 реакций. Итак:
Выбранная цепочка ферментативных реакций:
путь: биосинтез фенилаланина, тирозина и триптофана;
цепочка: фосфоенолпируват → L-фенилаланин.
Карта с путем превращения фосфоенолпирувата в L-фенилаланин была сохранена в отдельном файле. Фосфоенолпируват на ней выделен красным, L-фенилаланин - зеленым, промежуточные соединения (7Ф-2-дегидро-3-дезокси-D-арабиногептонат, 3-дегидроквинат, 3-дегидрошикимат, шикимат, шикимат-3-фосфат, 5-О-(1-карбоксивинил)-3-фосфошикимат, хоризмат, префенат и фенилпируват) - желтым. Как видно из карты, 6 из 10 реакций превращения фосфоенолпирувата в L-фенилаланин - обратимые. Лишь превращение фосфоенолпирувата в 3-дегидроквинат (состоит из 2 необратимых реакций), превращение шикимата в шикимат-3-фосфат и превращение 5-O-(1-карбоксивинил)-3-фосфошикимата в хоризмат - необратимые реакции.
Сравнение метаболических путей у разных организмов
Определим, возможна ли выбранная в п. 4 цепочка ферментативных реакций превращения фосфоенолпирувата в L-фенилаланин у организмов, перечисленных ниже. Для этого переведем общую карту в режим, соответствующий выбранному организму. На основании результатов была заполнена таблица:| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | да | найдены гены ферментов, катализирующих все реакции выбранной цепочки |
| Archaeoglobus fulgidus | неизвестно | не найдены гены ферментов, катализирующих две реакции цепочки - первую (превращение фосфоенолпирувата в 7Ф-2-дегидро-3-дезокси-D-арабиногептонат; EC=2.5.1.54) и вторую (превращение 7Ф-2-дегидро-3-дезокси-D-арабиногептоната в 3-дегидроквинат; EC=4.2.3.4); этой архее необходимо синтезировать фенилаланин, поэтому либо эта цепочка реакций все же существует (просто не найдены гены соответствующих ферментов), либо существует другой путь синтеза (например найден ген фермента, катализирующего превращение 2-амино-3,7-дидезокси-D-трео-гепт-6-улосонической кислоты в 3-дегидроквинат; EC=1.4.1.-; см. карту) |
| Arabidopsis thaliana | да, скорее всего | не найден ген фермента, катализирующего всего одну реакцию из 10 (EC=4.2.1.51) - предпоследнюю реакцию цепочки - превращение префената в фенилпируват; но гены ферментов, катализирующих все другие реакции цепочки, найдены, поэтому, скорее всего, такая цепочка реакций в этом организме возможна (тем более этому организму - растению - необходимо синтезировать фенилаланин, поэтому должен быть путь его синтеза) |
| Homo sapiens | нет | найден ген фермента, катализирующего лишь одну (последнюю) реакцию цепочки - превращение фенилпирувата в фенилаланин (EC=2.6.1.1 и EC=2.6.1.5 |
Выбранная цепочка превращений состоит из немалого числа реакций, катализируемых многими ферментов. Поэтому неудивительно, что, например, в Arabidopsis thaliana не изучены ферменты, катализирующие все реакции цепочки. У человека выбранная цепочка вообще не протекает (известно, что фенилаланин является для человека незаменимой аминокислотой).
Сравнение ферментов у далеких организмов
Задача - сравнить ферменты с EC=2.1.2.1 у далеких организмов. Для начала изучим EC код фермента:EC=2.1.2.1:
ЕС=2.1.2.1. Класс фермента имеет номер 2, что соответствует классу "трансферазы" (Transferases). Ферменты этого класса катализируют реакции переноса функциональной группы с одного субстрата на другой. Могут переноситься метильные, ацильные, амино-, фосфатные группы и др.
EC=2.1.2.1. Подкласс фермента имеет номер 1 - трансферазы, переносящие одно-углеродные группы (Transferring one-carbon groups).
EC=2.1.2.1. Подподкласс фермента имеет номер 2 - гидроксиметил-, формил- и родственные трансферазы (Hydroxymethyl-, Formyl- and Related Transferases).
EC=2.1.2.1. Порядковый номер фермента - 1 - глицин-гидроксиметилтрансфераза (glycine hydroxymethyltransferase).
Реакция, катализируемая этим ферментом, выглядит так:
Reaction: 5,10-methylenetetrahydrofolate + glycine + H2O = tetrahydrofolate + L-serineИли по-русски:
Реакция: 5,10-метилентетрагидрофолат + глицин + Н2О = тетрагидрофолат + L-серин
-
Для начала найдем ферменты с EC=2.1.2.1 у человека и археи Archaeoglobus fulgidus, пользуясь БД UniProt.
Для этого воспользуемся SRS. Опцию маски снимем, чтобы при поиске EC белка находились лишь ферменты с EC=2.1.2.1, и не находились ферменты с EC=2.1.2.10 или 2.1.2.13 и т.д. Для упрощения запроса воспользуемся тем, что для белков приняты короткие имена. ID белков человека заканчиваются на _HUMAN, белки археи Archaeoglobus fulgidus - на _ARCFU. В результате, запрос для поиска выглядит так:([uniprot-ECNumber:2.1.2.1] & ([uniprot-ID:*_HUMAN] | [uniprot-ID:*_ARCFU]))
Всего было получено 18 последовательностей с таким EC кодом у человека и одна последовательность с таким кодом у археи Archaeoglobus fulgidus. Для отсева идентичных последовательностей воспользуемся ссылкой на UNIREF100. После этого из 18 последовательностей, найденных в человеке, неидентичных остается 17. На самом деле, среди этих 17 последовательностей у человека действительно достоверных только две (GLYC_HUMAN и GLYM_HUMAN), причем первый фермент (GLYC_HUMAN) работает в цитоплазме, а второй (GLYM_HUMAN) - в митохондриях. Остальные последовательности являются либо частями этих последовательностей, либо отличаются от них всего на несколько букв. Впрочем, еще одна последовательность у человека сильно отличалась от упомянутых двух последовательностей, об этом см. следующий пункт.
Аналогичный запрос по БД SwissProt в качестве результата выдал один белок у Archaeoglobus fulgidus (GLYA_ARCFU) и два белка у человека (GLYC_HUMAN и GLYM_HUMAN). Запрос по SwissProt выглядел так:(([swissprot-ID:*_HUMAN] | [swissprot-ID:*_ARCFU]) & [swissprot-ECNumber:2.1.2.1])
- Сравним доменную организацию (по PFAM) найденных белков.
Для этого воспользуемся режимом SW_InterProMatches для просмотра находок. Все последовательности, найденные SwissProtом, имеют абсолютно идентичную доменную организацию (и не только согласно PFAM). Единственный домен в них, согласно PFAM, имеет AC PF00464 и ID SHMT (Serine hydroxymethyltransferase - серин-гидроксиметилтрансфераза - синоним глицин-гидроксиметилтрансферазы). Для примера доменная структура белка GLYC_HUMAN представлена ниже:
Все последовательности, найденные UniProtом, кроме одной, имеют такую же доменную организацию. Исключение составляет белок B4DJ63_HUMAN. Согласно PFAM, он состоит из двух доменов SHMT, один из которых расположен на N-конце, а второй ближе к C-концу последовательности:
Однако больший интерес представляет доменная структура белков согласно БД Gene3D. Согласно Gene3D, все последовательности, кроме B4DJ63_HUMAN, содержат один субдомен Pyridoxal phosphate-dependent transferase, major region, subdomain 1, в то время как B4DJ63_HUMAN содержит и этот субдомен (он соответствует "левому" домену согласно БД PFAM) и субдомен Pyridoxal phosphate-dependent transferase, major region, subdomain 2 (соответствующий "правому" домену в доменной организации белка согласно БД PFAM).
Это удивило меня. Я изучил функции белка B4DJ63_HUMAN по ассоциированным с ним терминам GO. Не обнаружилось никаких новых функций, отличных от функций белков GLYC_HUMAN и GLYM_HUMAN. Тогда я подробнее изучил описание субдоменов 1 и 2. Оказалось, что субдомен 1, встречающийся чаще субдомена 2, имеет трехслойную альфа/бета/альфа сэндвичевую топологию. Про субдомен 2 сказано лишь, что он имеет сложную альфа/бета структуру. Тогда я предположил, что белки GLYC_HUMAN и GLYM_HUMAN тоже имеют последовательность субдомена 2, расположенную вплотную к субдомену 1, из-за чего БД Gene3D распознает только субдомен 1 (ведь он состоит из повторяющихся альфа/бета/альфа слоев и вполне соответствует описанию "сложной альфа/бета структуры" субдомена 2). А в белке B4DJ63 субдомены 1 и 2 разделены последовательностью между ними, из-за чего БД Gene3D распознает субдомены отдельно друг от друга. Для проверки этой гипотезы я получил последовательности трех белков (GLYC_HUMAN, GLYM_HUMAN и B4DJ63_HUMAN (к сожалению, не было термина, ассоциированного с B4DJ63_HUMAN, из онтологии Cellular Component, поэтому я не знал, где он работает - в цитоплазме или митохондрии)) и провел глобальное выравнивание белка B4DJ63_HUMAN c каждым из двух других белков программой needle. В результате глобальное выравнивание белка B4DJ63_HUMAN с белком GLYM_HUMAN подтвердило выдвинутую гипотезу. Выравнивание выглядит так:B4DJ63_HUMAN 0 -------------------------------------------------- 0 GLYM_HUMAN 1 MLYFSLFWAARPLQRCGQLVRMAIRAQHSNAAQTQTGEANRGWTGQESLS 50 B4DJ63_HUMAN 0 -------------------------------------------------- 0 GLYM_HUMAN 51 DSDPEMWELLQREKDRQCRGLELIASENFCSRAALEALGSCLNNKYSEGY 100 B4DJ63_HUMAN 0 -------------------------------------------------- 0 GLYM_HUMAN 101 PGKRYYGGAEVVDEIELLCQRRALEAFDLDPAQWGVNVQPYSGSPANLAV 150 B4DJ63_HUMAN 1 -----------MGLDLPDGGHLTHGYMSDVKRISATSIFFESMPYKLNPK 39 ||||||||||||||||||||||||||||||||||||||| GLYM_HUMAN 151 YTALLQPHDRIMGLDLPDGGHLTHGYMSDVKRISATSIFFESMPYKLNPK 200 B4DJ63_HUMAN 40 TGLIDYNQLALTARLFRPRLIIAGTSAYARLIDYARMREVCDEVKAHLLA 89 |||||||||||||||||||||||||||||||||||||||||||||||||| GLYM_HUMAN 201 TGLIDYNQLALTARLFRPRLIIAGTSAYARLIDYARMREVCDEVKAHLLA 250 B4DJ63_HUMAN 90 DMAHISGLVAAKVIPSPFKHADIVTTTTHKTLRGARSGSLRSGLAFPCLQ 139 ||||||||||||||||||||||||||||||||||| GLYM_HUMAN 251 DMAHISGLVAAKVIPSPFKHADIVTTTTHKTLRGA--------------- 285 B4DJ63_HUMAN 140 AYSWGTVGLDLRGIHSHLSHRSGLIFYRKGVKAVDPKTGREIPYTFEDRI 189 |||||||||||||||||||||||||||||| GLYM_HUMAN 286 --------------------RSGLIFYRKGVKAVDPKTGREIPYTFEDRI 315 B4DJ63_HUMAN 190 NFAVFPSLQGGPHNHAIAAVAVALKQACTPMFREYSLQVLKNARAMADAL 239 |||||||||||||||||||||||||||||||||||||||||||||||||| GLYM_HUMAN 316 NFAVFPSLQGGPHNHAIAAVAVALKQACTPMFREYSLQVLKNARAMADAL 365 B4DJ63_HUMAN 240 LERGYSLVSGGTDNHLVLVDLRPKGLDGARAERVLELVSITANKNTCPGD 289 |||||||||||||||||||||||||||||||||||||||||||||||||| GLYM_HUMAN 366 LERGYSLVSGGTDNHLVLVDLRPKGLDGARAERVLELVSITANKNTCPGD 415 B4DJ63_HUMAN 290 RSAITPGGLRLGAPALTSRQFREDDFRRVVDFIDEGVNIGLEVKSKTAKL 339 |||||||||||||||||||||||||||||||||||||||||||||||||| GLYM_HUMAN 416 RSAITPGGLRLGAPALTSRQFREDDFRRVVDFIDEGVNIGLEVKSKTAKL 465 B4DJ63_HUMAN 340 QDFKSFLLKDSETSQRLANLRQRVEQFARAFPMPGFDEH 378 ||||||||||||||||||||||||||||||||||||||| GLYM_HUMAN 466 QDFKSFLLKDSETSQRLANLRQRVEQFARAFPMPGFDEH 504Очевидно, что последовательности отличаются только тем, что N-конец белка B4DJ63_HUMAN "обрезан" по сравнению с белком GLYM_HUMAN, и в середине последовательности белка B4DJ63_HUMAN имеется участок, отсутствующий у белка GLYM_HUMAN. Существование белка B4DJ63_HUMAN доказано только существованием транскрипта, поэтому вполне возможно, что "обрезанный" N-конец - результат оплошности при выделении транскрипта или секвенировании, а внутренняя последовательность, разделяющая субдомены, - интрон. Если же белок и вправду существует в таком виде, в каком он дан в БД UniProt, то внутренняя последовательность - скорее всего, результат дупликации.
Для дальнейшей работы выберем у человека белок GLYC_HUMAN (он работает в цитоплазме, интересно будет сравнить его с белком, выполняющим ту же функцию в цитоплазме археи Archaeoglobus fulgidus). -
Определим % совпадения последовательностей гомологичных доменов из археи и человека.
У человека для выравнивания выберем домен белка GLYC_HUMAN (длины доменов белка GLYM_HUMAN и GLYC_HUMAN одинаковы, согласно данным БД PFAM, и составляют 400 а/к; длина домена в белке GLYA_ARCFU составляет 375 а/к). Вырежем из последовательностей белка последовательности доменов и сохраним их в отдельных файлах в fasta-формате с помощью программы seqret. Домен белка GLYC_HUMAN человека был сохранен в файле glycdom.fasta, а домен белка GLYA_ARCFU археи - в файле glyadom.fasta. Глобальное выравнивание проводилось программой needle. Результат сохранен в файле dom.needle.
Как видно, последовательности гомологичных доменов человека и археи достаточно похожи друг на друга, особенно если принимать во внимание огромное эволюционное расстояние между ними. Процент совпадения равен 32,6%, процент сходства - 50,5%. Последовательности совпадают примерно на треть, похожи на половину. Домены достаточно близки друг другу, такой процент совпадения гарантирует сходство 3D-структур доменов. Впрочем, домены похожи не настолько, чтобы гарантировать выполнение одинаковых функций (и одинаковые EC номера), однако в нашем случае это, прежде всего, следствие огромного эволюционного расстояния, пройденного от археи до человека (ведь мы знаем, что домены изучаемых белков имеют одинаковые EC номера и выполняют одинаковые функции). -
С помощью инструментов KEGG найдем для человеческого белка GLYC_HUMAN лучшего ортолога из архей, а для архейного GLYA_ARCFU - лучшего ортолога у эукариот.
Имя гена GLYC_HUMAN - SHMT1, имя гена GLYA_ARCFU - glyA. Найдем в БД KEGG описания генов белков GLYC_HUMAN и GLYA_ARCFU, после чего щелкнем по кнопке "Ortholog" и выберем в открывшемся окне опцию Best-best (bidirectional best hit), укажем нужную группу организмов (Archaea и Eukaryotes) и щелкнем по кнопке "GO".
Для человеческого белка GLYC_HUMAN лучшим ортологом среди архей оказался белок с именем гена Mthe_1241 из термофильной археи Methanosaeta thermophila. Этот белок имеет такой же EC номер и похожее название (глицин-гидроксиметилтрансфераза у археи, серин-гидроксиметилтрансфераза у человека - синонимы), участвует в тех же метаболических путях (кроме одного). Длина выравнивания последовательностей - 414 а/к, процент совпадения - 44,4%.
Для белка GLYA_ARCFU лучшим ортологом среди эукариот оказался белок с именем гена IscW_ISCW016048 из черноногого клеща Ixodes scapularis (black-legged tick). Белок имеет такой же EC номер. Длина выравнивания - 397 а/к, процент совпадения равен 38,9%.