Мутагенез в белке записи 1LMP
Средствами Tc1/Tk интерфейса (Wizard->Mutagenesis) проведем мутацию в белке записи 1LMP, которая должна привести к потере связывания с лигандом. Для этого вначале найдем те аминокислотные остатки, которые предположительно связывают лиганд. Определим множество атомов цепи белка, близко расположенных к лиганду, так:select bind, (not het) and (((het and (not resn hoh)) around 5.5) show surface, bindВ результате получаем изображение поверхности контакта полипептидной цепи с лигандом:
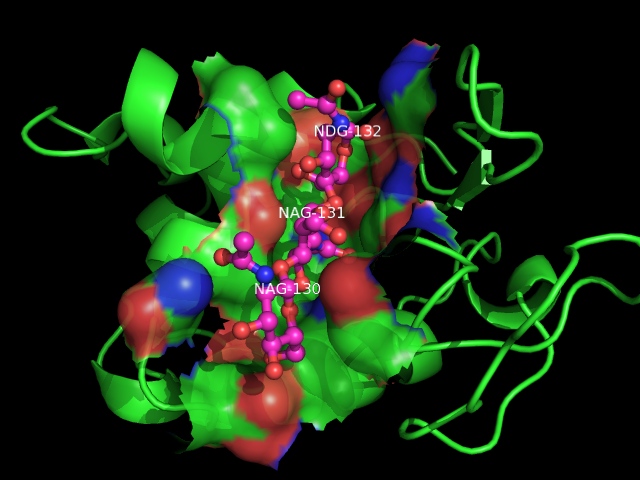
Проведем мутацию в аминокислоте, образующей водородную связь с лигандом. Причем, для большей точности, возьмем остаток, прилегающий ближе всего к лиганду. В качестве такого аминокислотного остатка возьмем аспарагиновую кислоту ASP-52. Заменим ее на гидрофобную аминокислоту, например, на валин:
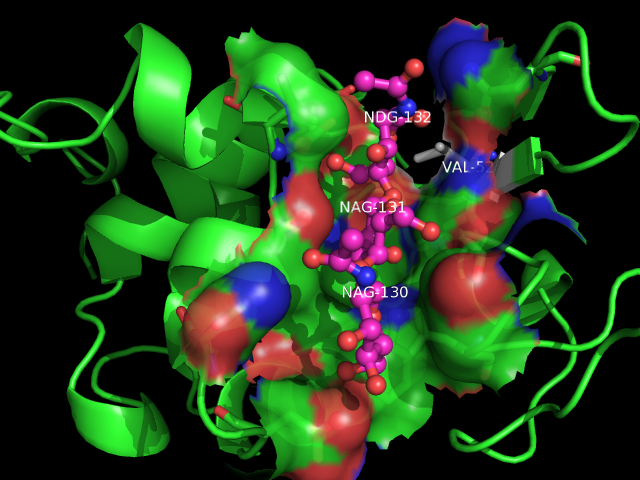
Сохраним измененную структуру в файле 1lmp_mut.pdb.
Создание анимационного ролика (mpeg)
Используя команды mset, mview, super, translate создадим анимационный ролик (mpeg), где происходит совмещение белков и показывается место мутации. Ролик был сохранен в файле mutation.mpgПостроение поли-аланиновой последовательности в форме валентинки
Создадим поли-аланиновую последовательность в форме валентинки. Для изменения формы будем использовать режим Editing и манипулирование торсионными углами. Ctrl+RightClick - выбор связи, Ctrl+LeftClick - вращение. Изображение последовательности представлено ниже:
Написание скрипта для построения поли-аланиновой альфа-спирали длиной 100 аминокислот
Скрипт был сохранен в файле alahelix.pml.