make_ndx -f box_38.gro -o 1.ndxТеперь создадим gro файл с одной молекулой и зададим ячейку. При запуске editconf выберем номер, соответствующий группе из одной молекулы:
editconf -f box_38.gro -o et1.gro -n 1.ndx #зададим ячейку и расположим молекулу по центру ячейку editconf -f et1.gro -o et.gro -d 2 -cВ результате получаем файл et.gro. Исправим файл топологии et.top из прошлого задания (изменим в разделе [molecules] количество молекул этана с 38 на 1) и сохраним топологию в файле et1.top.
be.mdp - метод Берендсена для контроля температуры.
vr.mdp - метод "Velocity rescale" для контроля температуры.
nh.mdp - метод Нуза-Хувера для контроля температуры.
an.mdp - метод Андерсена для контроля температуры.
sd.mdp - метод стохастической молекулярной динамики.
Скрипт для работы был сохранен в файле tempdin.bash.
grompp -f ${i}.mdp -c et.gro -p et1.top -o et_${i}.tpr
# где i: be,vr,nh,an,sd список mdp файлов
Задавать i вне скрипта будем командой export i="be".
mdrun -deffnm et_${i} -v -nt 1
trjconv -f et_${i}.trr -s et_${i}.tpr -o et_${i}.pdb
Среди всех систем очень выделяется метод Андерсона. Молекула этана в практически неподвижна. Происходят лишь небольшие изменения в валентных углах и длине связи С-С.
Видимо, эта модель хорошо отражает систему, в которой этан прочно связан - например, в кристалле.
В методе стохастической молекулярной динамики молекула очень быстро перемещается в пространстве. Даже отдельные кадры не позволяют уловить постепенные изменения в углах или длинах связей.
Методы Берендсена и "Velocity rescale" похожи друг на друга. Вначале молекула расположена достаточно неподвижно, вращаются лишь атомы водорода (связанные с атомами углерода). Однако, через некоторое время молекула раскручивается, как юла, и начинает все быстрей вращаться и менять свое положение. Причем, в методе Берендсена это происходит гораздо быстрее, чем в методе "Velocity rescale".
В методе Нуза-Хувера атомы водорода так же вращаются вокруг углеродов, но при этом атомы углерода расположены достаточно неподвижно. Молекула не раскручиватся, как в случае методов Берендсена и "Velocity rescale".
g_energy -f et_${i}.edr -o et_${i}_en.xvg
Построим графики изменения энергий.
Для этого воспользуемся Gnuplot:
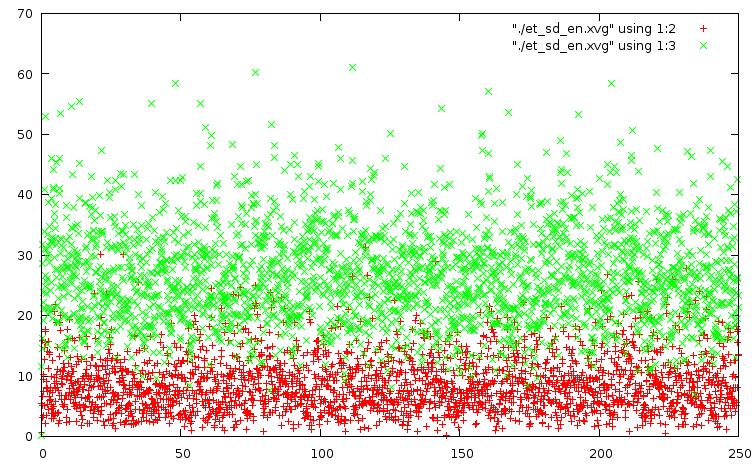
set datafile commentschars "#@&" plot "./et_be_en.xvg" using 1:2, "./et_be_en.xvg" using 1:3 .... plot "./et_sd_en.xvg" using 1:2, "./et_sd_en.xvg" using 1:3Полученные в результате графики для разных методов представлены ниже:
метод Берендсена:
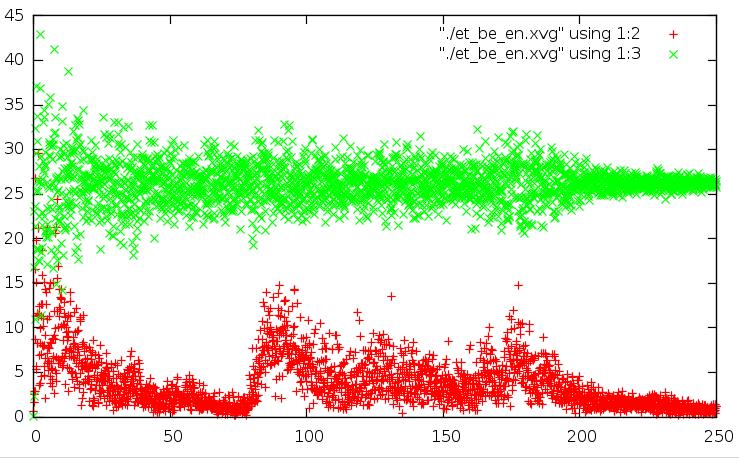
метод "Velocity rescale":
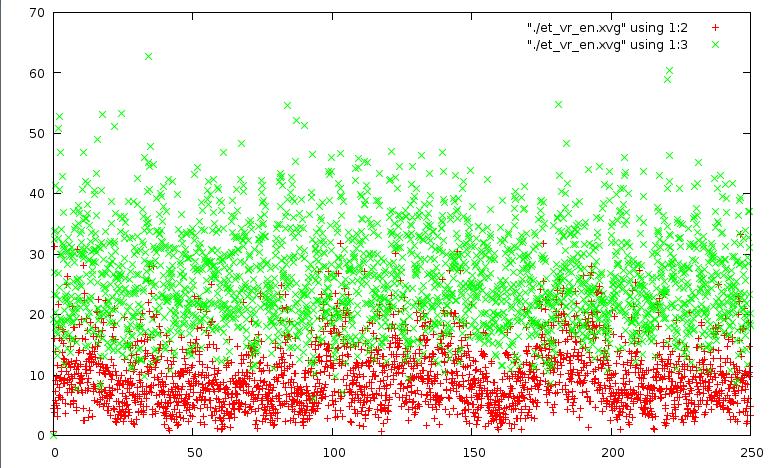
метод Нуза-Хувера:
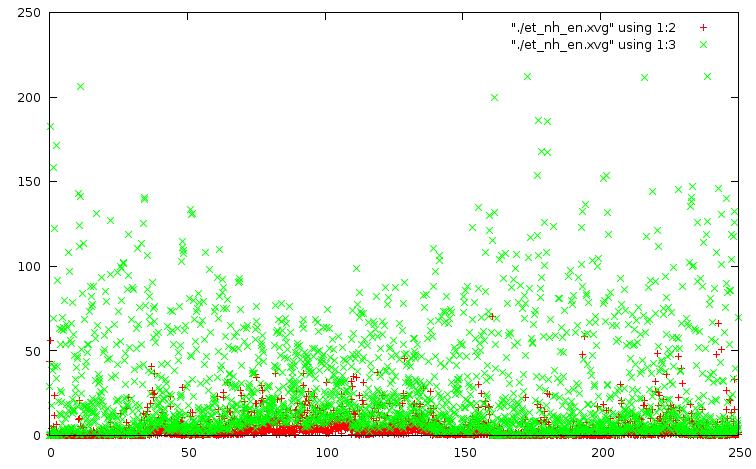
метод Андерсена:
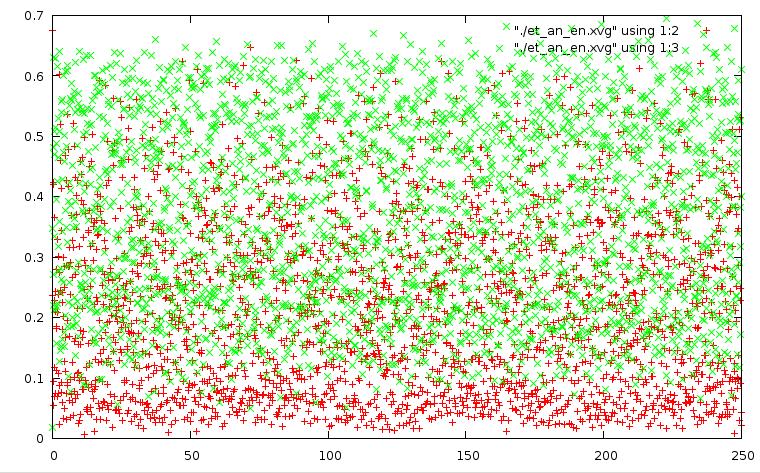
метод стохастической молекулярной динамики:
[ b ] 1 2Запустим утилиту по анализу связей g_bond:
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
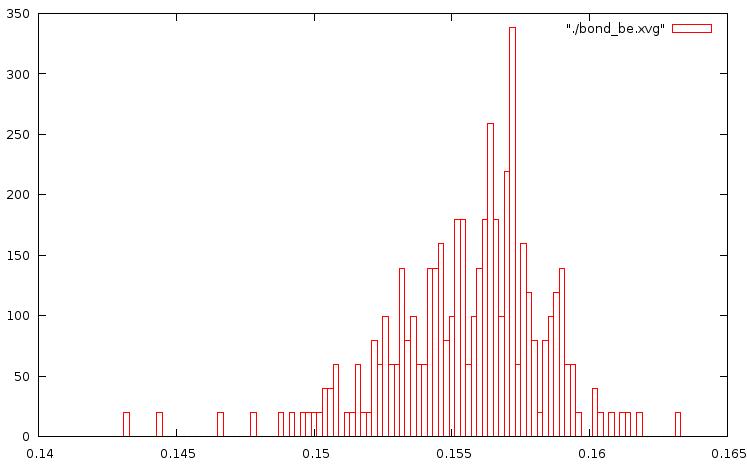
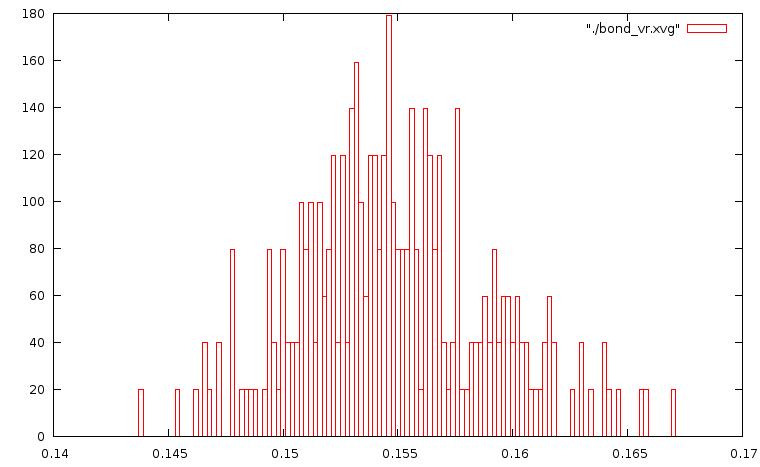
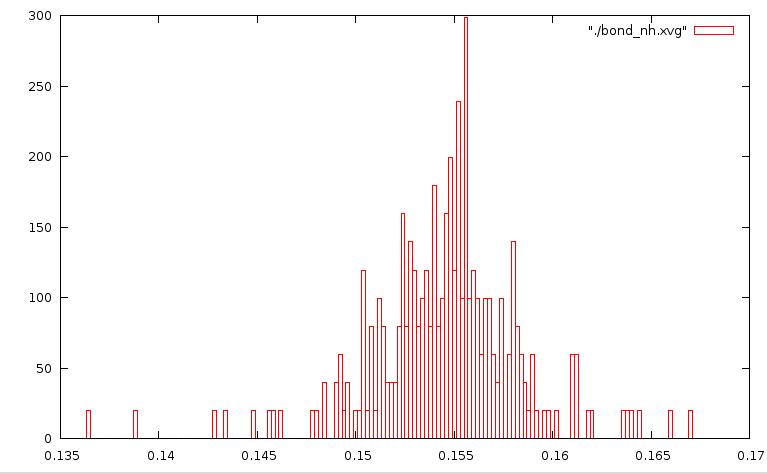
Построим графики распределения длин связей, используя boxes в Gnuplot:
plot "./bond_be.xvg" with boxes .... plot "./bond_sd.xvg" with boxesПолученные в результате графики для разных методов представлены ниже:
метод Берендсена:
метод "Velocity rescale":
метод Нуза-Хувера:
метод Андерсена:
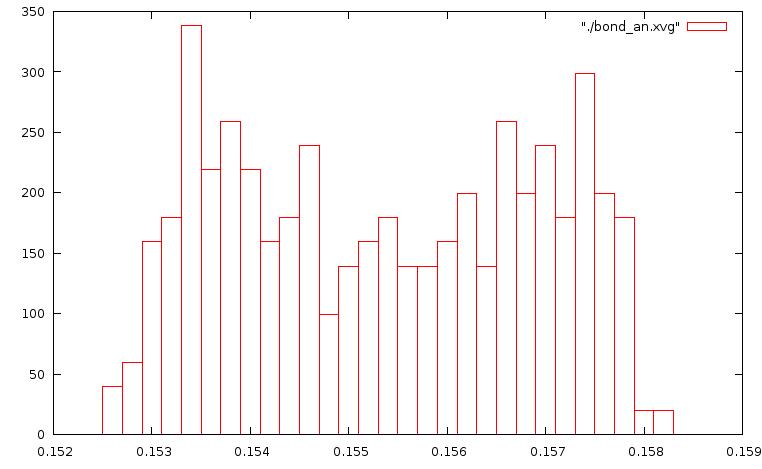
метод стохастической молекулярной динамики:
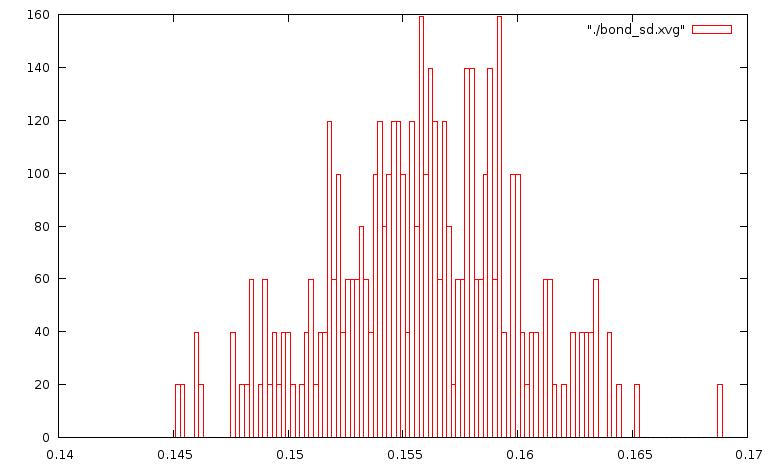
Такому распределению в достаточной мере соответствуют графики методов "Velocity rescale", Нуза-Хувера и стохастической молекулярной динамики.
В целом достаточно неплохо себя показали методы: "Velocity rescale" и Нуза-Хувера. Именно они, на мой взгляд, позволяют наиболее реалистично поддерживать температуру в системе.