trjconv -f b_md.xtc -s b_md.tpr -o b_pbc_1.pdb -skip 20В результате получаем файл b_pbc_1.pdb. Однако, при этой визуализации атомы, расположенные за стенкой ячейки, переносятся на другой край ячейки, в результате чего всю ячейку пересекают нереально длинные связи. Чтобы такого не было, будем переносить на другой край ячейки только молекулы целиком:
trjconv -f b_md.xtc -s b_md.tpr -o b_pbc_2.pdb -skip 20 -pbc molВ результате получаем файл b_pbc_2.pdb. В данном случае все выглядит вполне реалистично. Длинного бислоя не образуется, однако образуется достаточно вытянутая мицелла. Мицелла образуется примерно с 25 номера модели. Это соответствует времени 12500 фемтосекунд.
g_traj -f b_md.xtc -s b_md.tpr -ob box_1.xvgВ файле box_1.xvg содержатся размеры ячейки. В первой колонке приводится время, а в следующих трёх - размер ячейки (длины по каждым из осей). Зависимость площади (нормированной на один липид в слое) в квадратных нанометрах по соответствующим осям (вычисленной как произведение длин по осям, не являющимися нормалью к поверхности бислоя, (то есть по осям Y и Z) деленным на 32 (общее число молекул липидов пополам)) представлена на графике:
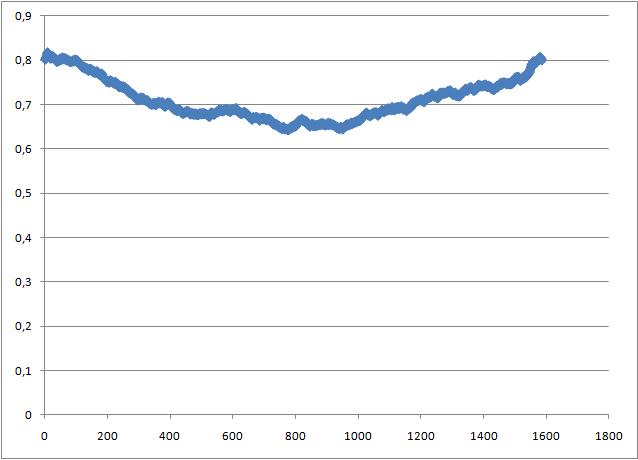
Зависимость была построена в Excel.
Таким образом, как видно, оптимальная площадь одного липида в мицелле равна около 0.8 квадратных нанометров. Как видно из графика, в середине моделирования молекулы липидов сгруппировались достаточно плотно (это соответствует примерно началу образования мицеллы). После этого мицелла стала вытягиваться и, соответственно, стала расти площадь, приходящаяся на каждую молекулу липида в отдельности.
g_sas -f b_md.xtc -s b_md.tpr -o sas_b.xvgВ результате получаем файл sas_b.xvg с данными о гидрофильной и гидрофобной поверхностях в каждый момент времени. Зависимость изменения гидрофобной (синяя) и гидрофильной (красная) поверхностей от времени представлена ниже:
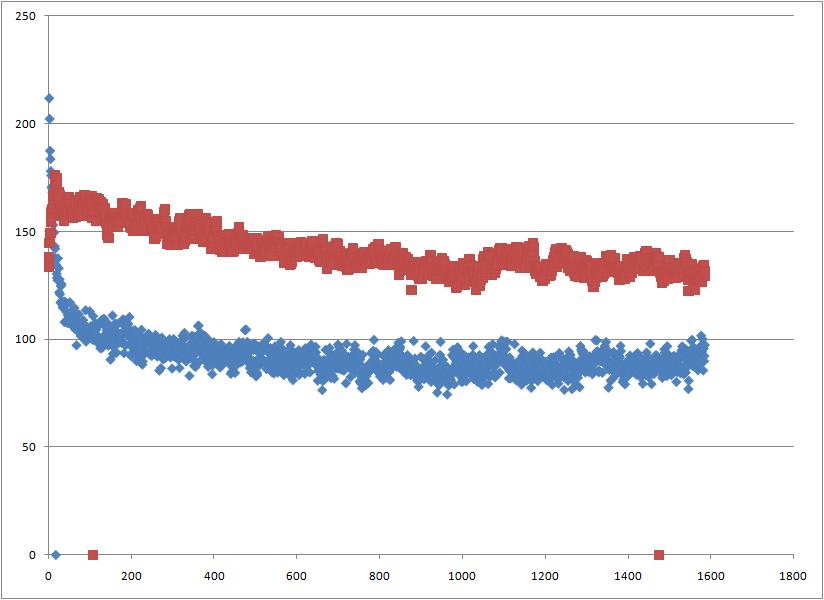
Таким образом, при образовании мицеллы (и бислоя) происходит уменьшение как гидрофобной, так и гидрофильной поверхностей. Такая компактизация приводит к уменьшения энергии системы в водном растворителе (что и является движущей силой образования мицеллы и бислоя).
g_order -s b_md -f b_md.xtc -o ord_end.xvg -n sn1.ndx -b 35000 -d XДля начала траектории:
g_order -s b_md -f b_md.xtc -o ord_start.xvg -n sn1.ndx -e 5000 -d XГрафик с изображением меры порядка для разных атомов липида (с головки до хвоста) для начала траектории представлен ниже:
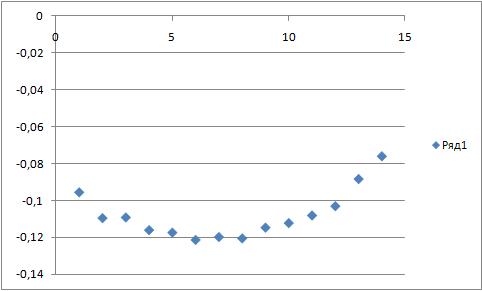
Такой же график для конца траектории:
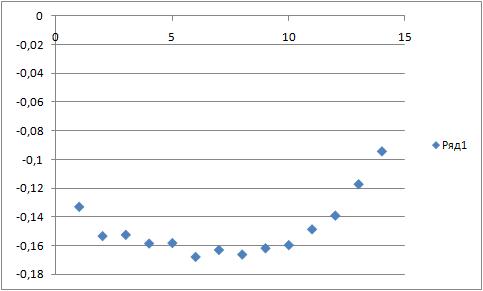
Как видно, графики имеют примерно одинаковый вид (но разные значения). Головки липидов подвижны, затем идет уплотнение (так как в этой части атомы образуют стерическую структуру мицеллы (или бислоя)), а затем - ближе к концу хвоста - атомы очень подвижны (во внутренней части мицеллы и бислоя хвосты могут достаточно свободно извиваться).
Естественно, в конце траектории, когда мицелла приобрела четко сформированную структуру, подвижность атомов молекул липидов падает, по сравнению с началом траектории, когда структуры еще не образовано (что и отражено на графиках).