sequence:ХХХХХ::::::: 0.00: 0.00Эта строчка описывает входные параметры последовательности для modeller. После имени последовательности белка-образца добавим строчку:
structureX:1lmp_now.ent:1 :A: 130 :A:undefined:undefined:-1.00:-1.00Эта строчка описывает, какой файл содержит структуру белка с этой последовательностью (1lmp_now.ent), номера первой и последней аминокислот в структуре, идентификатор цепи и т.д. В конце каждой последовательности добавим символы:
/.Этот символ означает конец цепи белка. Точка указывает на то, что имеется один лиганд. При этом оставляем строчку со звездочкой (*).
-
что нужно использовать стандартные валентные углы в полипептидной цепи (строчка 4);
что дополнительно нужно сохранять взаимное расположение определенных пар атомов (3.5 ангстрема) - в данном случае трех атомов белка, образующих водородные связи с тремя атомами лиганда - строчки 5-7 с ID пар атомов;
параметры взаимного расположения атомов пары описаны в строчке 9-10.
3 точки могут однозначно расположить сложную структуру в пространстве, поэтому мы выбираем водородные связи как источник данных точек. - что ковалентные связи в гетероатомах нужно вычислять по расстояниям между атомами, строчка 12;
- имя файла с выравниванием и имена последовательностей образца и моделируемого белка, строчка 13 (а имя файла со структурой содержится в выравнивании);
- число и номера моделей, которые нужно построить (в данном примере 5 моделей), строки 14-15;
- что пора строить модель строчка 16.
mod9v7 lysc_human.py &
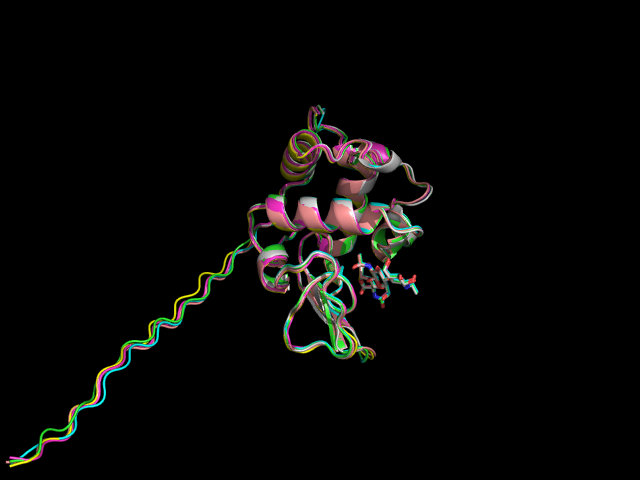
Как видно, структуры моделей практически не отличаются друг от друга (малейшие отличия наблюдаются лишь в петлях). N-конец всех моделей представляет собой длинный хвост. Возник он потому, что в структуре белка-образца не было соответствующих аминокислотных остатков. Следовательно, при моделировании по гомологии данный участок белка никак не структурировался (ведь в гомологе его не было). Более того, на самом деле в человеческом белке этот N-концевой участок впоследствии отщепляется (при созревании белка), поэтому особой значимости он не несет.
Оценим качество карты Рамачандрана (Ramachandran plot evaluation). Для 1 модели z-score равен 0.264, для 2 - 0.439, для 3 - 0.222, для 4 - 0.021, для 5 - 0.265. По этому показателю лучшая модель - модель №4. Впрочем, все модели показали хороший результат.
Оценим длины связей моделей (anomalous bond lengths). Для 1 модели z-score равен 0.951, для 2 - 0.948, для 3 - 0.955, для 4 - 0.948, для 5 - 0.947. По этому показателю лучшая модель - модель №5. Впрочем, z-score для всех моделей достаточно похожи.
Оценим валентные угла моделей (anomalous bond angles). Для 1 модели z-score равен 1.318 (29 углов с z-score больше 4), для 2 - 1.274 (26 углов с z-score больше 4), для 3 - 1.275 (26 углов с z-score больше 4), для 4 - 1.291 (29 углов с z-score больше 4), для 5 - 1.209 (18 углов с z-score больше 4). По этому показателю лучшая модель - модель №5.
Таким образом, из трех этих показателей в двух лучший результат показала модель №5. Однако, все модели очень похожи друг на друга и могут использоваться для дальнейшего анализа белка LYSC_HUMAN.