- SERYL-TRNA SYNTHETASE (Серил-тРНК-синтетаза), две цепи (A и B).
- TRNA(SER) (Сериновая тРНК), одна цепь (Т).
Эти молекулы были получены из организма THERMUS THERMOPHILUS.
Для исследования была выбрана цепь Т, представляющая сериновую тРНК со следующей последовательностью:
[1] 5' GGAGAGGUGCCCGAGUGGC H2U GAAGGGACACGACUGGAAAUCGUGUAGGGGGG
CUUAAACCUCCCUCGCGGG 5MU PSU CGAAUCCCGCCCUCUCCGCCA 3' [76],где 1 и 76 - номера первого и последнего нуклеотида.
Как видно, в последовательности содержатся три модифицированных нуклеотида:
- H2U - 5,6-дигидроуридин-5'-монофосфат;
- 5MU - 5-метилуридин-5'-монофосфат;
- PSU - псеводуридин-5'-монофосфат.
Для упрощения работы с тРНК был создан отдельный файл только с координатами нуклеиновой кислоты (без координат белка): 1ser_r.pdb.
акцепторный стебель состоит из участка 4-7 и комплементарного ему участка 66-69 (координаты других остатков, формирующих акцепторный стебель, в файле не приведены).
Т-стебель - из участка 49-53 и комплементарного ему 61-65;
� D-стебель - из участка 10-12 и комплементарного ему 23-25; забавно, что еще один нуклеотид, образующий обычно D-стебель (13-й), в моем случае образует водородную связь не с 22-м нуклеотидом (с которым обычно ее образует), а с 9-м, несмотря на то, что координаты 22-го нуклеотида в файле содержатся;
� антикодоновый стебель в файле не представлен вовсе (координаты соответствующих нуклеотидов вместе с антикодоновой петлей и антикодоном GGA отсутствуют). Имеются координаты лишь двух нуклеотидов, формирующих антикодоновый стебель (42-й и 43-й), комплементарные нуклеотиды которых (27-й и 28-й), к сожалению, не представлены тоже.
Вот фрагмент выходного файла, предоставивший эти данные:
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.014) T:...4_:[..G]G-----C[..C]:..69_:T (0.014) |
2 (0.012) T:...5_:[..A]A-----U[..U]:..68_:T (0.025) |
3 (0.011) T:...6_:[..G]G-----C[..C]:..67_:T (0.016) |
4 (0.027) T:...7_:[..G]Gx----C[..C]:..66_:T (0.011) |
5 (0.023) T:..49_:[..G]G-----C[..C]:..65_:T (0.014) |
6 (0.010) T:..50_:[..C]C-----G[..G]:..64_:T (0.012) |
7 (0.011) T:..51_:[..G]G-----C[..C]:..63_:T (0.010) |
8 (0.016) T:..52_:[..G]G-----C[..C]:..62_:T (0.013) |
9 (0.019) T:..53_:[..G]G----xC[..C]:..61_:T (0.014) |
10 (0.020) T:..54_:[5MU]u-**-xA[..A]:..58_:T (0.018) |
11 (0.036) T:..55_:[PSU]Px**+xG[..G]:..18_:T (0.020) x
12 (0.019) T:..10_:[..C]C-----G[..G]:..25_:T (0.014) |
13 (0.016) T:..11_:[..C]C-----G[..G]:..24_:T (0.014) |
14 (0.015) T:..12_:[..C]C----xG[..G]:..23_:T (0.015) |
15 (0.021) T:..13_:[..G]G-**--G[..G]:...9_:T (0.016) |
16 (0.021) T:..14_:[..A]A-**-xU[..U]:...8_:T (0.031) |
17 (0.026) T:..15_:[..G]Gx**+xC[..C]:..48_:T (0.021) x
18 (0.012) T:..19_:[..G]Gx---xC[..C]:..56_:T (0.014) +
19 (0.020) T:..45_:[..A]A-----U[..U]:..47Q:T (0.020) |
20 (0.013) T:..46_:[..G]G-----C[..C]:..47P:T (0.021) |
21 (0.016) T:..47_:[..G]G-----C[..C]:..47O:T (0.007) |
22 (0.009) T:..47A:[..G]G-----C[..C]:..47N:T (0.016) |
23 (0.012) T:..47B:[..G]G-*---U[..U]:..47M:T (0.017) |
24 (0.011) T:..47C:[..G]G-----C[..C]:..47L:T (0.014) |
25 (0.019) T:..47D:[..G]G-----C[..C]:..47K:T (0.013) |
Note: This structure contains 6[5] non-Watson-Crick base-pairs.
С помощью скрипта rna.spt, представленного ниже, в RasMol можно получить изображение остова изучаемой молекцлы тРНК, в которой акцепторный стебель выделен красным цветом, Т-стебель - зеленым, D-стебель - синим, антикодоновый стебель (точнее единственные представленные в файле 2 нуклеотида антикодонового стебля) - оранжевым. Кроме того, фиолетовым я выделил огромную вариабельную петлю (нуклеотиды №№45-47) (которая, на самом деле, должна быть еще больше, но в файле некоторые нуклеотиды, ее формирующие, увы, не представлены). А в шарнирной модели я представил остаток 5,6-дигидроуридина в D-петле (нуклеотид №20A). Тимидины Т-петли в этой молекуле, к сожалению, не представлены.
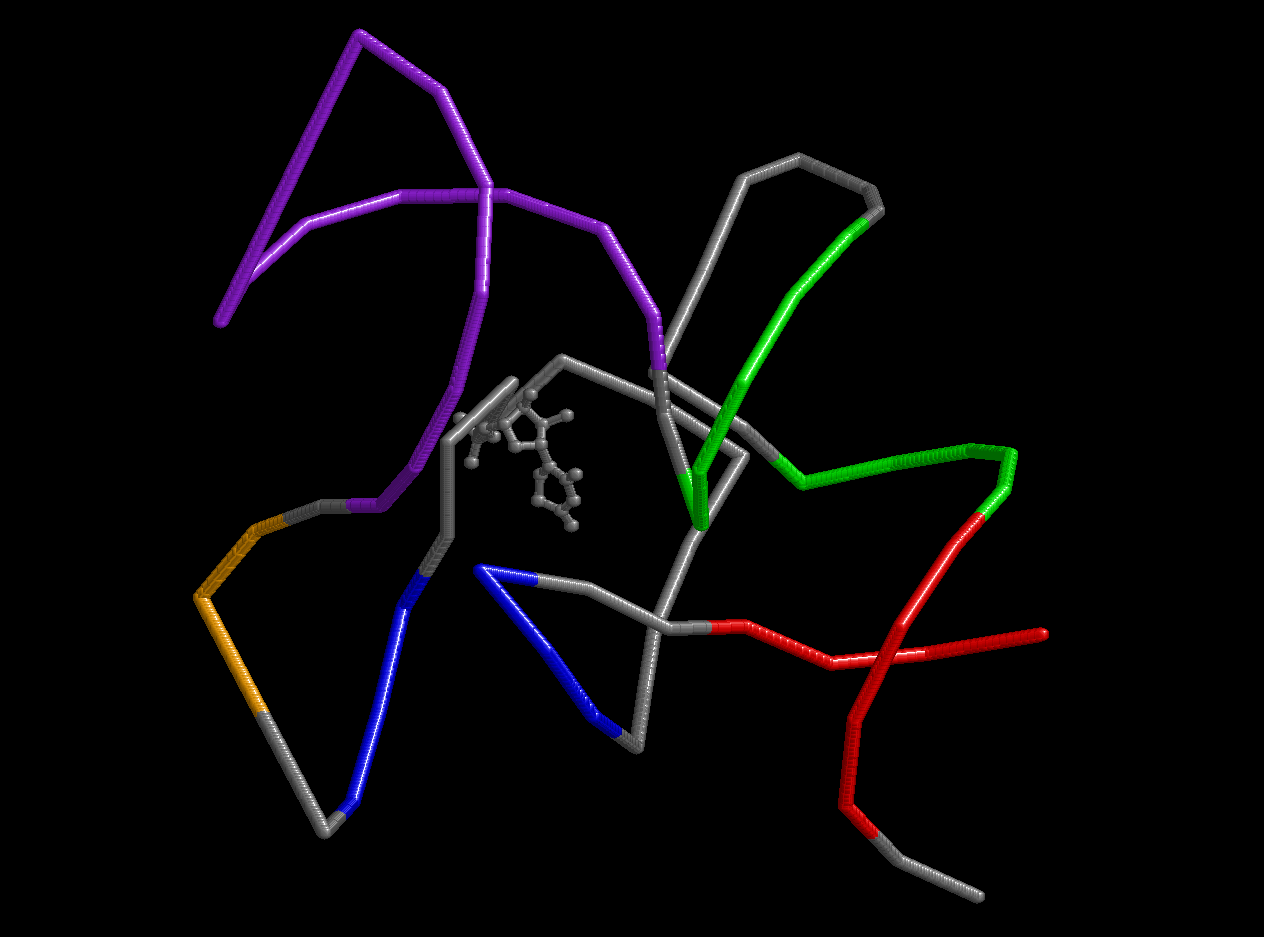
Рис.1. Вторичная структура сериновой тРНК из 1ser_r.pdb |
restrict none select all restrict nucleic backbone 100 color grey select 4-7:T or 66-69:T color red select 49-53:T or 61-65:T color green select 10-12:T or 23-25:T color blue select 42-43:T color orange select 45-47:T color purple select 20:T and (not G20) and (not C20) wireframe 100 cpk 50 |
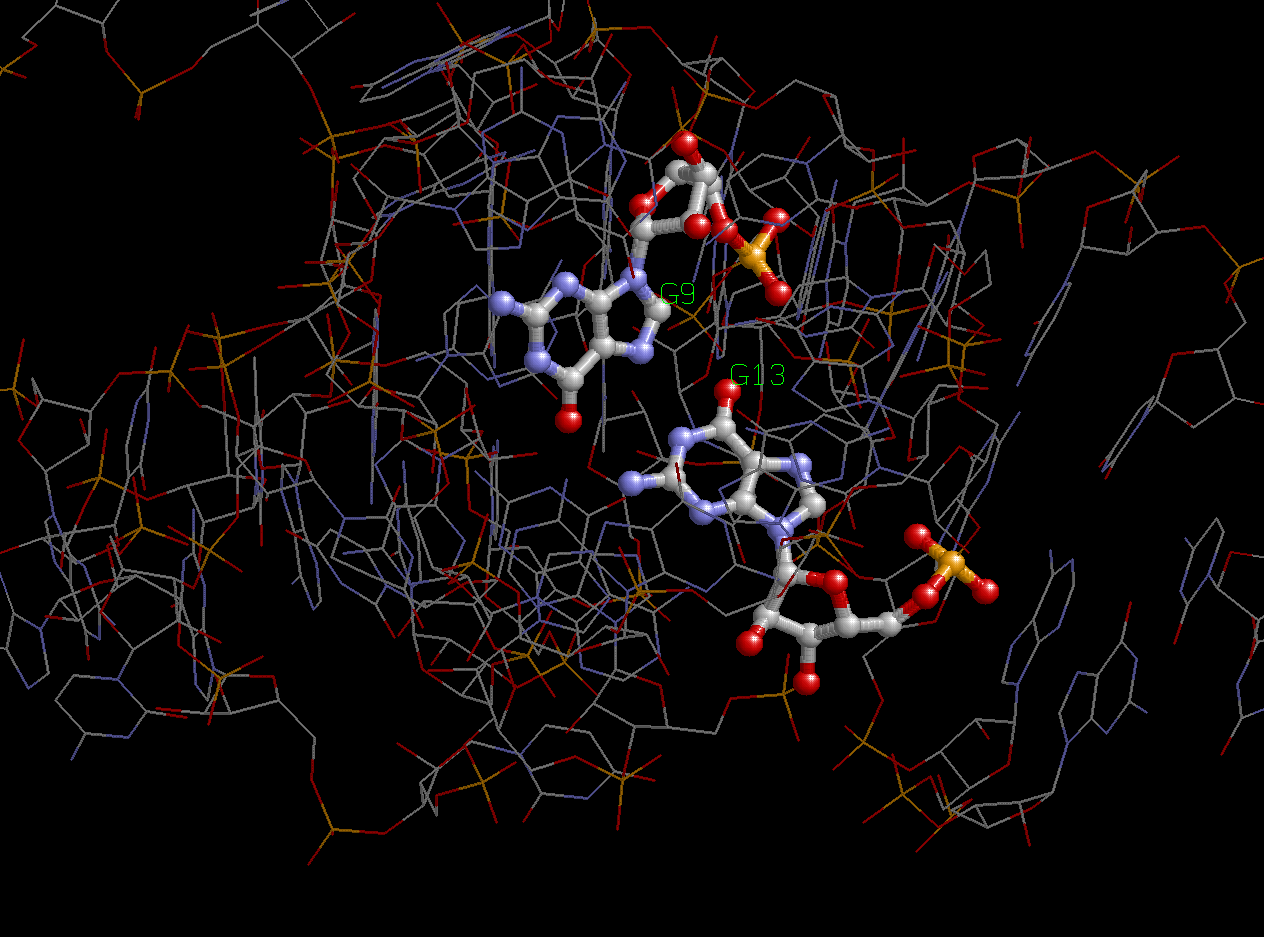
Рис.2. Неканоническое взаимодействие между нуклеотидами G9 и G13
- Стекинг-взаимодействие возможно между основаниями нуклеотидных пар G7-C66 из конца акцепторного стебля и G49-С65 из начала Т-стебля. Это подтверждает структура под номером 4 в таблице из файла 1ser_r_old.out, представленной ниже:
� step i1-i2 i1-j2 j1-i2 j1-j2 sum 1 GA/UC 2.73( 1.22) 0.00( 0.00) 0.00( 0.00) 0.16( 0.00) 2.90( 1.22) 2 AG/CU 3.12( 2.81) 0.00( 0.00) 0.30( 0.00) 0.00( 0.00) 3.42( 2.81) 3 GG/CC 3.38( 2.01) 0.00( 0.00) 0.03( 0.00) 1.28( 0.07) 4.69( 2.09) 4 GG/CC 2.69( 1.19) 0.00( 0.00) 0.74( 0.00) 0.20( 0.00) 3.63( 1.19) 5 GC/GC 4.57( 1.67) 0.00( 0.00) 0.00( 0.00) 7.18( 4.42) 11.76( 6.09) 6 CG/CG 0.27( 0.00) 0.00( 0.00) 5.00( 2.02) 0.00( 0.00) 5.27( 2.02) 7 GG/CC 4.52( 3.03) 0.00( 0.00) 0.49( 0.00) 0.00( 0.00) 5.01( 3.03) 8 GG/CC 3.64( 1.76) 0.00( 0.00) 1.54( 0.03) 0.00( 0.00) 5.18( 1.79) 9 Gu/AC 8.20( 2.78) 0.00( 0.00) 0.00( 0.00) 4.97( 2.05) 13.16( 4.83) 10 uP/GA 7.08( 2.21) 0.00( 0.00) 0.00( 0.00) 5.60( 3.14) 12.67( 5.35) 11 PC/GG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 12 CC/GG 3.19( 0.87) 0.00( 0.00) 0.00( 0.00) 3.63( 1.70) 6.82( 2.57) 13 CC/GG 0.46( 0.00) 0.00( 0.00) 0.19( 0.00) 1.32( 0.21) 1.97( 0.21) 14 CG/GG 4.81( 2.73) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 4.81( 2.73) 15 GA/UG 0.00( 0.00) 4.88( 1.59) 0.00( 0.00) 0.00( 0.00) 4.88( 1.59) 16 AG/CU 3.50( 1.24) 0.00( 0.00) 0.05( 0.00) 0.00( 0.00) 3.55( 1.24) 17 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 18 GA/UC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 19 AG/CU 1.77( 1.57) 0.00( 0.00) 0.01( 0.00) 1.37( 0.43) 3.15( 2.00) 20 GG/CC 4.52( 3.29) 0.00( 0.00) 0.14( 0.00) 0.00( 0.00) 4.66( 3.29) 21 GG/CC 3.20( 1.70) 0.00( 0.00) 0.52( 0.00) 0.00( 0.00) 3.73( 1.70) 22 GG/UC 1.99( 0.57) 0.00( 0.00) 0.58( 0.00) 0.00( 0.00) 2.57( 0.57) 23 GG/CU 3.75( 2.35) 0.00( 0.00) 0.00( 0.00) 2.97( 1.15) 6.72( 3.50) 24 GG/CC 4.05( 2.44) 0.00( 0.00) 0.55( 0.00) 0.00( 0.00) 4.60( 2.44)
Площадь перекрывания этих пар очень незначительно (всего 3.63 ангстрема в квадрате). Поэтому стекинг-взаимодействие не очень сильное, хотя оно, безусловно, присутствует. Это показано на следующем рисунке (полученном с помощью программы stack2img):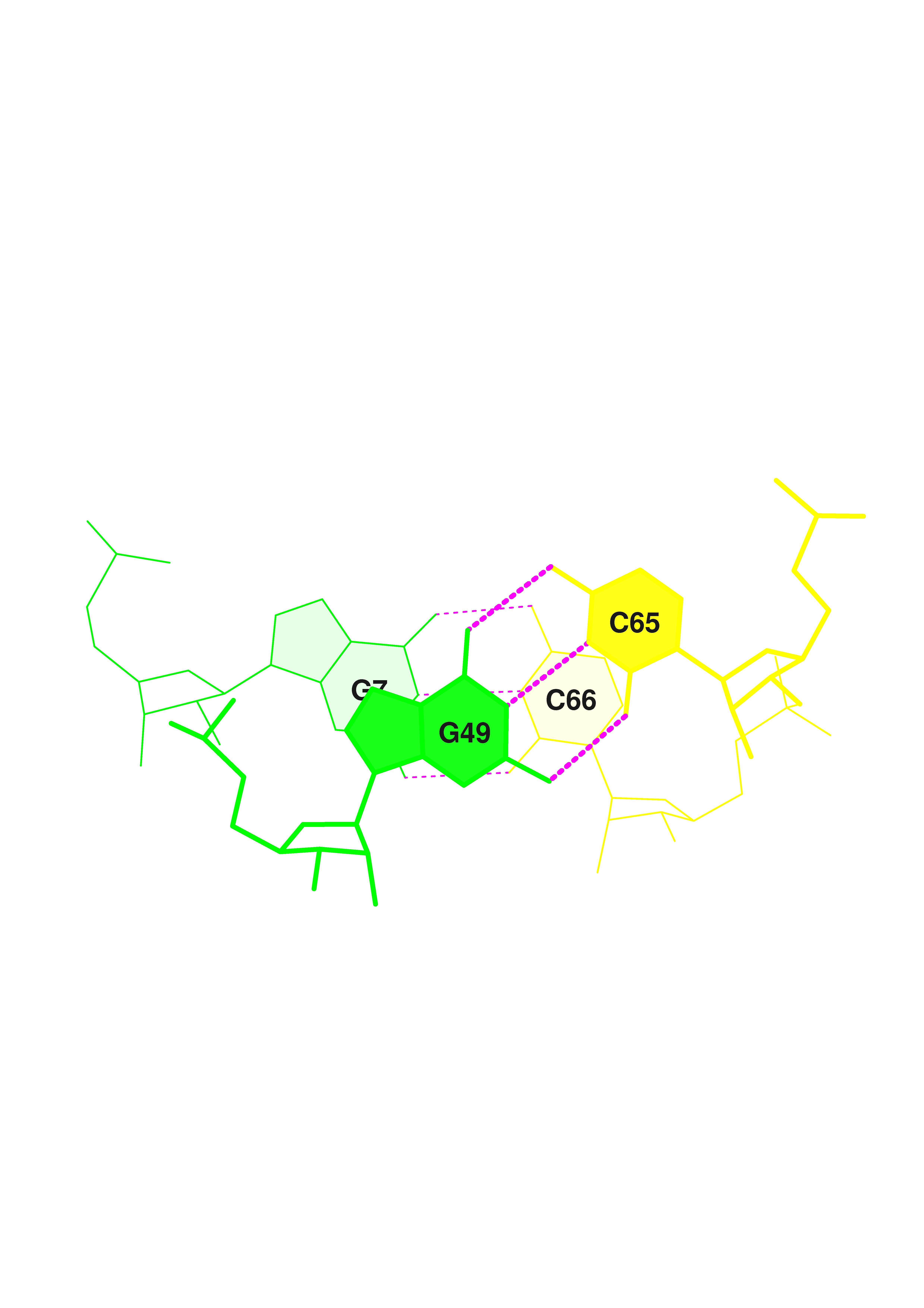 �
�
Рис.3. Стекинг-взаимодействие между G7-C66/G49-C65
Ниже изображено то же самое стекинг-взаимодействие, полученное с помощью программы RasMol. Как можно с легкостью заметить, перекрывание действительно очень незначительно, такое взаимодействие не является очень уж крепким:
Рис.5. Стекинг-взаимодействие между G7-C66/G49-C65 (RasMol)
- Между основаниями Т- и D-петель есть две дополнительные водородные связи. Это связи G19-C56 и PSU55-G18 (где PSU - псевдоурацил). Первая из них, таким образом, является канонической, а вторая - неканонической. Кроме того, имеется одна водородная связь между двумя основаниями Т-петли. Это связь 5MU54-A58 (где 5MU - 5-метилурацил - по сути, взаимодействие это почти не отличается от канонического взаимодействия U-A). Ниже изображено самое интересное взаимодействие из этих трех - неканоническое взаимодействие псевдоурацила с гуанином:
Рис.4. Неканоническое взаимодействие PSU55-G18
Реальная и предсказанная вторичная структура тРНК из файла 1ser_r.pdb
| Участок структуры | Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' 4-7 3' 5' 66-69 3' Всего 4 пары |
Предсказано 7 пар (из которых 4 содержались в файле 1ser_r.pdb) | Предсказано 7 пар (из которых 4 содержались в файле 1ser_r.pdb) |
| D-стебель | 5' 10-12 3' 5' 23-25 3' Всего 3 пары |
Предсказано 0 пар из 3 реальных | Предсказано 3 пары из 3 реальных |
| T-стебель | 5' 49-53 3' 5' 61-65 3' Всего 5 пар |
Предсказано 0 пар из 5 реальных | Предсказано 5 пар из 5 реальных |
| Антикодоновый стебель | 5' 42-43 3' Комплементарных пар в файле нет |
Предсказано 0 пар | Предсказано 6 пар (в файле 1ser_r.pdb не содержащихся) |
| Общее число канонических пар нуклеотидов | 21 | 7 | 21 |
Для подготовки к предсказанию вторичной структуры тРНК я создал файла с последовательностью в fasta-формате: 1ser_r.fasta.
Что касается программы einverted, то она меня совсем не поразила. Пока "Minimum score threshold" не было снижено до 20, а "gap penalty" до 8, никаих резульатов программа вообще не выдавала. После установки таких значений она выдала единственный вариант - 7 комплементарных пар в акцепторном стебле. Это, конечно, был правильный результат (получилось 7 пар, а не 4, как у меня, потому что последовательность в einverted была загружена полностью, а в pdb-файле не содержалось координат этих остатков), но, во-первых, его было очень тяжело достигнуть, а во-вторых, никаких других канонических пар программа не выдала. Изменения параметров "Match score" и "Mismatch score" ничего разумного не приносили, как и дальнейшее снижение штрафа за гэпы и минимального порогового значения. Да и в этом случае, почти единственное, что сделала программа - это продлила число пар в начале акцепторного стебля с 7 до 10. Результат, вообще говоря, не ахти.
Чего не скажешь про алгоритм Зукера. Вначале он не хотел выдавать ожидаемого результата, почему-то обрывая последовательность РНК до 80-го нуклеотида (в моей последовательности их 94). Поэтому мне пришлось удалить из fasta-файла с последовательностью нуклеотиды, образующие вариабельную петлю. После этого число оснований стало равно 76 (1ser.fasta), и программа mfold смогла обработать всю последовательность. Признаться, я был приятно удивлен. При стандартном значении параметра P алгоритм Зукера выдал две возможные вторичные структуры сериновой тРНК. Первая из них была очень похожа на правду, но не все ее канонические пары совпадали с аналогичными в файле 1ser_r_old.out. Но вторая структура предсказала все канониеские взаимодействия, отмеченные в файле 1ser_r_old.out, добавив к этому взаимодействия между нуклеотидами, координаты которых в pdb-файле не содержались. Антикодон GGA занял почетное место в антикодоновой петле, где ему и следует быть. В общем, структура была предсказана идеально. Увеличения параметра P не давали лучших результатов (а давали скорее смешные структуры типа одной длинной цепочки). На рисунке, представленном снизу, отображен лучший (второй) результат, полученный с помощью алгоритма Зукера. На нем можно увидеть и акцепторный стебель (снизу), и антикодоновую петлю (наверху), и Т-петлю (справа), и D-петлю (слева) с 5,6-дигидроуридином-5'-монофосфатом под 20 номером.
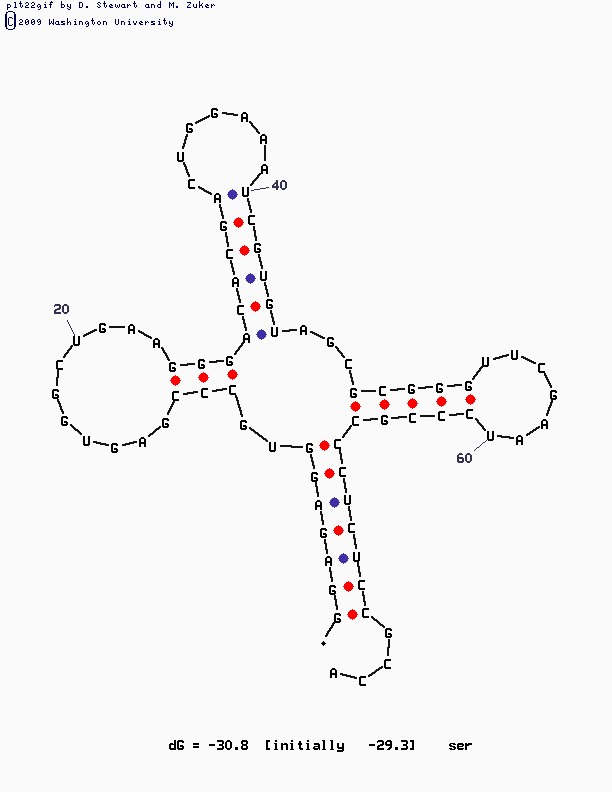
Рис. 5. Лучшее предсказание вторичной структуры сериновой тРНК по алгоритму Зукера
Таким образом, в то время как программа einverted дает очень маленькое представление о вторичной структуре тРНК, алгоритм Зукера создает верное и надежное предсказание. Единственным минусом является ограниченность длины последовательности, подвергающейся обработке в алгоритме Зукера (не более 80 нуклеотидов).