Определение вторичной структуры белка с помощью программы DSSP
Определим вторичную структуру белка 1F30 программой DSSP. Для этого выполним на kodomo команду:dsspcmbi 1f30.pdb 1f30.dsspВыходной файл 1f30.dssp импортируем в Excel. Для каждой цепи посчитаем число элементов вторичной структуры (то есть число блоков идущих подряд букв H (альфа-спираль) или Е (бета-тяж)).
Таблица с результатом была сохранена в файле 1f30.xls (лист vtor). Для каждой цепи программа DSSP нашла 5 элементов альфа-спирали, но ни одного элемента бета-тяжа. Координаты альфа-спиралей для каждой цепи выглядят так:
Координаты альфа-спиралей по данным программы DSSP
| Номер цепи | 1-я альфа-спираль | 2-я альфа-спираль | 3-я альфа-спираль | 4-я альфа-спираль | 5-я альфа-спираль |
| A | 23-53 | 59-86 | 95-101 | 114-138 | 142-164 |
| B | 23-53 | 59-86 | 95-101 | 114-137 | 142-164 |
| C | 23-53 | 59-86 | 95-101 | 114-138 | 142-164 |
| D | 23-53 | 59-86 | 95-101 | 114-138 | 142-164 |
| E | 23-53 | 59-86 | 95-101 | 114-138 | 142-165 |
| F | 23-53 | 59-86 | 95-101 | 114-137 | 142-164 |
| G | 23-53 | 59-86 | 95-98 | 114-137 | 142-164 |
| H | 23-53 | 59-86 | 95-101 | 114-137 | 142-164 |
| I | 23-53 | 59-86 | 95-101 | 114-137 | 142-164 |
| J | 23-53 | 59-86 | 95-99 | 114-137 | 142-164 |
| K | 23-53 | 59-86 | 95-99 | 114-137 | 142-164 |
| L | 23-53 | 59-86 | 95-101 | 114-138 | 142-164 |
Теперь обратимся к полям HELIX и SHEET файла 1f30.pdb. По данным файла PDB, белок не содержит бета-тяжей (не имеется раздела SHEET), что совпадает с данными программы DSSP. Сравним данные файла PDB о координатах альфа-спиралей с данными программы DSSP. Координаты альфа-спиралей по данным файла PDB выглядят так:
Координаты альфа-спиралей по данным файла 1f30.pdb
| Номер цепи | 1-я альфа-спираль | 2-я альфа-спираль | 3-я альфа-спираль | 4-я альфа-спираль | 5-я альфа-спираль |
| A | 22-54 | 58-87 | 94-102 | 113-139 | 141-165 |
| B | 22-54 | 58-87 | 94-102 | 113-136 | 141-165 |
| C | 22-54 | 58-87 | 94-102 | 113-139 | 141-165 |
| D | 22-54 | 58-87 | 94-102 | 113-139 | 141-165 |
| E | 22-54 | 58-87 | 94-102 | 113-139 | 141-166 |
| F | 22-54 | 58-87 | 94-102 | 113-138 | 141-165 |
| G | 22-54 | 58-87 | 94-99 | 113-136 | 141-165 |
| H | 22-54 | 58-87 | 94-102 | 113-136 | 141-165 |
| I | 22-54 | 58-87 | 94-102 | 113-136 | 141-165 |
| J | 22-54 | 58-87 | 94-100 | 113-136 | 141-165 |
| K | 22-54 | 58-87 | 94-100 | 113-136 | 141-165 |
| L | 22-54 | 58-87 | 94-102 | 113-139 | 141-165 |
Анализ значений торсионного угла φ в выдаче DSSP
Проанализируем теперь колонку PHI выдачи DSSP. Определим остатки, имеющие положительные значения угла φ. Для этого вновь воспользуемся программой Excel. Список остатков можно увидеть на листе phi файла 1F30.xls. Всего нашлось 52 таких остатка:Треонин 12 цепи А; глицин 56 цепи А; аспарагин 58 цепи А; глицин 88 цепи А; треонин 12 цепи B; глицин 56 цепи B; аспарагин 58 цепи B; глицин 88 цепи B; треонин 12 цепи C; аспарагин 13 цепи С; глицин 56 цепи C; аспарагин 58 цепи C; глицин 88 цепи C; треонин 12 цепи D; глицин 56 цепи D; аспарагин 58 цепи D; глицин 88 цепи D; треонин 12 цепи E; глицин 56 цепи E; аспарагин 58 цепи E; глицин 88 цепи E; треонин 12 цепи F; глицин 56 цепи F; аспарагин 58 цепи F; глицин 88 цепи F; треонин 12 цепи G; глицин 56 цепи G; аспарагин 58 цепи G; глицин 88 цепи G; глицин 100 цепи G; треонин 12 цепи H; глицин 56 цепи H; аспарагин 58 цепи H; глицин 88 цепи H; треонин 12 цепи I; аспарагин 13 цепи I; глицин 56 цепи I; аспарагин 58 цепи I; глицин 88 цепи I; треонин 12 цепи J; глицин 56 цепи J; аспарагин 58 цепи J; глицин 88 цепи J; треонин 12 цепи K; глицин 56 цепи K; аспарагин 58 цепи K; глицин 88 цепи K; треонин 12 цепи L; лейцин 14 цепи L; глицин 56 цепи L; аспарагин 58 цепи L; глицин 88 цепи L.Полученный список остатков вызвал у меня некоторое удивление. Во-первых, странным показалось, что хотя цепи идентичны по последовательности, некоторые остатки в некоторых цепях имеют положительное значение угла φ, в то время как эти же остатки в других цепях положительного значения не имеют. Скорее всего, это ошибка программы, вписывавшей атомы согласно электронной плотности молекулы. Более того, в файле PDB в поле REMARK описаны 3 из 4 таких остатка (все кроме глицина 88 цепи K, для которого положительное значение угла φ возможно вследствие маленького размера радикала):
REMARK 500 TORSION ANGLES OUTSIDE THE EXPECTED RAMACHANDRAN REGIONS: REMARK 500 (M=MODEL NUMBER; RES=RESIDUE NAME; C=CHAIN IDENTIFIER; REMARK 500 SSEQ=SEQUENCE NUMBER; I=INSERTION CODE). REMARK 500 REMARK 500 STANDARD TABLE: REMARK 500 FORMAT:(10X,I3,1X,A3,1X,A1,I4,A1,4X,F7.2,3X,F7.2) REMARK 500 REMARK 500 M RES CSSEQI PSI PHI REMARK 500 ASN C 13 124.64 83.45 REMARK 500 ASN I 13 -30.23 79.70 REMARK 500 LEU L 14 77.60 120.31Кроме того, удивило то, что среди остатков, имеющих положительное значение угла φ, оказались не только глицины, но и аспарагин и лейцин, имеющие достаточно большие по размеру радикалы, мешающие вращению вокруг N-Cα связи (что и определяет угол φ). Треонины в списке, также имеющие положительное значение угла φ, не вызывают удивления, потому что они являются первыми остатками каждой цепи, для которых известны координаты, а значит угол φ для них определить невозможно. Поэтому он принимается равным 360.
Создадим в PyMOL изображение, представляющее остовную модель (ribbon) цепи А белка, на котором различными цветами выделены альфа-спирали, остатки с положительным значением угла φ и все прочие:
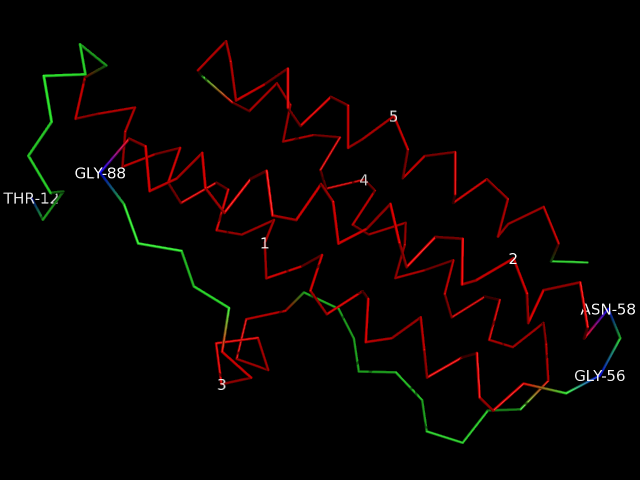
На этом рисунке красным изображены альфа-спирали, синим - остатки с положительным значением угла φ, зеленым - все остальные остатки. Остатки с положительным значение угла φ, кроме того, подписаны трехбуквенным названием и номером остатка в цепи. Альфа-спирали подписаны порядковым номером их расположения в цепи.
Из рисунка видно, что цепь действительно состоит из 5 альфа-спиралей, из которых 3-я короткая, а остальные - длинные. Остатки с положительными значениями угла φ расположены в местах поворота цепи перед или после альфа-спирали (кроме треонина 12, который располагается в самом начале цепи).
Определение водордных связей с помощью программы HBPlus
Определим водородные связи в записи 1f30.pdb с помощью программы HBPlus. Для этого оставим в PDB-файле белка лишь координаты атомов полипептидной цепи А и лигандов, после чего сохраним их в файле 1f30b.pdb и выполним на kodomo следующую команду:hbplus 1f30b.pdbНа выходе получаем файл 1f30b.hb2.
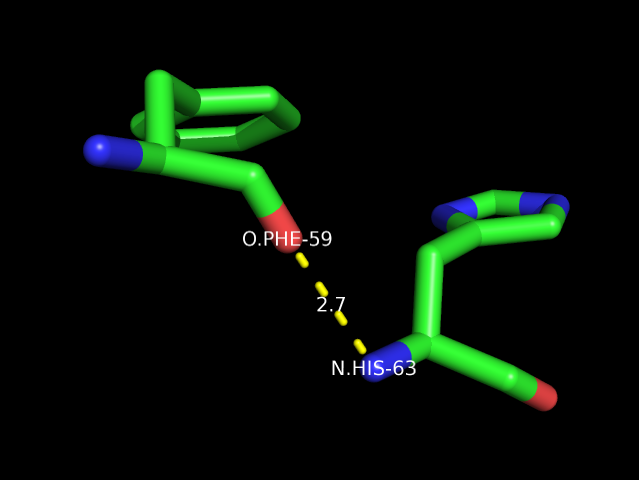
- Примером водородных связей, участвующих в стабилизации вторичной структуры, является водородная связь между атомом кислорода остатка Phe-59 и атомом азота остатка His-63 (оба эти остатка входят в альфа-спираль и иллюстрируют правило, согласно которому остовный атом кислорода n-го остатка образует водородную связь с остовным азотом (n+4)-го остатка):
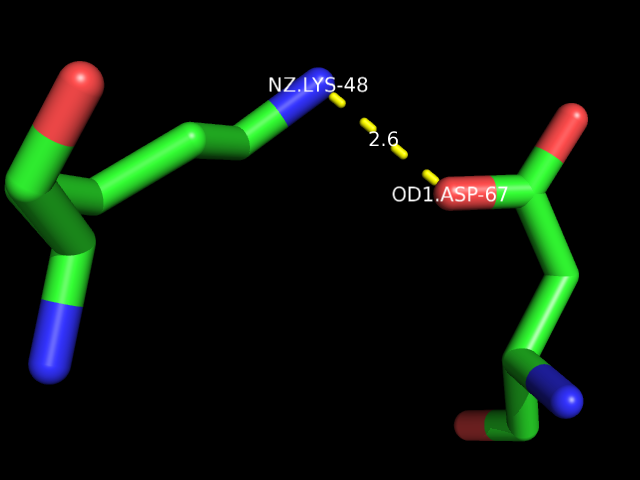
- Примером водородных связей между боковыми цепями аминокислотных остатков является водородная связь между атомом азота аминогруппы боковой цепи остатка Lys-48 и атомом кислорода карбоксильной группы боковой цепи остатка Asp-67:
- Примером водородных связей между боковой цепь одного остатка и остовным атомом другого является водородная связь между атомом кислорода гидрокисльной группы боковой цепи остатка Thr-79 и остовным атомом кислорода остатка Asp-75:
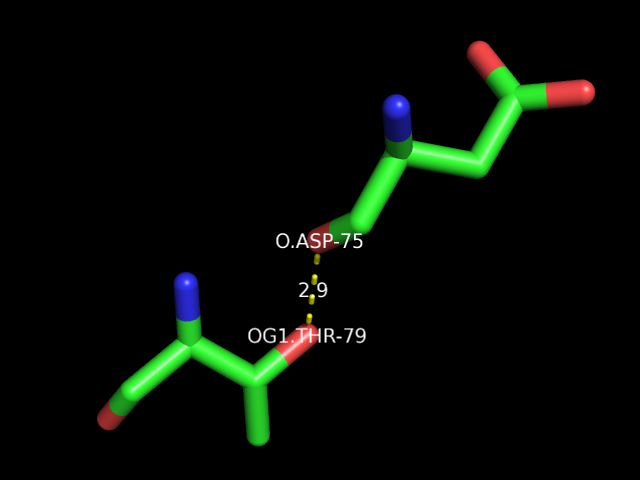
- Водородных связей между белком и лигандом (TRS - 2-амино-2-гидроксиметил-пропан-1,3-диолом) программа HBplus, к сожалению, не обнаружила.
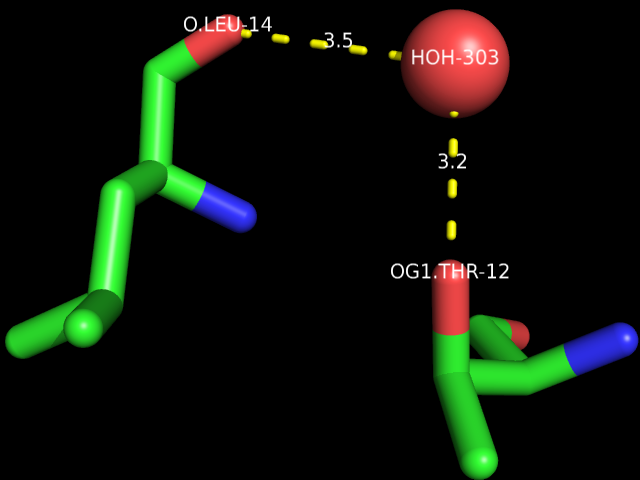
- Что касается водяных мостиков, то примером таких структур может служить мостик между атомом кислорода гидроксильной группы боковой цепи остатка Thr-12 и остовным атомом кислорода остатка Leu-14, образованный водородными связями с молекулой воды HOH-303:
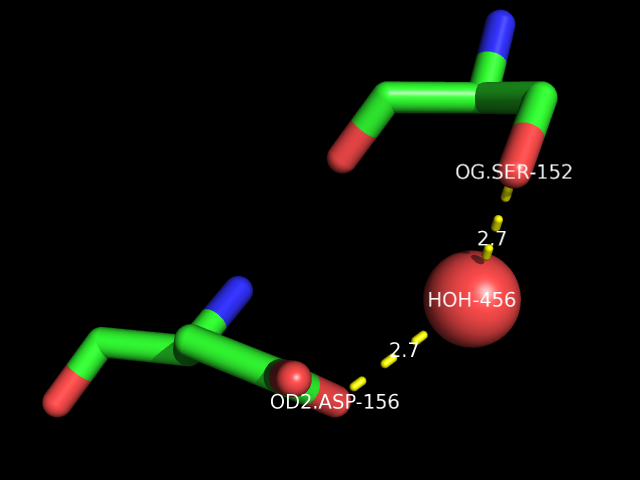
Другой пример - связь атома кислорода гидроксильной группы боковой цепи остатка Ser-152 с атомом кислорода карбоксильной группы боковой цепи остатка Asp-156, осуществляемая через водородные связи с молекулой воды HOH-456: