Классификация доменов записей PDB 1F30 и 1U94 согласно SCOP
Определим классификацию доменов записи 1F30 согласно SCOP. К сожалению для этой записи нашелся всего один домен - додекамерный гомолог ферритина из E. coli (Dodecameric ferritin homolog from Escherichia coli):- расположен во всех двенадцати цепях белка 1F30, занимает всю цепь целиком;
- относится к семейству ферритинов (Ferritin); суперсемейству ферритин-подобных (Ferritin-like); укладке ферритин-подобных (Ferritin-like); классу альфа-спиральных белков (All alpha proteins);
- суперсемейство ферритин-подобных (Ferritin-like) содержит 9 семейств;
- укладку ферритин-подобных (Ferritin-like) содержат 6 суперсемейств.
Возьмем для дальнейшего исследования запись 1U94 (белок RecA из E. coli). Согласно SCOP белок состоит из 2 доменов.
Первый из них - домен АТФазы белка RecA из E. coli (RecA protein, ATPase-domain from Escherichia coli):
- расположен в цепи А белка 1U94, занимает в цепи позиции 6-268;
- относится к семейству RecA-подобных (домен АТФазы) (RecA protein-like (ATPase-domain)); суперсемейству нуклеозидтрифосфат-гидролаз, содержащих P-петлю (P-loop containing nucleoside triphosphate hydrolases); укладке нуклеозидтрифосфат-гидролаз, содержащих P-петлю (P-loop containing nucleoside triphosphate hydrolases); классу альфа/бета белков (Alpha and beta proteins (a/b));
- суперсемейство нуклеозидтрифосфат-гидролаз, содержащих P-петлю (P-loop containing nucleoside triphosphate hydrolases), содержит 24 семейства;
- укладку нуклеозидтрифосфат-гидролаз, содержащих P-петлю, (P-loop containing nucleoside triphosphate hydrolases) содержит 1 суперсемейство.
Второй из них - С-концевой домен белка RecA из E. coli (RecA protein, C-terminal domain from Escherichia coli):
- расположен в цепи А белка 1U94, занимает в цепи позиции 269-328;
- относится к семейству C-концевых доменов белка RecA (RecA protein, C-terminal domain); суперсемейству C-концевых доменов белка RecA (RecA protein, C-terminal domain); укладке доменов анти-липополисахаридного фактора или recA (Anti-LPS factor/recA domain); классу альфа+бета белков (Alpha and beta proteins (a+b));
- суперсемейство C-концевых доменов белка RecA (RecA protein, C-terminal domain) содержит 1 семейство;
- укладку доменов анти-липополисахаридного фактора или recA (Anti-LPS factor/recA domain) содержат 2 суперсемейства.
Классификация доменов записей PDB 1F30 и 1U94 согласно CATH
Определим для тех же записей классификацию доменов согласно CATH.Для записи 1F30 CATH определил 12 доменов (расположенных, соответственно, в 12 цепях). Впрочем, они ничем друг от друга не отличаются (12 цепей в белке 1F30 идентичны), поэтому рассмотрими один из них - домен 1f30A00:
- расположен в цепи А белка 1F30, занимает в цепи позиции 12-167;
- классификация по CATH: 1.20.1260.10.3.2.1.2.13;
- домен относится к топологии ферритинов (Ferritin) (1.20.1260); архитектуре Up-down bundle (1.20); классу доменов, состоящих, в основном, из альфа-спиралей (Mainly alpha) (1);
- в суперсемействе 1.20.1260.10 содержится 26 семейств;
- топологию ферритинов (Ferritin) (1.20.1260) имеет 1 суперсемейство.
Для записи 1U94 CATH определил 2 домена.
Первый из них - домен 1u94A01:
- расположен в цепи А белка 1U94, занимает в цепи позиции 27-269;
- классификация по CATH: 3.40.50.300.33.1.1.1.2;
- домен относится к суперсемейству нуклеотидтрифосфат-гидролаз, содержащих P-петлю (P-loop containing nucleotide triphosphate hydrolases) (3.40.50.300); топологии укладки Россманна (Rossmann fold) (3.40.50); архитектуре 3-слойного сэндвича (3-Layer(aba) Sandwich) (3.40); классу альфа и бэта (Alpha Beta) (3).
- в суперсемействе нуклеотидтрифосфат-гидролаз, содержащих P-петлю (P-loop containing nucleotide triphosphate hydrolases) (3.40.50.300), содержится 208 семейств;
- топологию укладки Россманна (Rossmann fold) (3.40.50) имеет 120 суперсемейств.
Второй из них - домен 1u94A02:
- расположен в цепи А белка 1U94, занимает в цепи позиции 270-328;
- классификация по CATH: 3.30.250.10.1.1.1.1.1;
- домен относится к суперсемейству домена 2 белка RecA (Rec A Protein, domain 2) (3.30.250.10); топологии домена 2 белка RecA (Rec A Protein, domain 2) (3.30.250); архитектуре 2-слойного сэндвича (2-Layer Sandwich) (3.30); классу альфа и бэта (Alpha Beta) (3);
- в суперсемействе домена 2 белка RecA (Rec A Protein, domain 2) (3.30.250.10) содержатся 2 семейства;
- топологию домена 2 белка RecA (Rec A Protein, domain 2) (3.30.250) имеет 1 суперсемейство.
Различия между CATH и SCOP в описании доменов записей 1F30 и 1U94
Доменная организация белка записи 1F30 определена в CATH и SCOP одинаково.Небольшое отличие в определении доменной организации белка записи 1U94 заключается в том, что SCOP выделил координаты первого домена записи как 6-268, а CATH - 27-269; соответственно в CATH начальная координата второго домена этой записи сдвинулась на один остаток (270-328).
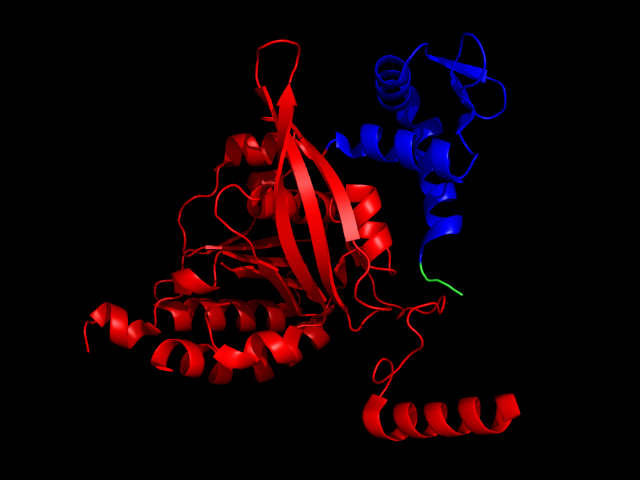
Доменная организация белка записи 1U94 согласно SCOP представлена ниже:
Доменная организация этого же белка согласно CATH:
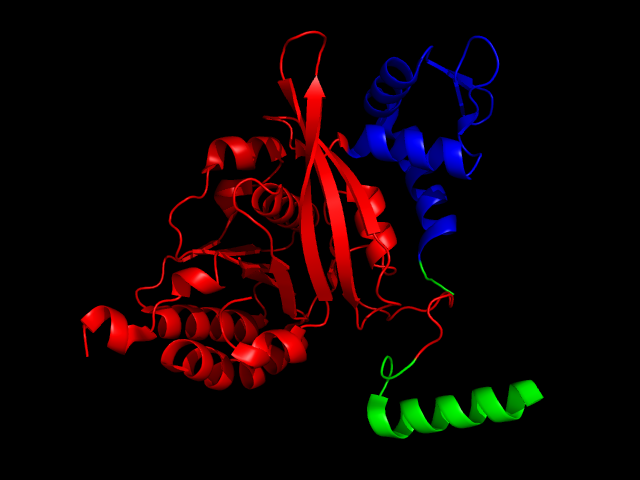
Красным окрашен первый домен, синим - второй домен.
Домен записи 1F30 описан в CATH и SCOP одинаково (принадлежит классу альфа-спиральных белков, укладке/топологии ферритинов). Единственная разница в том, что в CATH классификация более подробная (больше уровней). В частности, согласно CATH 1F30 принадлежит архитектуре Up-down Bundle (такого аналога в SCOP нет).
Домен АТФазы белка 1U94 определен в CATH и SCOP практически одинаково (за исключением того, что в SCOP его укладка называется "нуклеозидтрифосфат-гидролазы, содержащие P-петлю", а в CATH топология называется "укладка Россманна"; впрочем, суперсемейства, следующие далее по классификации, для этого домена по CATH и SCOP вновь совпадают).
Наконец, уровни C-концевого домена белка 1U94 в CATH и SCOP называются немножко по-разному. Укладка этого домена по SCOP - "домен анти-LPS фактора или белка RecA"; топология по CATH - "домен 2 белка RecA". Суперсемейство по SCOP - "C-концевой домен белка RecA"; суперсемейство по CATH - "домен 2 белка RecA".
Некоторые домены имеют разную классификацию в CATH и SCOP.
Например, белок S046, имеющий согласно SCOP ту же укладу, что и C-концевой домен белка 1U94, в CATH просто не представлен.
Другой пример - домен 1v96A00 из записи 1V96, имеющий согласно CATH ту же топологию, что и домен АТФазы белка 1U94 (укладка Россманна), но имеющий другую укладку согласно SCOP (не нуклеозидтрифосфат-гидролазы, содержащие P-петлю, а PIN-подобные домены (PIN domain-like)).
Наконец, последний пример - домен 2qipA00 из записи 2QIP, имеющий ту же топологию, что и 1u94A01 (укладка Россманна), представлен в CATH, но отсутствует в SCOP.
Причинами таких различий в доменной классификации могут быть: разное количество уровней (в СATH уровней 9 в то время, как в SCOP их всего 6), а значит и различная степень подробности; дата последнего релиза (CATH обновился позже - 7 июля 2009 года, в то время как SCOP обновился в июне 2009); разное суммарное число доменов (в CATH их 128688, в то время как в SCOP - 110800). Даже по изучению одних и тех же доменов в SCOP и CATH в заданиях 1 и 2, видно, что одна и та же топология в CATH и укладка в SCOP могут содержать разное количество суперсемейств (например, топология ферритинов в CATH содержит всего 1 суперсемейство, в то время как укладка ферритин-подобных содержит 6 суперсемейств). Такие же различия можно увидеть на всех уровнях классификации доменов в CATH и SCOP.
Выравнивание доменов, имеющих одну укладку, но состоящих в разных суперсемействах
Построим выравнивание двух доменов с одной укладкой по SCOP, но из разных суперсемейств. Для задания были выбраны цепь А записи 1so0 (мутаротаза галактозы из человека (Galactose mutarotase from Human (Homo sapiens))) и цепь А записи 1jmm (V-участок поверхности атигена I/II (SA I/II, PAC) из Streptococcus mutans). Оба домена имеют укладку суперсэндвич (Supersandwich), но первый из них принадлежит суперсемейству галактозы мутаротаз-подобных доменов (Galactose mutarotase-like), а второй - суперсемейству V-участка поверхности антигена I/II (V-region of surface antigen I/II (SA I/II, PAC)). Воспользуемся программой PDBeFOLD (SSM). На выходе получаем файл с жестким выравниванием последовательностей в формате fasta 1jmm1so0.fasta. Подадим его на вход программе Geometrical core для нахождения геометрического ядра с порогом 2Å. На выходе получаем таблицу с остатками из структур, образующими геометрическое ядро:| Pos. | 1JMM_A | 1SO0_A |
| 142 | SER605 | THR39 |
| 144 | THR607 | THR41 |
| 145 | ALA608 | ALA42 |
| 147 | TYR610 | GLU44 |
| 166 | TYR628 | VAL56 |
| 183 | TYR630 | GLY73 |
| 184 | THR631 | ALA74 |
| 185 | VAL632 | VAL75 |
| 258 | VAL643 | VAL148 |
| 275 | TYR660 | ARG162 |
| 282 | LYS666 | THR169 |
| 283 | ASN667 | PRO170 |
| 284 | THR668 | VAL171 |
| 285 | SER669 | ASN172 |
| 286 | ILE670 | LEU173 |
| 287 | PHE671 | THR174 |
| 288 | ILE672 | ASN175 |
| 289 | LYS673 | HIS176 |
| 290 | ASN674 | SER177 |
| 291 | GLU675 | TYR178 |
| 343 | LYS726 | LEU202 |
| 475 | ASN781 | GLY303 |
| 477 | VAL783 | CYS305 |
| 479 | VAL785 | GLU307 |
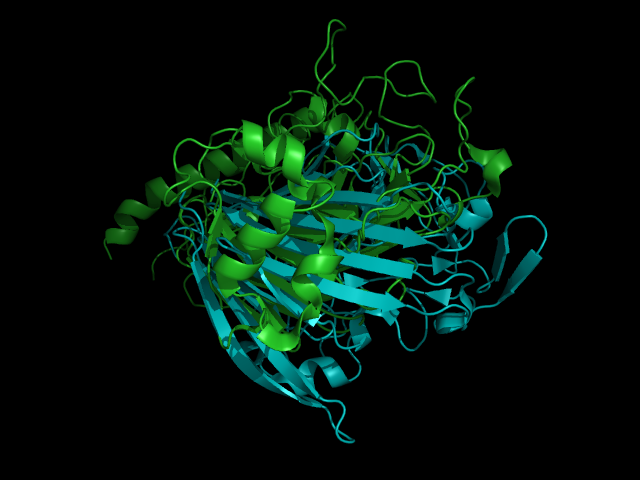
Синим на рисунке окрашена цепь А записи 1so0, зеленым - цепь А записи 1jmm. RMSD совмещения по геометрическому ядру составляет 1.711 (24 to 24 atoms). Это не очень хороший результат (учитывая, что совмещались геометрические ядра).
Из рисунка видно, что структуры выравнились плохо, хотя в них легко угадывается один и тот же тип супервторичной структуры. Гибкое выравнивание на сервисе FATCAT не сильно улучшает картину:
FATCAT нашел 5 кластеров плюс-блоков, причем лишь два из них имеют rmsd меньше 3, что говорит о плохом выравнивании структур. Гибкое выравнивание было сохранено в файле 1so01jmmflex.txt. Впрочем, в целом фрагменты супервторичной структуры выравнялись лучше, чем в результате жесткого выравнивания.
Теперь построим выравнивание двух доменов с одной топологией по CATH, но из разных суперсемейств. Выберем для изучения домен, занимающий позиции 326-649 в цепи A записи 1gxm, и домен, занимающий позиции 1-404 в цепи А записи 1h12. Оба домена имеют топологию гликозилтрансфераз (Glycosyltransferase - 1.50.10). Но домен цепи А записи 1h12 относится к суперсемейству 1.50.10.10 и выделен из Pseudoalteromonas haloplanktis, а домен цепи А записи 1gxm относится к суперсемейству 1.50.10.20 и выделен из Cellvibrio japonicus.
Для этого вновь воспользуемся программой PDBeFOLD (SSM). На выходе получаем файл с жестким выравниванием последовательностей в формате fasta 1h121gxm.fasta. Подадим его на вход программе Geometrical core для нахождения геометрического ядра с порогом 2Å. На выходе получаем таблицу с остатками из структур, образующими геометрическое ядро:
| Pos. | 1GXM_A | 1H12_A |
| 95 | SER352 | GLU94 |
| 150 | THR393 | GLU146 |
| 158 | ALA401 | LEU154 |
| 161 | TYR404 | SER157 |
| 165 | GLY408 | GLY161 |
| 166 | ASN409 | ASN162 |
| 171 | LYS411 | ASN167 |
| 172 | TYR412 | TYR168 |
| 173 | ARG413 | TYR169 |
| 174 | ASP414 | ASN170 |
| 176 | VAL416 | ALA172 |
| 177 | ARG417 | ILE173 |
| 195 | PRO434 | ARG190 |
| 209 | HIS446 | LEU198 |
| 210 | ALA447 | THR199 |
| 211 | THR448 | ASP200 |
| 223 | VAL460 | PHE208 |
| 224 | LEU461 | TYR209 |
| 245 | THR482 | ASN223 |
| 247 | PHE484 | TRP225 |
| 248 | LYS485 | ARG226 |
| 249 | THR486 | GLN227 |
| 250 | ALA487 | VAL228 |
| 251 | VAL488 | ALA229 |
| 252 | THR489 | THR230 |
| 253 | LYS490 | LYS231 |
| 254 | GLY491 | SER232 |
| 255 | THR492 | ARG233 |
| 256 | ASP493 | THR234 |
| 257 | TYR494 | LEU235 |
| 258 | ILE495 | LEU236 |
| 259 | LEU496 | LYS237 |
| 260 | LYS497 | ASN238 |
| 261 | ALA498 | HIS239 |
| 262 | GLN499 | PHE240 |
| 274 | TRP509 | PRO252 |
| 275 | CYS510 | THR253 |
| 276 | ALA511 | PHE254 |
| 277 | GLN512 | LEU255 |
| 278 | HIS513 | SER256 |
| 286 | PRO520 | PRO262 |
| 287 | LYS521 | VAL263 |
| 298 | LEU528 | PRO274 |
| 302 | SER532 | TYR278 |
| 374 | TYR598 | SER330 |
| 381 | GLY605 | GLY333 |
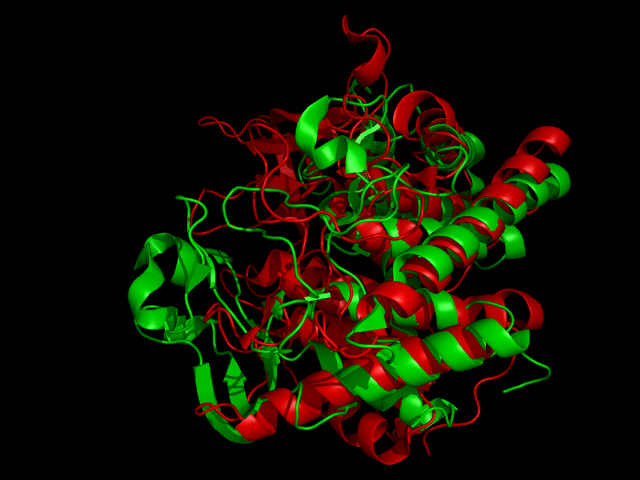
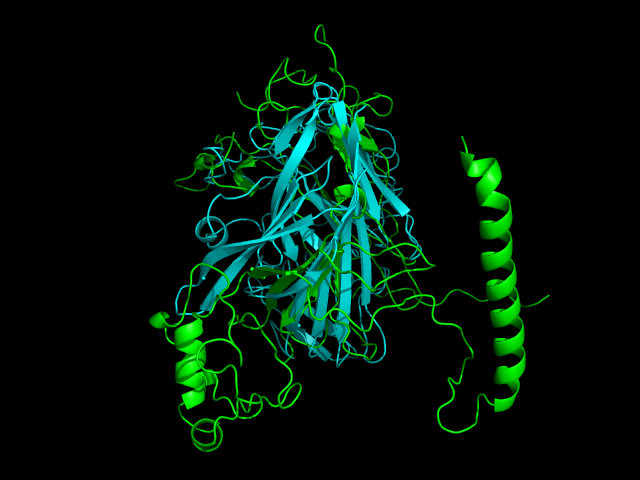
Красным на рисунке окрашен домен цепи А записи 1h12, зеленым - домен цепи А записи 1gxm. RMSD выравнивания Сα-атомов, входящих в геометрическое ядро, равно 1.590 (46 to 46 atoms).
Из рисунка видно, что домены совместились гораздо лучше, чем в предыдущем случае. Однако, опять же, структуры плохо накладываются друг на друга, хотя сходство их супервторичной структуры не вызывает никакого сомнения.
Воспользуемся сервисом FATCAT для улучшения выравнивания. Полученное гибкое выравнивание было сохранено в файле 1h121gxmflex.txt. FATCAT нашел 4 кластера плюс-блоков. Изображение гибкого выравнивания представлено ниже:
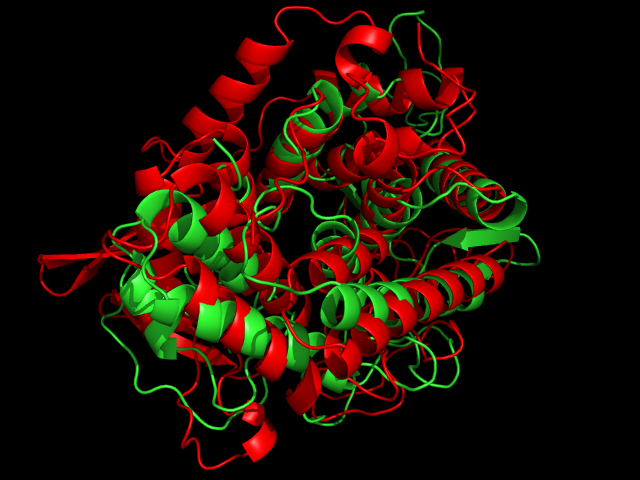
Выравнивание улучшилось незначительно. Структуры плохо выравниваются целиком, но элементы их супервторичной структуры устроены очень похоже.
Таким образом, можно сделать вывод, что домены, имеющие одинаковую укладку (топологию), но входящие в разные суперсемейства, плохо выравниваются друг с другом, хотя элементы их супервторичной структуры (укладка) устроены одинаково. В некоторых случаях укладки похожи в общих чертах, но все же не очень хорошо накладываются друг на друга (как, например, в доменах записей 1so0 и 1jmm), в других же укладки прекрасно совмещаются друг с другом (как, например, в доменах записей 1h12 и 1gxm).
Можно объяснить полученный результат тем, что разделение доменов на классы, архитектуры и топологии (укладки) основано исключительно на их пространственной структуре (что уже гарантирует сходство типов строения), а разделение на суперсемейства и семейства подразумевает очень высокий процент сходства в струткурах (выходящий за уровень сходства лишь элементов вторичной структуры), а также сходство в функциях и последовательностях доменов. Таким образом, одна и та же топология (укладка) двух разных доменов подразумевает неплохое соответствие элементов супервторичной структуры, но слабое соответствие структур в целом.