 |
 |
Учебный сайт Морозова Александра |
<< Назад к странице 3 семестра
Практикум 14I. Подготовка чтений1) Удаление адаптеров. Все адаптеры были собраны в один файл командой: cat *.fa >> adapters.fasta. Команда, использованная для удаления адаптеров: java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240361.fastq noadapter.fastq ILLUMINACLIP:adapters.fasta:2:7:7 Выдача: Input Reads: 7272621 Surviving: 7238064 (99,52%) Dropped: 34557 (0,48%) Это значит, что 34557 ридов состояли только из адаптеров и были удалены. 2) Удаление плохих нуклеотидов Плохие - нуклеотиды с качеством ниже 20. Использованная команда: java -jar /usr/share/java/trimmomatic.jar SE -phred33 noadapter.fastq cleared.fastq TRAILING:20 Выдача: Input Reads: 7238064 Surviving: 7153776 (98,84%) Dropped: 84288 (1,16%) 3) Удаление ридов размера меньше 30 Использованная команда: java -jar /usr/share/java/trimmomatic.jar SE -phred33 cleared.fastq trimmed.fastq MINLEN:30 Выдача: Input Reads: 7153776 Surviving: 6881690 (96,20%) Dropped: 272086 (3,80%) Таким образом, из 7272621 ридов осталось только 6881690. II. Velveth1) Получение к-меров Был запущен velveth с командой: velveth kmers 29 -short -fastq trimmed.fastq где kmers - название директории, куда был помещен файл с полученными к-мерами; -short - параметр, означающий, что чтения (риды) короткие и непарные; -fastq - формат входного файла, параметр, который, хотя и является опциональным, должен быть введен обязательно, иначе velveth выдает ошибку вида: velveth: trimmed.fastq does not seem to be in FastA format, т.к. по умолчанию использует формат fasta; trimmed.fastq - файл с ридами, поданный на вход программе. В созданной папке kmers содержатся 3 файла: Log, содержащий некоторую информацию о параметрах выполнения команды, Roadmaps и Sequences, содержащий полученные к-меры. III. Velvetg1) Сборка генома ВАЖНО: velvetg не имеет опции -help. Чтобы понять, как он работает, нужно запустить его без параметров. Программа velvetg была запущена c командой: velvetg assembly/, где assembly - название рабочей директории. На момент запуска в ней находились файлы: trimmed.fastq, содержащий риды, а также файлы Log Roadmaps Sequences, полученные в результате работы программы velveth. По завершении работы программа выдала сообщение: Final graph has 1209 nodes and n50 of 49972, max 155850, total 690701, using 0/6881690 reads Было получено 3 файла: contigs.fa, содержащий контиги в fasta-формате, stats.txt, содержащий описание всех полученных контигов, и LastGraph, содержащий описание графа, созданного velvet. Из сообщения видно, что было получено 1209 контигов. По умолчанию программа не записывает в файл contigs.fa контиги с длиной меньше, чем 2k, где k - длина слова, использованная в работе прогаммы velveth. В нашем случае были удалены контиги длины меньше, чем 58, хотя их описание можо найти в файле stats.txt (так, просмотрев этот файл, обнаружил, что, начиная примерно с 30-ого номера, встречается очень много контигов двузначной длины, а ближе к концу файла - и вовсе однозначной). N50 полученной сборки - 49972. Самые длинные контиги: 1. NODE_3 с длиной 155850 и средним покрытием 33.079514;
2. NODE_11 с длиной 85024 и средним покрытием 34.670528;
3. NODE_1 с длиной 72780 и средним покрытием 35.516788
IV. Megablast1) NODE_3 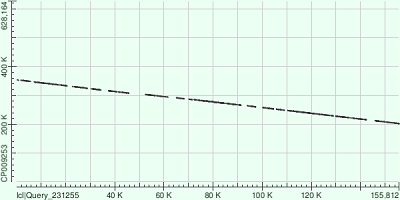 Рис. 1. Изображение карты локального сходства хромосомы бактерии Buchnera aphidicola и контига node_3.
Как можно видеть из карты локального сходства, в целом, данный контиг ложится на хромосому хорошо, за исключением некоторых участков, которые будут рассмотрены подробнее ниже. Координаты участков хромосомы, соответствующих контигу:
№ Коорд. контига Коорд. хромосомы № Коорд. контига Коорд. хромосомы
1. 37-843 353822-353014 17. 72300-73409 285070-283963
2. 1672-4246 352456-349918 18. 73657-81878 283706-275566
3. 4495-5937 349674-348233 19. 81937-91416 275551-266073
4. 7060-10399 346547-343228 20. 93966-97537 263784-260224
5. 10545-11817 343052-341781 21. 100315-104637 257546-253223
6. 12176-20526 341508-333222 22. 105728-108930 252161-248967
7. 22517-23190 331006-330333 23. 110407-121104 247596-236918
8. 23571-26340 330003-327227 24. 121202-125731 236859-232358
9. 26632-28838 326950-324747 25. 125880-129000 232057-228944
10. 30158-34371 323043-318826 26. 129900-134011 228137-224057
11. 37405-40713 315982-312679 27. 134400-138502 223720-219625
12. 41358-45669 312179-307878 28. 138550-139210 219491-218821
13. 52503-59856 303252-295935 29. 139649-142334 218384-215717
14. 60165-61691 295755-294227 30. 145910-148869 212243-209294
15. 65113-68479 291560-288181 31. 149315-150205 208904-208017
16. 70269-71603 286535-285200 32. 150564-155812 207661-202390
Как можно видеть из представленных координат, несовпадение некоторых участков, видимое на карте локального сходства, можно объяснить крупными геномными перестройками. Так, например, разность между концом участка №12 и началом участка №13 контига составляет 6834 нуклеотида, в то время как для соответствующих участков хромосомы бактерии она составляет всего лишь 4626 нуклеотидов, что может свидетельствовать о вставке 2208 нуклеотидов в контиге или, наоборот, делеции в хромосоме бактерии. 2) NODE_11 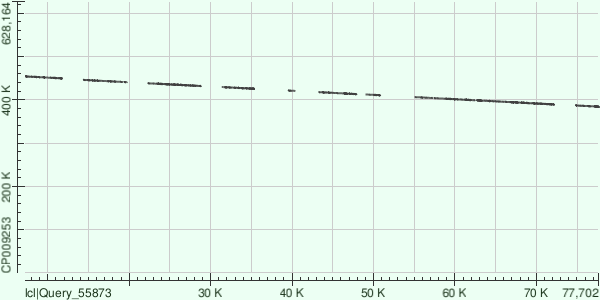 Рис. 2. Изображение карты локального сходства хромосомы бактерии Buchnera aphidicola и контига node_11.
Как можно видеть из карты локального сходства, данный контиг в сравнении с предыдущим также ложится на хромосому хорошо, однако заметны большие участки несоответствия, которые могут быть интерпретированы как геномные перестройки. Также при сравнении двух карт хорошо видно, что данный контиг является продолжением предыдущего при наложении на хромосому. Координаты участков хромосомы, соответствующих контигу:
№ Коорд. контига Коорд. хромосомы № Коорд. контига Коорд. хромосомы
1. 7198- 11850 454069-449411 9. 39557-40410 421327-420477
2. 14422-17449 445895-442877 10. 43342-47936 417677-413081
3. 17579-19254 442817-441135 11. 49023-50837 412321-410512
4. 19376-19557 440944-440755 12. 55085-57253 406218-404050
5. 19647-19728 440732-440652 13. 57496-62479 403823-398904
6. 22353-25276 438139-435267 14. 62724-72165 398726-389348
7. 25338-28767 435241-431839 15. 74888-76355 386887-385425
8. 31369-35447 429483-425412 16. 76419-77702 385420-384182
3) NODE_1 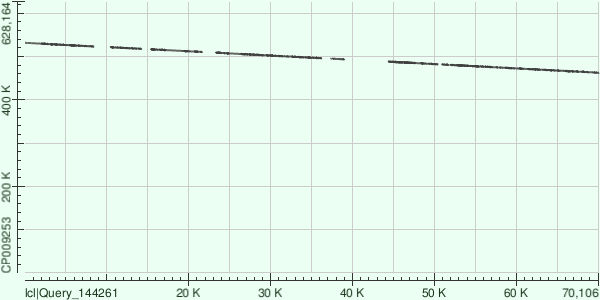 Рис. 3. Изображение карты локального сходства хромосомы бактерии Buchnera aphidicola и контига node_1.
При сравнении данной карты локального сходства с предыдущими видно, что данный контиг является продолжением контига node_11 при наложении на хромосому. При сравнении с хромосомой также видны 5 крупных перестроек. Координаты участков хромосомы, соответствующих контигу:
№ Коорд. контига Коорд. хромосомы № Коорд. контига Коорд. хромосомы
1. 5-2796 531590-528794 9. 44428-50485 488106-481997
2. 2866-8454 528679-523105 10. 50960-51639 481545-480874
3. 10457-14186 521500-517766 11. 51845-57719 480660-474844
4. 15452-21631 516539-510438 12. 57822-65135 474667-467412
5. 23323-31768 508806-500370 13. 65165-70106 467421-462496
6. 31884-36161 500325-36161
7. 37327-37445 495148-495033
8. 37579-38955 494864-493487
|
|||||||||||||||||||||||||||